Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista Peruana de Biología
versión On-line ISSN 1727-9933
Rev. peru biol. vol.26 no.4 Lima oct./dic 2019
http://dx.doi.org/10.15381/rpb.v26i4.17220
NOTA CIENTÍFICA
Caracterización molecular de los microorganismos presentes durante el proceso fermentativo de los granos de cacao (Theobroma cacao)
Molecular characterization of microorganisms during cocoa-bean fermentation process (Theobroma cacao)
Juana Inés Machuca-Guevara* 1, 3, Erick Antonio Suárez-Peña 2 ORCID iD: 0000-0003-0137-8251, Emmerik Motte Darricau 3, 4 ORCID iD: 0000-0001-9577-2887, Eric Louis Mialhe-Matonnier 3, 4
1 Universidad Nacional de Tumbes, Perú.
2 Cooperativa de Trabajadores BIOTECOOP, Dpto de Biotecnología Agrícola, Perú.
3 Empresa de investigación y servicio Inca Biotec SAC, Tumbes, Perú
4 Concepto Azul, Guayaquil, Ecuador.
Resumen
La fermentación de granos de cacao es un proceso espontáneo de post cosecha muy importante para el desarrollo de aroma y sabor a chocolate el cual involucra un sin número de actividades microbianas complejas. En el presente estudio se identifican los microorganismos presentes en granos de cacao antes, durante y después del proceso de fermentación aplicando dos métodos: el análisis de secuenciamiento de ADN y la espectrometría de masas MALDI TOF/TOF. Dentro del grupo de bacterias y levaduras predominantes identificadas por el primer método se encontro a Lactobacillus plantarum (29%), L. brevis (18%), Bacillus cereus (15%), Pediococcus acidilactici (12%), y Pichia kudriavzevii (100%). Asimismo se caracterizó por huella de masas las secuencias peptídicas más importantes de cada cepa identificada. Por otro lado, aplicando el segundo método, se identificaron 57 especies de microorganismos, siendo el 73.7% especies bacterianas y el 26.3% especies de levaduras. Adicionalmente se detectaron secuencias peptídicas de la proteína vicilina responsable del aroma característico de los granos de cacao fermentados y a la proteína albumina de 21KDa.
Palabras clave: Granos de cacao; fermentación; microorganismos; secuenciamiento de ADN; MALDI-TOF/TOF MS.
Abstract
Cocoa beans fermentation is a spontaneous process of post-harvest very important for the development of chocolate aroma and flavor, which involves a number of complex microbial activities. In this work, we identify the microorganisms present in cocoa beans before, during and after the fermentation process, applying DNA sequencing analysis and MALDI TOF / TOF mass spectrometry. With the first method, the predominant bacteria and yeast identified were Lactobacillus plantarum (29%), L. brevis (18%), Bacillus cereus (15%), Pediococcus acidilactici (12%), and Pichia kudriavzevii (100%). The most important peptide sequences of each identified strain by mass fingerprint were characterized too. By the second method, 51 species of microorganisms being 73.7% bacterial species and 26.3% yeast species were identified. Additionally peptide sequences responsible Vicilin protein characteristic aroma of the fermented cocoa beans and the albumin protein of 21KDa were detected.
Keywords: Cocoa beans; fermentation; microorganisms; DNA sequencing; MALDI-TOF/TOF MS.
Introducción
El cacao (Theobroma cacao L.) es una especie económicamente importante, cultivada por más de 6 millones de agricultores a nivel global, además es el medio de subsistencia para más de 40 millones de personas, y aproximadamente entre el 80 – 90% de la producción mundial de cacao proviene de pequeños productores (Wickramasuriya & Dunwell 2018). Aunque, los granos de cacao son la fuentes principal para la producción de chocolate y otros derivados, ellos no contienen los determinantes del aroma y sabor del chocolate, sino que es necesario un procesamiento post-cosecha (fermentación, secado y tostado) para la obtención del sabor (Wickramasuriya & Dunwell 2018, Lima et al. 2011, Camu et al. 2007 & Camu et al. 2008).
El proceso de fermentación es llevado a cabo por una sucesión de varias especies de levaduras, bacterias acido lácticas (BAL), bacterias acido acéticas (BAA) (Arana et al. 2015, Bortolini et al. 2016) y, posiblemente, especies de Bacillus, otras bacterias y hongos filamentosos (Ho et al. 2014, 2015) que influenciarían en la calidad del producto (Moreira et al. 2013 & Visintin et al. 2015). Al final de la fermentación se producen los precursores del sabor como son los ácidos orgánicos, azúcares reducidos y aminoácidos libres, asimismo la disminución de polifenoles y alcaloides presentes en el grano de cacao fresco que dan lugar al amargor y astringencia desagradable (Wickramasuriya & Dunwell 2018).
En el proceso fermentativo del cacao han sido reportadas las levaduras Saccharomyces cereviciae (Ardhana & Fleet et al. 2003, Lima et al. 2011, Moreira et al. 2013 & Visintin et al. 2015), Hanseniaspora guilliermondii, Pichia kudriavzevii, Kluyveromyces marxianus (Ho et al. 2014, 2015); las bacterias ácido lácticas Lactobacillus plantarum y L. fermentum (Camu et al. 2007, 2008; Lefeber et al. 2011, Lima et al. 2011, Moreira et al. 2013, Ho et al. 2014, 2015 & Visintin et al. 2015), las bacterias ácido acéticas Acetobacter pasteurianus (Camu et al. 2008; Lefeber et al. 2011), A. aceti (Lima et al. 2011), y otras como Bacillus cereus, B. subtilis, B. pumilis (Ardhana & Fleet et al. 2003; Lima et al. 2011).
La espectrometría de masas del tipo ionización/desorción de laser asistida por matriz con tiempo de vuelo (MALDI-TOF MS) es una técnica alternativa para la identificación de microorganismos (Sun et al. 2006, Miescher et al. 2016, Pedrozo et al. 2017) que tiene un número de ventajas en rapidez, costo y eficiencia, cuando se le compara con los métodos basados en el secuenciamiento de ADN y cultivo tradicional (Böhme et al. 2013). Schmidt et al. (2009) mencionan que existen dos métodos principales para el análisis de proteínas de bacterias por MS. El método de perfil de proteínas intactas (IPP), que determina las masas moleculares de proteínas intactas, mientras que la combinación de péptidos resultantes a partir de proteínas digeridas por enzimas son analizados mediante el mapeo de masas shotgun (SMM).
En este trabajo, estudiamos la diversidad microbiana presente durante el proceso fermentativo de los granos de cacao empleando el secuenciamiento del gen ADNr 16S y la región ITS para la identificación de bacterias y levaduras respectivamente, asimismo se caracterizan estos microorganismos a través de sus secuencias peptídicas mediante la espectrometría de masas (MALDI-TOF MS). Adicional se realiza un análisis metaproteómico, identificar otros microorganismos que puedan presentarse en este proceso e identificar secuencias peptídicas de precursores de aroma en el grano de cacao.
Material y métodos
Descripción de la zona de muestreo y toma de muestra.- La toma de muestras se realizó en octubre 2016 y diciembre 2017, en el centro de acopio de la Asociación Regional de Productores de Cacao de Tumbes (ARPROCAT), ubicada en el departamento de Tumbes, provincia de Zarumilla en la Villa Uña de Gato (3°32'18"S, 80°13'50"W).
Se evaluaron dos cajones fermentadores (var. Criollo y Forastero), de los cuales se colectaban de 3 – 4 granos de cacao cada día durante los 8 días que duró el proceso de fermentación y se depositaron en tubos Falcon® que contenían 30 mL de solución Ringer (Visintin et al. 2015) y medio MRS (Man, Rogosa y Sharpe). Además, al final del proceso, se colectaron y almacenaron en bolsas herméticas granos fermentados y secos. Las muestras fueron procesadas en las instalaciones del laboratorio IncaBiotec SAC.
Aislamiento y purificación de microorganismos presentes en el proceso fermentativo de los granos de cacao.- Aplicando técnicas microbiológicas convencionales se procedió a realizar los aislamientos y purificaciones respectivas realizando diluciones seriadas, siembra en placa por esparcimiento y siembra por estriado en superficie en medios de cultivo YPD (extracto de levadura, peptona y dextrosa), MRS (Man, Rogosa y Sharpe) y YGC (extracto de levadura, glucosa y carbonato de calcio), sembrándose en cada medio 25 µL de las diluciones 10-4 y 10-5 (Melo et al, 2012). Las cepas puras fueron duplicadas y una réplica se conservó a -20 °C con 15% de glicerol y la otra replica fue utilizada para su extracción de ADN genómico.
Extracción de ADN genómico bacteriano.- Se aplicó solución salina tamponada con fosfato (PBS 1X) para la extracción de ADN genómico, se centrifugó 1 mL de cultivo bacteriano de 24 h de crecimiento a 10000 rpm por 2 min, luego se eliminó el sobrenadante y se resuspendió el sedimento con 500 μL de PBS 1X. Posteriormente se centrifugó a 10000 rpm por 2 min y se descartó el sobrenadante. El pellet resuspendido con 200 μL de TE 1X (Tris-EDTA) se llevó a ebullición durante 10 min e inmediatamente se colocó sobre hielo por 5min. Se centrifugó a 10000 rpm por 2min. Se transfirió 100 μL del sobrenadante a un nuevo microtubo y se le añadió 1μL de ARNasa (0.1 mg/mL). Se incubó a 65 °C por 15 min y se conservó a -20 °C.
Amplificación del Gen 16S ADNr mediante la PCR (reacción en cadena de la polimerasa).- El ADN extraído se utilizó para realizar la PCR y amplificar el gen ADNr 16S. El mix de reacción 25 μM contiene Buffer Taq 1X, MgCl2 a 1.5 mM, dNTPs a 0.2 mM, primer 27F (AGAGTTTGATCMTGGCTCAG) a 0.36 μM y primer 1492R (GGTTACCTTGTTACGACTT) a 0.36 μM (Galkiewicz, 2008), 0.5 U de Taq ADN polimerasa recombinante, 2 µL de ADN y agua libre de nucleasas. La programación en el termociclador (BIOMETRA UNO-Thermoblock) fue: temperatura de pre-desnaturalización de 95 °C por 5 min; 35 ciclos de desnaturalización a 95 °C por 30 seg, hibridación a 58 °C por 45 seg y polimerización de 72 °C por 1 min 30 seg, además un paso final de Extensión de 72 °C por 4 min y conservación a 4 °C.
Extracción de ADN genómico de levaduras.- Se extrajo ADN de levaduras a partir de 1 mL de cultivo puro de 24 h de crecimiento y se empleó el siguiente protocolo: se macera el cultivo celular y se le adiciona 100 μL de buffer de lisis (200 mM de acetato de litio y 1% de dodecil sulfato de sodio (SDS)), se mezcló suavemente e incubó a 70 °C por 15 min en baño maría.
Se centrifugó a 13000 rpm por 3 min. Se transfirió 90 μL del sobrenadante a un nuevo microtubo y se le adicionó 300 μL de etanol al 95%, se mezcló y se centrifugó a 13000 rpm por 1 min. Se eliminó el sobrenadante y el pellet se resuspendió en 100 μL de solución TE 1X. Luego se centrifugó a 13000 rpm por 1 min. El sobrenadante se transfirió a un nuevo microtubo, se agregó 1 μL de ARNasa e incubó por 30 min a 37 °C. El ADN se conservó a -20 °C.
Amplificación de la región ITS (espaciador transcrito interno) mediante la PCR (Reacción en cadena de la polimerasa).- El ADN extraído se utilizó para realizar la PCR y amplificar la región ITS. El mix de reacción de 25 μM contiene: Buffer Taq 1X, MgCl2 a 1.5mM; dNTP's a 0.2 mM; primer ITS1 a 0.36 µM (TCCGTAGGTGAACCTGCGG) y ITS4 a 0.36 µM (TCCTCCGCTTATTGATATGC), 0.5 U de Taq DNA polimerasa, 2 μL de ADN y completado con agua ultra pura. La programación del termociclador (BIOMETRA UNO-Thermoblock) fue: temperatura de pre-desnaturalización a 94 °C por 5 min, seguido de 35 ciclos de desnaturalización de 94 °C por 45 seg, hibridación de 54 °C por 35 seg y polimerización 72 °C por 45 seg, además extensión final de 72 °C por 5 min y conservación a 4 °C.
Secuenciamiento y análisis de resultados.- Los productos de PCR fueron confirmados en gel de agarosa al 1.5%, luego fueron enviados a Macrogen Inc. para su secuenciación. Finalmente las secuencias fueron alineadas con el banco de datos en línea Nucleotide BLAST (Basic Local Alignment Search Tool) del NCBI (National Center for Biotechnology Information; https://blast.ncbi.nlm.nih.gov/Blast.cgi).
Análisis proteómico mediante Espectrometría de Masas del tipo Ionización/Desorción de Laser Asistida por Matriz con Tiempo de Vuelo (MALDI-TOF MS
Huella de masas peptídicas de cepas aisladas por Maldi MS.- Se empleó el protocolo de lavado celular descrito por Schmidt et al. (2009) con previas modificaciones. Luego, 1 μL de células son depositadas en la placa MALDI OptiTof™ por triplicado, y se dejó secar. Se agregó 1 μL de solución matriz de ácido sinapínico (AS) y se dejó secar. Para el caso de las levaduras, se aplicó un paso adicional de lisis antes de ser depositado en la placa MALDI OptiTof™. Los espectros de masa fueron obtenidos utilizando el espectrómetro de masas MALDI-TOF (5800, AB SCIEX System), los espectros fueron captados modo MS, lineal positivo con una intensidad de 4500 dentro de un rango de masas entre 2000 a 18000 m/z (Böhme et al. 2013, Quintela-Baluja et al. 2013).
Las masas en común fueron analizadas en el programa en línea Speclust, (http://co.bmc.lu.se/speclust/ 189 cluster.pl) (Santos et al. 2015). Se examinaron cuatro espectros por muestra con un error de medición de ±3Da (Böhme et al. 2013).
Análisis de mapeo de masas Shotgun por MALDI TOF/TOF.- Para el siguiente análisis se empleó el protocolo desarrollado por Schmidt et al. (2009) con previas modificaciones (el ácido alfa-ciano-4-hidroxi cinámico (CHCA) se usó como matriz).
Los péptidos tripsinados se obtuvieron a partir de un sistema analizador proteómico MALDI-TOF/TOF (5800, AB SCIEX System), los espectros fueron captados modo MS, reflector positivo con un láser Nd: YAG de 349 nm con una intensidad de 2800 y una velocidad de 600 μM/seg, 750 disparos dentro de un rango de masas entre 800 a 4000 m/z, y procesados en el software ProteinPilotTM v 4.5. Para la calibración del equipo se utilizó un kit Mass Standards AB SCIEX TOF/TOFTM Instruments.
Extracción de proteínas totales (metaproteómica) de los microorganismos presentes en los co-cultivos de granos de cacao en fermentación.- Las muestras contenidas en tubos falcon con medio MRS se incubaron a 37 °C durante 24 h a 48 h. Luego se extrajo 1 mL del co-cultivo, se depositó en un microtubo y se centrifugo para obtener el pellet microbiano (se repitió lo mismo 3 veces más hasta ser concentrado). Luego se aplicó el protocolo de extracción de proteínas con Qproteome Bacterial Protein Prep Kit de QIAGEN (www.qiagen.com) y se cargaron en gel de electroforesis de poliacrilamida dodecil sulfato de sodio (SDS-PAGE) al 12% a 90V por 2h.
Tinción del gel.- Se usó azul de Coomasie para el revelado del gel. Luego fue sumergido en agua destilada por 20 min. Se cubrió el gel con solución de fijación e incubó por 30 minutos. Se lavó, se agregó la solución colorante y se incubó por 12 h agitándose ocasionalmente. Al gel lavado se le agregó la solución de decoloración y se incubó por 15min (agitándose constantemente). Nuevamente el gel fue lavado y se dejó en agua destilada. Finalmente, se observaron las bandas y se cortaron con bisturí según sus pesos moleculares colocándose cada una de las bandas en un microtubo de 0.2 mL (Lomonte 2007).
Decoloración de bandas, saturación de la pieza del gel con buffer tripsina, digestión y extracción de péptidos.- Se siguió el protocolo empleado por Shevchenko et al. (2006).
Extracción metaproteómica de granos de cacao fermentados y secados.- Mediante espectrometría de masas MALDI-TOF/TOF, se hizo un análisis metaproteómico de los granos de cacao fermentados y secos; asimismo, se analizaron los péptidos precursores de aroma específicos del cacao del endospermo, producidos durante el proceso de fermentación, empleado el protocolo de extracción de péptidos descrito por Pirovani et al. (2008) con previas modificaciones.
Los péptidos extraídos se migran en un gel de poliacrilamida al 12% a 90 voltios por 2 horas. Pasado ese tiempo se siguió el mismo procedimiento para la tinción del gel y corte de bandas descrito anteriormente. Seguido, se aplicó el protocolo de decoloración de bandas, saturación de la pieza del gel con buffer tripsina, digestión y extracción de péptidos descrito por Shevchenko et al. (2006).
Análisis de secuencias peptídicas obtenidas por espectrometría de masas MALDI TOF/TOF.- Las secuencias peptídicas fueron alineadas con el banco de datos del Protein BLAST (Basic Local Alignment Search Tool) del NCBI (National Center for Biotechnology Information) (https://blast.ncbi.nlm.nih.gov/Blast.cgi).
Resultados
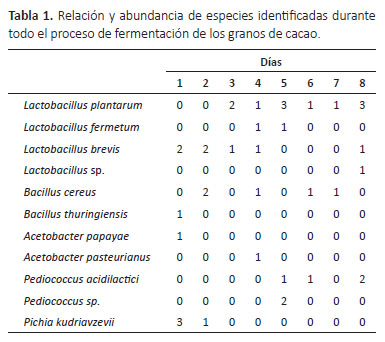
Identificación de microorganismos presentes durante el proceso fermentativo de los granos de cacao.- Se identificaron 34 cepas bacterianas y 4 cepas de levaduras durante el proceso de fermentación. Las cepas más predominantes son Lactobacillus plantarum (29%), L. brevis (18%), Bacillus cereus (15%) y Pediococcus acidilactici (12%), y dentro de las levaduras está Pichia kudriavzevii (100%). (Tabla 1).
Análisis proteómico mediante Espectrometría de Masas de Ionización/ Desorción de Láser Asistida por Matriz con Tiempo de Vuelo (MALDI-TOF MS).
Huella de masa de cepas aisladas por MALDI TOF MS.- Mediante espectrometría de masas se obtuvieron los espectros de masas de péptidos representativos de Lactobacillus plantarum, L. brevis, L. fermentum, B. cereus, B. thuringiensis, Acetobacter pasteurianus subsp. pasteurianus, P. acidilactici y P. kudriavzevii (Apéndice 1).
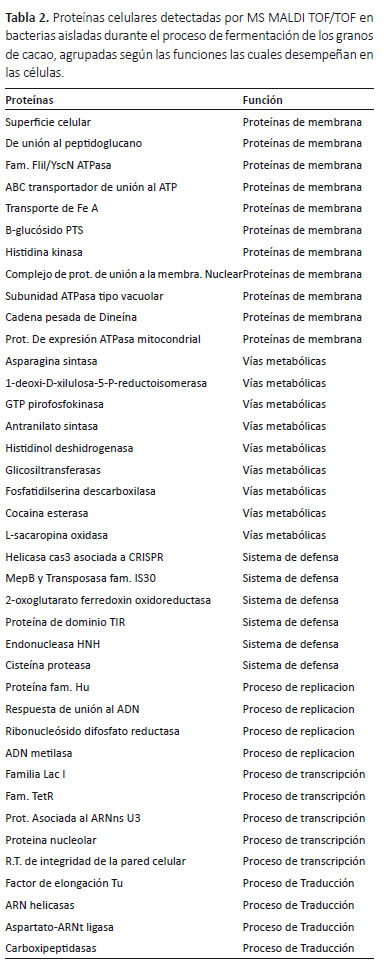
Shotgun de las cepas aisladas por MALDI TOF/TOF MS.- Por espectrometría de masas MALDI TOF/TOF se obtuvieron secuencias de proteínas celulares de L. plantarum, L. fermentum y Bacillus cereus agrupadas según las funciones las cuales desempeñan en las células. (Tabla 2).
Metaproteómica de co-cultivos de granos de cacao en proceso de fermentación.
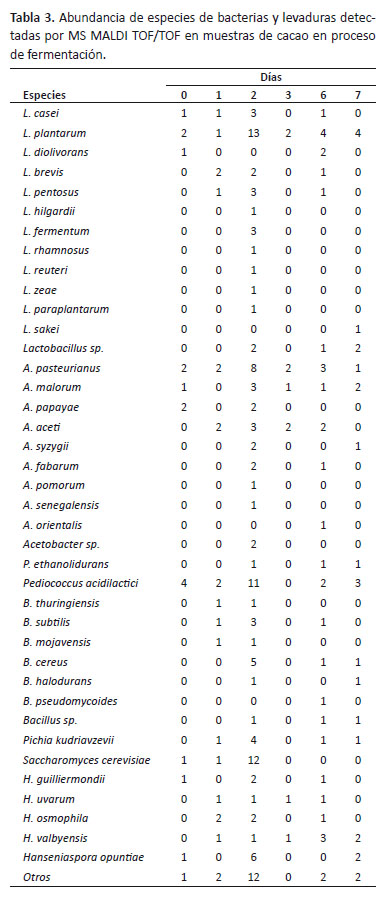
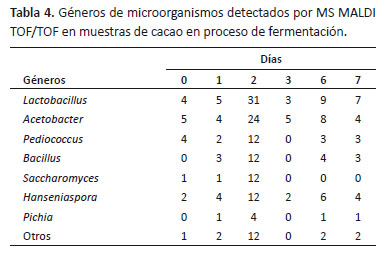
El análisis metaproteómico se basó en identificar los microorganismos presentes en los granos de cacao en proceso de fermentación a través de dominios conservados en sus secuencias péptidas. Partiendo de ello, se pudieron identificar 37 especies de bacterias y 14 especies de levaduras. (Tablas 3 y 4).
Metaproteómica de granos fermentados y secos
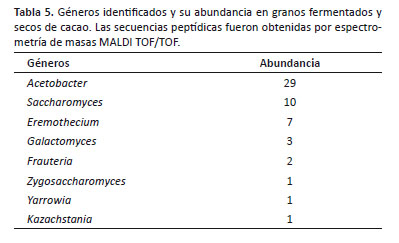
Adicionalmente se lograron identificar 16 especies entre bacterias y levaduras (Acetobacter y Saccharomyces fueron los géneros más abundante, respectivamente) a partir de granos fermentados y secos. (Tabla 5).
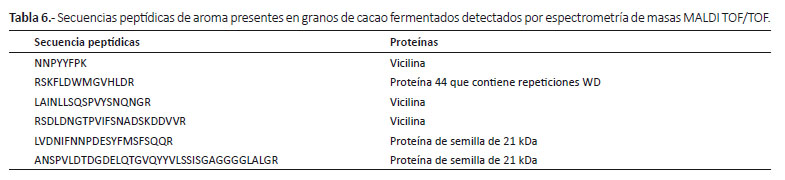
Asimismo se detectaron secuencias peptídicas del tipo globulinas y albuminas en el endospermo de los granos. Algunos péptidos detectados pertenecían a la proteína vicilina, responsable del aroma en los granos fermentados (Tabla 6).
Discusión
Bortolini et al. (2016) y Lefeber et al. (2011) trabajando en la fermentación del cacao reportaron como bacterias acido lácticas dominantes a Lactobacillus fermentum seguido de L. plantarum. Por el contrario, en esta investigación L. plantarum fue la especie dominante, lo que concuerda con los resultados de Melo et al. (2012) y Ho et al. (2015); además, también fue identificada P. acidilactici dentro de las bacterias acido lácticas. Bortolini et al. (2016) reportaron como bacterias acido acéticas dominantes a Acetobacter syzygii, A. pasteurianus, A. senegalensis; sin embargo en esta investigación fueron dominantes Acetobacter pasteurianus y A. papayae. Visintin et al. (2016) y Ho et al. (2015) también han reportado a A. pasteurianus como especie de bacteria acido acética dominante en el proceso de fermentación de los granos de cacao.
Visintin et al. (2016) y de Melo et al. (2012) identificaron levaduras amplificando la región ITS (Espaciador de Transcrito Interno) encontrando a Saccharomyces cerevisiae como la levadura más redundante en el proceso de fermentación. Por el contrario, aquí reportamos como especie dominante a Pichia kudriavzevii, lo cual coincide con los resultados obtenidos por Ho et al. (2015), Ho et al. (2014), los cuales además reportan a otras levaduras dominantes como Kluyveromyces marxianus y Hanseniaspora guilliermondii, ésta última detectada por nosotros mediante MS MALDI TOF/TOF).
Adicionalmente, reportamos la presencia de Bacillus cereus y B. thuringiensis por ADNg y B. subtilis detectado sólo por proteómica, mientras que Ho et al. (2014) y Melo et al. (2012) reportaron a Bacillus subtilis dentro de la microbiota presente en el proceso de fermentación de granos de cacao.
La identificación de microorganismos a nivel de ADN es un método muy costoso y que demanda de mucho tiempo para obtener resultados; sin embargo, en los últimos años, la espectrometría de masas como técnicas de identificación ha demostrado ser un método rápido, sensible y de bajo costo a comparación con otras técnicas. Miescher et al. (2016) y Pedrozo et al. (2017) realizaron estudios con espectrometría de masas MALDI TOF/TOF para la identificación de microorganismos en procesos fermentativos de granos de cacao, y sus resultados coincidieron con los nuestros, como por ejemplo Hanseniaspora opuntiae, Pichia kudriavzevii, Saccharomyces cerevisiae, Lactobacillus plantarum, L. fermentum, Pediococcus acidilactici, Acetobacter pasteurianus.
Andrés-Barrao et al. (2012) analizando el proteoma expresado de Acetobacter pasteurianus, para estudiar la fermentación acido acética, encontraron proteínas de shock térmico como GrpE, DnaK, DnaJ, GroES y GroEL, las cuales ayudan a proteger a otras proteínas de la desnaturalización (chaperonas). En nuestra investigación, estas proteínas, además de la chaperonina de 10KDa, también fueron identificadas (Apéndice 2), detectándose solamente en el grupo de bacterias acido acéticas conformadas por las especies Acetobacter pasteurianus A. papayae, A. malorum, A. ghanensis y A. pomorum.
También detectamos por espectrometría de masas MALDI TOF/TOF en el endospermo de granos de cacao fermentados y secos, péptidos derivados de la proteína globular de almacenamiento de la clase vicilina, los cuales han sido estudiados por espectrometría de masas en diferentes trabajos (v.g. Janek et al. 2016, Kumari et al. 2016). Asimismo, se detectamos la presencia de péptidos correspondientes a una proteína albúmina de 21KDa.
Literatura citada
Andrés-Barrao C., M. Saad, M.L. Chappuis, et al. 2012. Proteoma analysis of Acetobacter pasteurianus during acetic acid fermentation. Journal of Proteomics, 75, 1701-1717. https://doi.org/10.1016/j.jprot.2011.11.027. [ Links ]
Arana-Sánchez A., I. Segura-García, M. Kirchmayr, et al. 2015. Identification of predominant yeasts associated with artisan Mexican cocoa fermentations using culture-dependent and culture-independent approaches. World Journal of Microbiology and Biotechnology, 359-369. https://doi.org/10.1007/s11274-014-1788-8. [ Links ]
Ardhana M., & G Fleet. 2003. The microbial ecology of cocoa bean fermentations in Indonesia. International Journal of Food Microbiology, 87-99, https://doi.org/10.1016/s0168-1605(03)00081-3. [ Links ]
Böhme K., I. Fernández, M. Pazos, et al. 2013. Identification and classification of seafood-borne pathogenic and spoilage bacteria: 16S rRNA sequencing versus MALDI-TOF MS fingerprinting. Electrophoresis, 877-887. https://doi.org/10.1002/elps.201200532. [ Links ]
Bortolini C., V. Patrone, E. Puglisi, et al. 2016. Detailed analyses of the bacterial populations in processed cocoa beans of different geographic origin, subject to varied fermentation conditions. International Journal of Food Microbiology, https://doi.org/10.1016/j.ijfoodmicro.2016.07.004 [ Links ]
Camu N., T. De Winter, K. Verbrugghe, et al. 2007. Dynamics and biodiversity of popilations of lactic acid bacteria and acid acetic bacteria involved in spontaneous heap fermentation of cocoa beans in Ghana. Applied and Environmental Microbiology, 1809-1824. https://doi.org/10.1128/aem.02189-06. [ Links ]
Camu N., T. De Winter, S. Addo, et al. 2008. Fermentation of cocoa beans: influence of microbial activities and polyphenol concentrations on the flavour of chocolate. Journal Science Food Agriculture, 2288-2297, https://doi.org/10.1002/jsfa.3349. [ Links ]
Ho V.T.T., J. Zhao & G. Fleet. 2014. Yeast are essential for cocoa bean fermentation. International Journal of Food Microbiology 205: 54-67. Australia. https://doi.org/10.1016/j.ijfoodmicro.2013.12.014. [ Links ]
Ho V.T.T., J. Zhao & G. Fleet. 2015. The effect of lactic acid bacteria on cocoa bean fermentation. International Journal of Food Microbiology 205: 54-67. Australia, https://doi.org/10.1016/j.ijfoodmicro.2015.03.031 [ Links ]
Janek K., A. Niewienda, J. Wöstemeyer, et al. 2016. The cleavage specificity of the aspartic protease of cocoa beans involved in the generation of the cocoa-specific aroma precursors. Food Chemistry, 211, 320-328. https://doi.org/10.1016/j.foodchem.2016.05.033. [ Links ]
Kumari N., K. Kofi, S. Grimbs, et al. 2016. Biochemical fate of vicilin storage protein during fermentation and drying of cocoa beans. Food Research International 90, 53-65. https://doi.org/10.1016/j.foodres.2016.10.033. [ Links ]
Lefeber T., W. Gobert, G. Vrancken, et al. 2011. Dynamics and species diversity of communities of lactic acid bacteria and acetic acid bacteria during spontaneous cocoa bean fermentation in vessels. Food Microbiology 28: 457-464. https://doi.org/10.1016/j.fm.2010.10.010. [ Links ]
Lima L.J.R., M.H. Almeida, M.J. Rob Nout, et al. 2011. Theobroma cacao L., "The food of the gods": quality determinants of commercial cocoa beans with particular reference to the impact of fermentation. Critical Reviews in Food. https://doi.org/10.1080/10408391003799913. [ Links ]
Melo G., K. Teixeira, E. Gonzaga, et al. 2012. Spontaneous cocoa bean fermentation carried out in a novel-design stainless steel tank: Influence on the dynamics of microbial populations and physical–chemical properties. International Journal of food Microbiology 121-133. https://doi.org/10.1016/j.ijfoodmicro.2012.11.018. [ Links ]
Miescher S., S. Freimüller & C. Gantenbein-Demarchi. 2016. High-throughput identification of the microbial biodiversity of cocoa bean fermentation by MALDI-TOF MS. Institute of Food and Beverage Innovation, Wändenswil, Switzerland. https://doi.org/10.1111/lam.12621. [ Links ]
Moreira I., M. Pedrozo, W. Ferreira, et al. 2013. Microbial succesion and the dynamics of metabolites and sugars during the fermentation of three different cocoa (Theobroma cacao L.) hybrids . Food Research International:9-17. https://doi.org/10.1016/j.foodres.2013.06.001. [ Links ]
Pedrozo M., L. de Castro, P. Efraim, et al. 2017. Cocoa fermentation: microbial identification by MALDI-TOF MS, and sensory evaluation of produced chocolate. LWT-FoodScience and Technology 77:362-369. https://doi.org/10.1016/j.lwt.2016.11.076. [ Links ]
Pirovani C., H. Santos, R. Reboucas, et al. 2008. Protein extraction for proteome analysis from cacao leaves and meristems, organs infected by Moniliophthora perniciosa, the causal agent of the witches’ broom disease. Electrophoresis Journal, 2391-2401, doi.org/10.1002/elps.200700743.
Quintela‐Baluja M., K. Böhme, I. C. Fernández‐No, et al. 2013 Characterization of different food‐isolated Enterococcus strains by MALDI‐TOF mass fingerprinting. Electrophoresis. 34(15):2240-2250. https://doi.org/10.1002/elps.201200699. [ Links ]
Santos T., J.L. Capelo, H.M. Santos, et al. 2015. Use of MALDI-TOF mass spectrometry fingerprinting to characterize Enterococcus spp. and Escherichia coli isolates. Journal of proteomics 127: 321-331. https://doi.org/10.1016/j.jprot.2015.02.017. [ Links ]
Schmidt F., T. Fiege, H. Hustoft, et al. 2009. Shotgun mass mapping of Lactobacillus species and subspecies from caries related isolates by MALDE-MS. Proteomics:1994-2003. https://doi.org/10.1002/pmic.200701028. [ Links ]
Shevchenko A., H. Tomas, J. Havlis, et al. 2006. In-gel digestion for mass spectrometric characterization of proteins and proteomes. Nature:2856-2860. https://doi.org/10.1038/nprot.2006.468. [ Links ]
Sun L., K. Teramoto, H. Sato, et al. 2006. Characterization of ribosomal proteins as biomarkers for matrix-assisted laser desorption/ionization mass spectral identification of Lactobacillus plantarum. Rapid Communication in Mass Spectrometry:3789-3798. https://doi.org/10.1002/rcm.2801. [ Links ]
Visintin S., V. Alessandria, A. Valente, et al. 2015. Molecular identification and physiological characterization of yeasts, lactic acid bacteria and acetic acid bacteria isolated from heap and box cocoa bean fermentations in West Africa. International Journal of Food Microbiology:69-78. https://doi.org/10.1016/j.ijfoodmicro.2015.09.004. [ Links ]
Wickramasuriya A. & J. Dunwell. 2018. Cacao biotechnology: current status and future prospects. Plant Biotechnology Journal 16:4-17. https://doi.org/10.1111/pbi.12848. [ Links ]
Agradecimientos:
A Verónica Sernaqué, Segundo Astudillo, Cesar Mogollón, Cesar Chanta, Petter Baca y Pedro Masías quienes apoyaron en la recolección de muestra y trabajo en laboratorio. A Rubén Carrasco y Abimel López ingenieros encargados de ARPROCAT. A Benoit Diringer y Mercedes Oyola que con sus observaciones contribuyeron a mejorar el manuscrito.
Conflicto de intereses:
Los autores no incurren en conflictos de intereses.
Rol de los autores:
JM-G, ES-P: recolección y procesamiento de las muestras. JM-G, ES-P, EM-D, EM-M: análisis de resultados, redactaron, revisaron y aprobaron el manuscrito.
Fuentes de financiamiento:
Proyecto de investigación fue financiado por beca Ciencia Activa del CONCYTEC en colaboración con la Universidad Nacional de Tumbes e Inca Biotec.
Aspectos éticos / legales:
Los autores declaran que no se violaron aspectos éticos. Los permisos de colecta se solicitaron al presidente de ARPROCAT.
Otros datos de los autores / biografía:
1 Universidad Nacional de Tumbes. Programa de maestría de Ciencia Activa en Biotecnología Molecular. Tumbes.
ORCID Erick A. Suárez-Peña: 0000-0003-0137-8251
ORCID Emmerik Motte D.: 0000-0001-9577-2887
Citación:
Machuca-Guevara J.I., E.A. Suárez-Peña, E. Motte Darricau, E.L. Mialhe-Matonnier. 2019. Caracterización molecular de los microorganismos presentes durante el proceso fermentativo de los granos de cacao (Theobroma cacao). Revista peruana de biología 26(4): 535 - 542 (Diciembre 2019). doi: http://dx.doi.org/10.15381/rpb.v26i4.17220
Correspondencia:
*Autor para correspondencia
JIM-G: juanamachucag310@gmail.com
EASP: esupeantonio@gmail.com
ELM-M: ericmialhe@yahoo.fr
Presentado: 25/10/2018
Aceptado: 26/07/2019
Publicado online: 16/12/2019