Introducción
Después de la domesticación, la variación fenotípica del color fue una de las primeras características consideradas en la selección artificial (Cieslak et al. 2011, Linderholm y Larson 2013, Librado et al. 2017). Los genes MC1R (receptor 1 de melanocortina) y ASIP (proteína de señalización agouti) son los principales genes que intervienen en la síntesis y distribución de eumelanina y feomelanina en el vellón de alpacas (Feeley & Munyard 2009, Cieslak et al. 2011, Marín et al. 2018), pero en otros animales se han descrito más de 150 genes asociados a la expresión del color (Montoliu et al. 2011). Estudios moleculares sugieren que la domesticación de la vicuña se inició hace 6000 7000 años en los andes centrales del Perú, siendo la alpaca descendiente de la vicuña (Kadwell et al. 2001, Gentry et al. 2004, Fan et al. 2020). Posteriormente la selección de alpacas por su vellón blanco favoreció la fijación de alelos específicos en los genes MC1R y ASIP (Marín et al. 2018). La variación alélica del gen MC1R fue descrita mediante los PNSs y sus haplotipos (Powell et al. 2008, Feeley & Munyard 2009, Guridi et al. 2011, Chandramohan et al. 2015, Daverio et al. 2016).
En las últimas décadas las empresas industriales han demandado fibra blanca, favoreciendo el incremento de alpacas blancas, light fawns (LFs) y las tonalidades del marrón (Oria et al. 2009). Esta tendencia de blanqueo de la fibra conllevó a la disminución de alpacas negras y grises (Oria et al. 2009, Jones et al. 2019). Sin embargo, en algunas regiones del Perú (Apurímac, Huancavelica, Cusco, Puno y Ayacucho) se mantienen rebaños de alpacas con fibra de diversos colores (Oria et al. 2009).
En consecuencia, es necesario generar información de marcadores moleculares de tipo PNSs para la selección de animales negros y así aumentar la producción de fibra de color natural y cubrir la demanda creciente en la industria textil. Los objetivos en el presente trabajo fueron cuantificar el color de la fibra e identificar los PNSs informativos del gen MC1R en alpacas marrones y negras.
Material y métodos
Medición cuantitativa del color
Nueve muestras de fibra de vicuña fueron obtenidas del Centro Ecológico Recreacional Taraccasa de la Municipalidad Provincial de Abancay, con autorización RDG N° 270-2019-MINAGRI-SERFOR-DGGSPFFS y otras cinco muestras fueron obtenidas de vellones esquilados, procedentes del Laboratorio de Fibras Textiles UNALM.
Los animales fueron muestreados al azar, previo un riguroso análisis de la genealogía (coancestría) de cada animal, eligiéndose a los animales que provenían de rebaños diferentes (sin evidencias de consanguinidad). Las muestras de fibra de alpaca fueron obtenidas del costillar medio de 79 animales del Fundo Pacomarca (Puno), evaluadas visualmente y registradas subjetivamente (Figura 1), que corresponden a 22 alpacas de color café claro (CC), 19 de café oscuro marrón (COM), 11 de café oscuro negro (CON) y 27 alpacas de color negro (N). Además, para una mejor caracterización de los fenotipos de color se registró la pigmentación de ojos, mucosas, pezuñas y piel, presencia de canas y manchas.
Por otro lado, el vellón de vicuña varió de una pigmentación clara en la espalda y ligeramente más intensa en el costillar (manto). Mientras que en la parte ventral (barriga-pecho) se observó mayor proporción de cerdas blancas sin pigmentación.
Las muestras de fibra fueron lavadas antes de su medición. El equipo Chroma Meter CR-210 (Konica Minolta) se calibró con un patrón estándar según las especificaciones del manual, siguiendo el procedimiento CIE L*a*b* (CIE 2004) y posteriormente se realizaron las mediciones de L*, a*, b* en 14 vicuñas y 79 alpacas Huacaya. L* = luminosidad, a* = [saturación de color rojo (+) al verde (-)] y b* = [saturación de color amarillo (+) al azul (-)]. Adicionalmente se calculó la intensidad = [(a*+ b* )05] y la tonalidad usando la función arco tangente = [tan (b*/ a*)], según lo propuesto por Druml et al. (2018). Los valores obtenidos fueron registrados en el software PacoPro Versión 5.10 (http://pacomarca.com/es/programagenetico/).
Extracción de ADN
El ADN fue extraído de 200 μL de sangre usando un kit comercial (Quick-DNA™ Miniprep Plus Kit). De esta manera se obtuvo el ADN genómico de 79 alpacas y dos vicuñas (control). La concentración y la calidad del ADN se determinaron por espectrofotometría con un NanoDrop 8000 en el Laboratorio de Biología Molecular y Genómica del Instituto Nacional de Innovación Agraria (INIA), Lima. La integridad se evaluó mediante electroforesis en gel de agarosa al 1% en el Instituto de Bioquímica y Biología Molecular de la Universidad Nacional Agraria La Molina.
Amplificación por reacción en cadena de polimerasa (PCR) y secuenciación del gen MC1R
La región codificante completa del gen MC1R se amplificó mediante la técnica de PCR usando un termociclador (Mastercycler® flexlid-Eppendorf), con dos cebadores que incluye el directo: GCTGCGAAGTGACCAGACTC y el reverso: CTCCTCATTGCCAAGTAACTGC (Daverio et al. 2016) a una concentración de 0.20 μM.
Se empleó máster mix comercial (con Taq polimerasa) amaR OnePCR (genedirex.com/wp-content/ uploads/2018/11/SM213-0250.pdf). El perfil de ciclado incluyó tres minutos de desnaturalización inicial a 94 °C, seguido de 30 ciclos: 30 segundos de desnaturalización a 94 °C, 1 minuto de hibridación a 61°C, 1 minuto de extensión a 72 °C y una extensión final de 5 minutos a 72 °C. Los productos de PCR del gen MC1R fueron purificados usando un kit comercial EnzSAP™ PCR Clean-up Reagent (biocat.com/bc/pdf/32-030v2_Flyer_EnzSAP_ PCR_Clean-up_Reagent.pdf), los mismos posteriormente fueron secuenciados mediante el método de Sanger en Macrogen Inc (Seoul, Korea).
Identificación de polimorfismos y genotipado
El alineamiento de las secuencias se realizó usando el programa ClustalW (Thompson et al. 1994), para este estudio se tomó como referencia la secuencia del gen MC1R obtenida de las dos vicuñas control. Además, se consideró dos secuencias disponibles en GenBank de alpaca de color negro y marrón con número de acceso FJ502229 y FJ502230, respectivamente. El alineamiento y la identificación de polimorfismos (PNSs y deleción) se realizaron usando el software Geneious Versión 11.1.5 (https://www.geneious.com/). Los genotipos de las dos vicuñas fueron similares, mientras que las 79 secuencias de alpacas de color fueron diferentes, que resultaron del alineamiento múltiple.
Además, las dos secuencias codificantes (CDS) del gen MC1R de vicuña se compararon con 89 CDS de vicuñas (subespecie mensalis y vicugna) obtenidas por Marín et al. (2018), que incluye los números de accesos MH596180-MH596268, el alineamiento se realizó usando BLAST (Multiple Sequence Alignment Viewer 1.11.1) del servidor NCBI. Para esta comparación se tomó solamente 72 de 89 CDS reportadas por Marín et al. (2018), asumiendo el criterio del parámetro de porcentaje de similitud mayor al 99.50%. Del total de las secuencias generadas en este estudio (81 secuencias), se eligió tres secuencias representativas que corresponden a una vicuña, una alpaca marrón claro y otra alpaca negra fueron depositadas en GenBank, con los números de acceso: MW587025, MW587026 y MW587027, respectivamente.
Identificación de PNSs no sinónimos y haplotipos
Los PNSs no sinónimos resultan en la sustitución de aminoácidos. Considerando estos PNSs se predijo la estructura 2D del gen MC1R usando el software PROTTER v1.0 online tools (http://wlab.ethz.ch/protter/#). Los haplotipos fueron inferidos de los PNSs no sinónimos, para cada haplotipo se consideró una probabilidad de fase mayor a 0.9. La fase gamética de cada haplotipo fue determinada mediante el Algoritmo Bayesiano ELB usando el software Arlequín Versión 3.5.2.2 (Excoffier & Lischer 2010).
Análisis de asociación entre los PNSs no sinónimos y fenotipos de color
La asociación de los cinco PNSs con los fenotipos de color evaluados mediante colorimetría se determinó mediante la prueba exacta de Fisher, usando el software R Versión 3.6.3 (R Core Development Team 2020).
Resultados y discusión
Caracterización cuantitativa del color
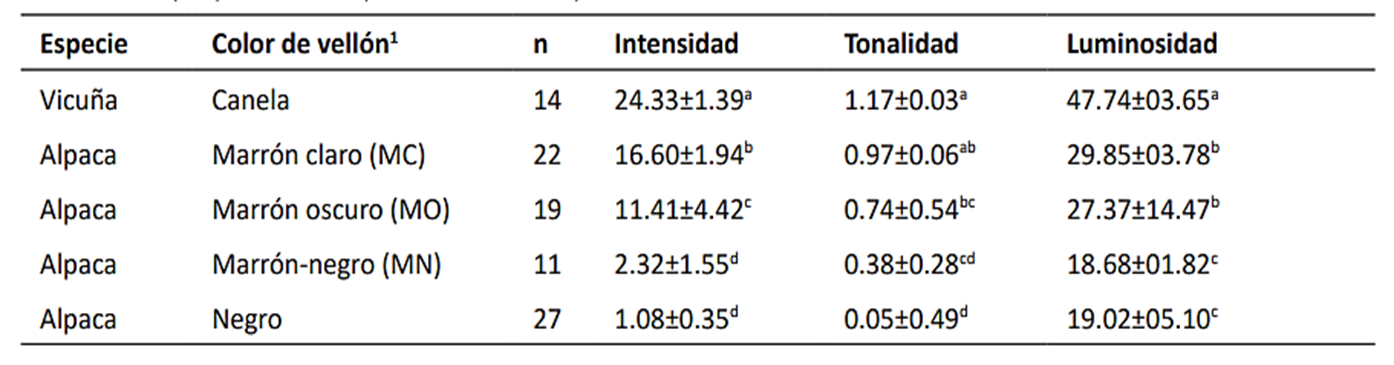
Después del análisis cuantitativo de la fibra, el fenotipo del vellón de las vicuñas resultó diferente a lo descrito en alpacas. Los fenotipos iniciales de color evaluados visualmente en alpacas quedaron reagrupados en tres fenotipos: fenotipo 1: café claro (CC), fenotipo 2: café oscuro marrón (COM) y el fenotipo 3: incluyó el café oscuro negro (CON) y negro (N), de acuerdo con los valores de intensidad (Tabla 1).
En el vellón marrón claro de alpaca, los valores promedio de luminosidad e intensidad fueron 29.85 y 16.60, respectivamente; estos valores fueron ligeramente superiores a 27.37 y 11.41 observados en el vellón marrón oscuro de alpaca. En alpacas marrones Guridi et al. (2011) reportaron un mayor valor de luminosidad (36.25) comparado a nuestros resultados obtenidos en animales de fenotipo marrón oscuro y marrón claro. En animales negros se observó baja luminosidad entre 18.62 y 19.02, ligeramente superior a 17.23 reportado por Guridi et al. (2011). Además, en el negro se observó baja intensidad (entre 1.08 y 2.32) similar a los reportes de Lupton et al. (2006) y Guridi et al. (2011).
En este trabajo no se cuantificaron los niveles de melanina, sin embargo, los valores obtenidos de CIE L*a*b*, principalmente la luminosidad e intensidad sugieren una relación positiva con la pigmentación feomelánica (marrón claro y oscuro), por el contrario, se observó una relación inversa con la pigmentación eumelánica del animal. En ovinos, caprinos y equinos se ha indicado que la eumelanina es responsable de la pigmentación negro/ marrón y la feomelanina del rojo/amarillento (Cieslak et al. 2011). Sin embargo, Sponenberg et al. (1988) y Cecchi et al. (2007) en llamas, Cecchi et al. (2011) y Cransberg et al. (2013) en alpacas, señalaron la posible ausencia de la pigmentacion eumelánica en animales de fenotipo marrón claro. En consecuencia, no es fácil diferenciar las alpacas de fenotipo marrón con pigmentación feomelánica versus eumelánica.
Tabla 1 Promedio ± desviación estándar de los valores de intensidad, tonalidad y luminosidad en fibra de vicuña y alpaca Huacaya cuantificadas por colorimetría usando Chroma Meter CR-210.
1 Fenotipos determinados por evaluación visual.
*Letras diferentes en cada columna indican diferencias estadísticas para los parámetros cuantificados que definen los fenotipos.
Prueba de Tukey, comparación múltiple de medias, α=0.05.
El color de fibra de la vicuña mostró mayor luminosidad (47.74) e intensidad (24.33) respecto a la alpaca marrón claro, aunque sus tonalidades fueron similares (Tabla 1). El patrón de pigmentación visualmente observado en el vellón de vicuña (Vicugna vicugna mensalis) fue homogéneo, aunque la intensidad del color puede aumentar con la edad (Quispe et al. 2019). Los fenotipos de las vicuñas también fueron homogéneos, aunque Marín et al. (2018) reportaron una tonalidad más clara en las vicuñas del sur (Vicugna vicugna vicugna), que atribuyeron a la mutación no sinónima c.265A>G y dos sinónimas c.72C>G y c.618G>A.
Variación de secuencia y haplotipos en el gen MC1R
En las vicuñas y las alpacas el gen MC1R presentó un solo exón de 954 pb, que constituye la secuencia codificante. Dentro de la secuencia codificante del gen MC1R de alpaca se observó alta variabilidad alélica, que incluye nueve PNSs y una deleción (Tabla 2). Esta deleción de 4pb (c.224_227 del) se observó en estado heterocigota en el 26.32% de los animales del fenotipo negro y 19.51% del fenotipo marrón, siendo similar a los reportes de Powell et al. (2008) y Chandramohan et al. (2015). La secuencia codificante del gen MC1R obtenida para las dos vicuñas mostró alta similitud (entre 99.58 y 100%) al comparar con las 72 secuencias de vicuña depositadas en GenBank (Marín et al. 2018). Dentro de la secuencia codificante del gen MC1R, que corresponde a las dos vicuñas, no se observó la deleción heterocigota de 4pb (c.224_227 del) similar al reporte de Marín et al. (2018).
En una muestra de las 19 alpacas marrón oscuro se observó un PNS sinónimo c.72C>G y otro no sinónimo c.265A>G (p.M89V) con genotipo heterocigota, que resulta similar a lo observado por Marín et al. (2018) en dos alpacas y las vicuñas del sur (Vicugna vicugna vicugna). Por otro lado, en alpacas marrones de Huancavelica Guridi et al. (2011) reportaron tres PNSs con sustituciones no sinónimas c.865C>T (p.C289G), c.874G>A (p.I292V) y c.886T>C (p.L296F), estos PNSs no fueron observados en la población de animales que consideramos en nuestro estudio.
De los nueve PNSs ubicados en la secuencia de codificación de MC1R (Tabla 2), cinco PNSs mostraron sustituciones no sinónimas, c.82A>G, c.259G>A, c.376G>A, c.587T>C, c.901C>T, que producen sustituciones de aminoácidos en las posiciones p.T28A, p.M87V, p.G126S, p.F196S y p.R301C, respectivamente. Estas sustituciones se encuentran localizadas en el extremo amino extracelular (p.T28A), dominios transmembrana TM2 (p.M87V), TM3 (p.G126S), TM5 (p.F196S) y el extremo carboxilo intracelular (p.R301C). Adicionalmente, se observó una nueva sustitución (p.R229C) en el tercer loop intracelular (Fig. 2), como resultado de un nuevo PNS no sinónimo c.685C>T, no incluido en la Tabla 2.
Los cuatro PNSs con sustituciones sinónimas, c.126C>T, c.354C>T, c.618G>A, c.933A>G fueron similares a los reportes de Guridi et al. (2011) y Chandramohan et al. (2015) en alpacas negras y marrones de Perú. Estos polimorfismos también fueron reportados por Powell et al. (2008) en alpacas de Estados Unidos, Feeley y Munyard (2009) y Feeley (2015) en alpacas australianas y por Daverio et al. (2016) en llamas de Argentina. Adicionalmente, en las regiones no traducidas 5´UTR y 3´UTR del gen MC1R se identificaron dos PNSs sinónimos c.-42C>G y c.*5T>C, respectivamente.
Considerando los cinco PNSs no sinónimos con una probabilidad de fase mayor de 0.9 se infirieron ocho haplotipos y sus respectivas frecuencias (Tabla 3), estos haplotipos se encuentran dentro de los 24 haplotipos teóricos analizados. El haplotipo 1: AAATC (MC1R*negro) se observó con más frecuencia en los negros (0.58) similar a lo observado por Chandramohan et al. (2015) y Daverio et al. (2016). El haplotipo 2: AGGTC (0.14) se observó en el grupo de alpacas negro y marrón, mientras que los haplotipos 3, 4 y 5 (GGGTT, GGGTC y GGGCC) solo se observaron en alpacas marrones. El haplotipo 4: GGGTC (0.07) corresponde al alelo silvestre de la vicuña (MC1R*vicuña).
Asociación entre PNSs y genotipos del gen MC1R con los fenotipos de color
Los genotipos AAATC/AAATC, AAATC/GGGCC y GGGTC/GGGTC de los cinco PNSs no sinónimos mostraron una asociación significativa (prueba exacta de Fisher p<0.001) con los fenotipos de color evaluados mediante colorimetría. Observándose mayor frecuencia del genotipo AAATC/AAATC en alpacas negra y marrón oscuro, mientras que el genotipo GGGTC/GGGTC se observó en 2 vicuñas de este estudio y 72 secuencias de vicuña reportadas por Marín et al. (2018), también este genotipo se observó en 4 muestras de 22 alpacas del fenotipo marrón claro (Figura 2).
Tabla 2 Genotipos identificados de los PNSs, con presencia/ausencia de una deleción (c.224_227del) en la secuencia codificante y las regiones no traducidas (5´UTR y 3´UTR) del gen MC1R en 2 vicuñas y 79 alpacas Huacaya. Primera fila muestra los PNSs, deleción heterocigota de 4pb (-D4) y sin deleción (-), en negrita se muestran los PNSs no sinónimos. Segunda fila muestra las sustituciones de aminoácidos, que incluye un marco abierto de lectura (ORF) debido a la deleción (c.224_227del). Última columna muestra la cantidad de animales con sus respectivos fenotipos de color, 22 marrón claros (MC), 19 marrón oscuros (MO), 11 marrón-negros (MN) y 27 negros.
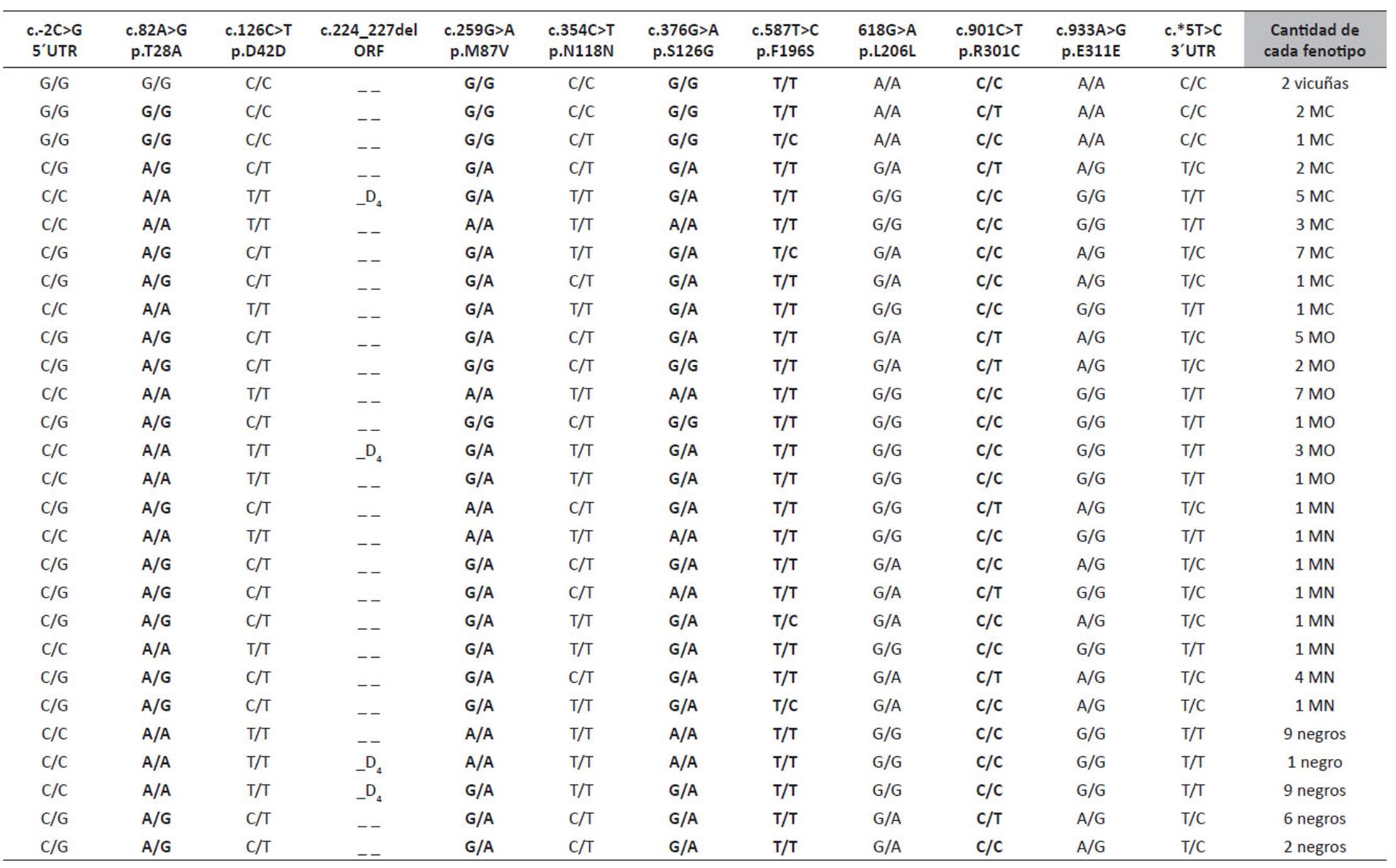
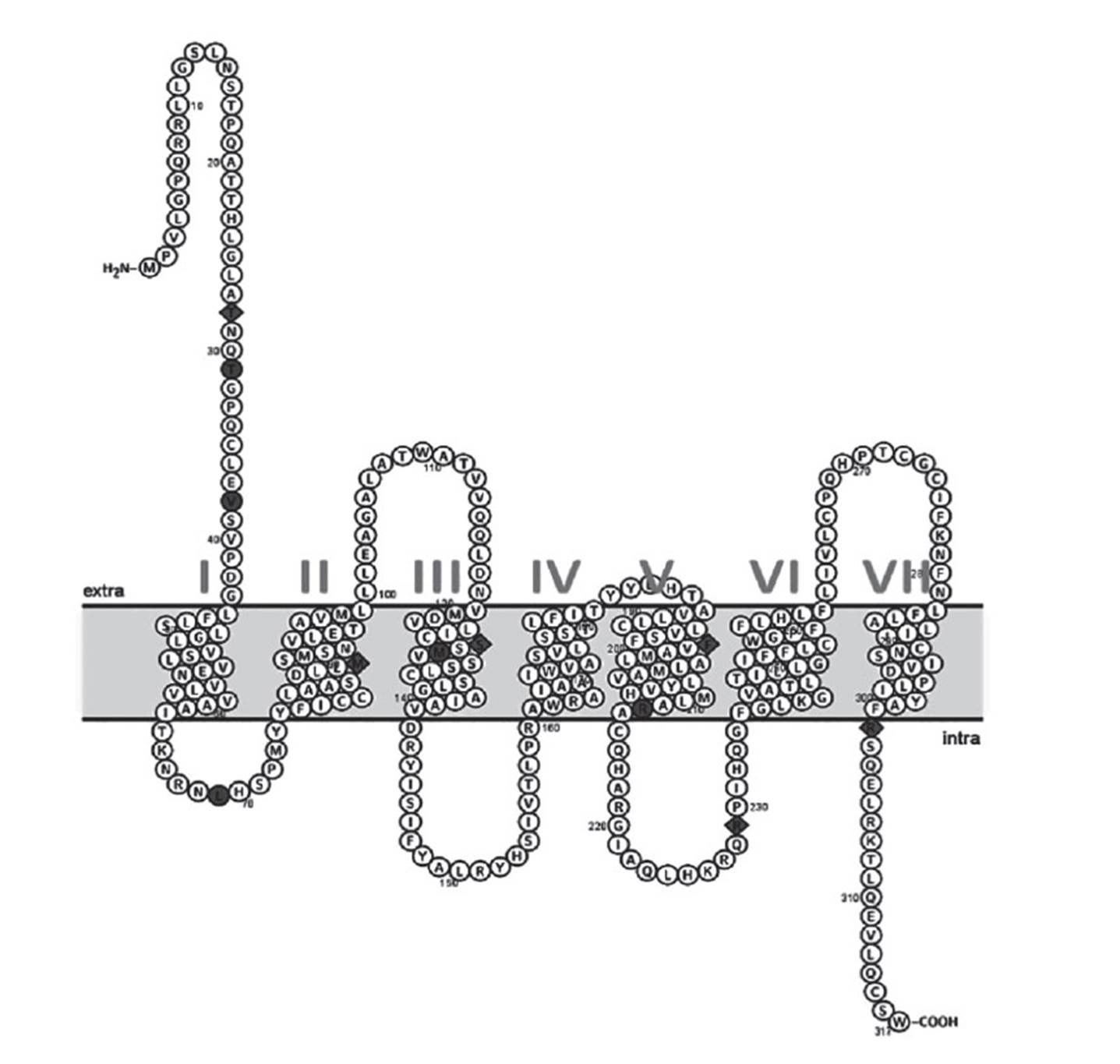
Figura 2 Estructura 2D del gen MC1R. Los aminoácidos sombreados en coco (♦) corresponde a sustituciones no sinónimas identificada en alpacas, en círculo y sombreado (●) corresponde a sustituciones no sinónimas reportada por Daverio et al. (2016) en llamas. I-VII son dominios transmembrana (TM), con extremo amino terminal extracelular (H2N) y extremo carboxilo terminal intracelular (-COOH).
Tabla 3 Definición de ocho haplotipos con sus frecuencias en el gen MC1R de 2 vicuñas y 79 alpacas Huacaya de color.
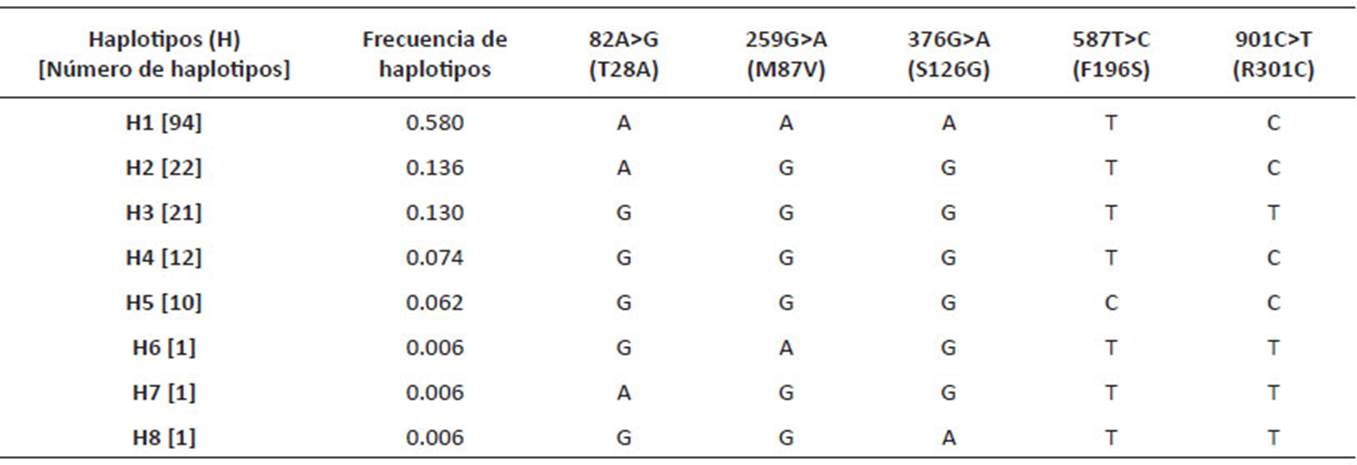
*Primera fila muestra los PNSs no sinónimos elegidos para construir los haplotipos y entre paréntesis se muestran las sustituciones de aminoácidos, respectivamente.
Feeley y Munyard (2009) consideraron solo tres de los 5 PNSs no sinónimos, c.82A>G, c.376G>A y c.901C>T, ellos observaron el genotipo AA AA 82 376 901 CC en la mayoría de las alpacas negras, siendo equivalente a nuestros hallazgos, mientras que en alpacas blancas y algunas de color light fawn ellos observaron el genotipo opuesto 82GG 376 GG901 TT.
Los genotipos del PNS c.82A>G mostraron una asociación significativa con los fenotipos de color evaluados mediante colorimetría (prueba exacta de Fisher p≤0.02), aunque Powell et al. (2008) no observaron ninguna asociación. En este PNS c.82A>G se identificó una sustitución p.T28A en el extremo N H de la secuencia de aminoácidos (Fig. 2), además se sabe que las sustituciones en este dominio son importantes para el funcionamiento normal de la proteína del gen MC1R (Garcia-Borron et al. 2005).
En ocho muestras de 22 alpacas marrón claro y en dos muestras de 11 alpacas marrón-negro se identificó una sustitución no sinónima c.587T>C (p.F196S) con genotipo T/C, sin embargo, el resto de las alpacas marrones y todas las alpacas negras al igual que las dos vicuñas mostraron el genotipo T/T (sin mutación) y no se observó la variante homocigota C/C en ninguno de los animales muestreados. Este polimorfismo fue también reportado por Powell et al. (2008) en alpacas de Estados Unidos y por Daverio et al. (2016) en llamas de Argentina. Considerando los genotipos T/T y T/C se observó una asociación significativa con los fenotipos de color evaluados mediante colorimetría (prueba exacta de Fisher p<0.001). Por otro lado, estudios del análisis de segregación reportados por Valbonesi et al. (2011) y Bathrachalam et al. (2019) indican que el fenotipo marrón claro sería dominante sobre el fenotipo negro.
En los otros tres PNSs, c.259G>A, c.376G>A y c.901C>T no se han observado asociaciones, probablemente debido a la presencia de heterocigotas en los tres fenotipos de color. El haplotipo constituido por los PNSs c.259G>A y c.376G>A no mostró ninguna asociación con los fenotipos de color. Para el PNS c.901C>T se observó solo dos genotipos C/C y C/T y no se observó la variante homocigota T/T en ninguno de los animales muestreados similar al reporte de Guridi et al. (2011). Por otro lado, en la mayoría de las alpacas de color blanco de otras poblaciones se ha reportado el genotipo T/T y en algunas alpacas blancas de Estados Unidos se observó la variante heterocigota (T/C) (Powell et al. 2008), asimismo en Australia (Feeley & Munyard 2009) y Perú (Chandramohan et al. 2015). Además, en el camello dromedario blanco también fueron reportados estos dos genotipos T/T y T/C (Almathen et al. 2018 y Alshanbari et al. 2019).
Inicialmente Powell et al. (2008) propusieron siete alelos para el gen MC1R, uno de ellos con pérdida de función (e) relacionado con la pigmentación feomelánica. Feeley y Munyard (2009) propusieron alelos dominantes (E) y recesivos (e). Sin embargo, se ha informado que estos alelos en combinación con los alelos del gen ASIP determinan el fenotipo negro o marrón (Chandramohan et al. 2013, Bathrachalam et al. 2019). En este estudio no se consideró la interacción epistática entre los genes MC1R y ASIP, pero se observó que un genotipo podría expresarse en más de un fenotipo o viceversa












 uBio
uBio 



