Introducción
La L-asparaginasa (E.C.3.5.1.1) hidroliza la L-asparagina a ácido L- aspártico y amonio (Shakambari et al. 2019). Se señala que esta enzima disminuye las concentraciones de L-asparagina sérica en pacientes con leucemia linfoblástica aguda, conllevando a la muerte de células cancerígenas (Cachumba et al. 2016). Sin embargo, también se ha observado reacciones negativas, como las causadas por algunas L-asparaginasas que presentan además actividad de L-glutaminasa, lo cual se relaciona con hepatitis, leucopenia, neurotoxicidad, hiperglicemia y coagulopatías (Offman et al. 2011; Fonseca et al. 2021). Asimismo, las L-asparaginasas comerciales muestran baja biodisponibilidad plasmática, desarrollo de anticuerpos y causan reacciones de hipersensibilidad en el 30% al 70% de los pacientes tratados (Zenatti et al. 2018).
La L-asparaginasa también se usa en la industria alimentaria para disminuir la concentración de acrilamida en alimentos termoprocesados, debido a que los azúcares reductores reaccionan con L-asparagina (Muttukumaru et al. 2017; Rifai & Saleh 2020). De esta manera, el tratamiento previo de estos alimentos con L-asparaginasa reduce los niveles de acrilamida en aproximadamente el 90% en papas fritas y productos de panificación, entre otros (Ciesarovát et al. 2009; Mohan et al. 2014).
La L-asparaginasa es producida por microorganismos, vegetales y animales, siendo los primeros la fuente más importante debido a su rápida producción, además de permitir una eficiente purificación de la enzima y su escalamiento (Cachumba et al. 2016). Entre los microorganismos utilizados en la producción de L-asparaginasa se encuentran los Streptomyces, Bacillus, Lysinibacillus, Staphylococcus, Rouxiella, Erwinia, Escherichia, Acinetobacter, Pseudomonas (Erva et al. 2018; Arévalo-Tristancho et al. 2019; Gilavand et al. 2019; Prihanto et al. 2019; Setiawan & Larasati 2019). Sin embargo, para lograr altos niveles de producción se requiere buena estabilidad en condiciones extremas, como la que han demostrado bacterias que mantienen actividad con alta eficiencia y termoestabilidad (Zuo et al. 2015). Así, microorganismos halotolerantes y halófilos poseen mecanismos celulares capaces de resistir elevadas concentraciones salinas y diferentes valores de pH y temperatura; además producen diversos metabolitos y enzimas (Abdelrazek et al. 2020; Aishwariyaa et al. 2020). Aunque los géneros bacterianos de ambientes halófilos como Halomonas, Marinobacter y Bacillus han sido caracterizados por la producción de L-asparaginasa, sigue pendiente la optimización de su producción y su posterior escalamiento y aplicación en la industria farmacéutica y alimentaria (Zolfaghar et al. 2019).
Por la importancia de la L-asparaginasa, en este trabajo se da a conocer la caracterización bioinformática y la optimización de la producción de L-asparaginasa producida por la cepa Bacillus sp. M62 aislada de las salinas de Maras (Cusco, Perú).
Material y métodos
Material biológico. La cepa Bacillus sp. M62 fue aislada de las salinas de Maras (13.303646°S, 72.154182°W), pertenece al cepario del Laboratorio de Biología Molecular de la Facultad de Farmacia y Bioquímica de la Universidad Nacional Mayor de San Marcos. Esta cepa es conservada a -20 °C en caldo tripticasa soya (NaCl 50 g, MgCl2.6H2O 5 g, MgSO4.7H2O 5.8 g, KCl 1.2 g, Tris 8 mM y CaCl2.2H2O 8 mM para 1 L, y glicerol 30% (v/v)) (Dyall-Smith et al. 2009).
Detección de producción de L-asparaginasa. Bacillus sp. M62 se cultivó a 37 °C por 72 h en medio M9 modificado: Na2HPO4 6 g/L, KH2PO4 2 g/L, NaCl 30 g/L, L-asparagina 5 g/L, MgSO4.7H2O 0.5 g/L, CaCl2.2H2O 0.15 g/L y glucosa 2 g/L, con azul de bromofenol 0.0075% a pH 7.4 (Gulati et al. 1997).
Producción enzimática. Bacillus sp. M62 se sembró en medio M9 modificado en proporción área/volumen de 5, inóculo 2% (v/v) (DO:1.0), se incubó a 37 °C con agitación a120 RPM y cultivó por 24, 48, 72 y 96 h. Después, se centrifugó a 7942 rcf por 15 min y se separó el sobrenadante (extracto crudo) que se usó para la cuantificación de la actividad enzimática extracelular. Asimismo, la biomasa fue lavada y resuspendida en buffer conteniendo Tris 50 mM, EDTA 10 mM, pH 7.5, sonicada durante 80 ciclos de 2 s activos y 5 s de descanso. Luego, se centrifugó a 9928 rcf por 25 min y el sobrenadante se usó para la cuantificación de la actividad enzimática intracelular.
Determinación de las actividades L-asparaginasa y L-glutaminasa. Se llevó a cabo según Shifrin y Parrott (1974). La determinación de ambas actividades se realizó mediante cuantificación del amonio liberado usando L-asparagina o L-glutamina 189 mM en buffer Tris 50 mM pH 8.6, que reaccionó con el reactivo de Nessler y se midió su absorbancia en un espectrofotómetro UV-visible (Eppendorf, Alemania) a 436 nm. Además, se realizó una curva de calibración usando sulfato de amonio, y se consideró una unidad internacional (UI) de actividad como la cantidad de enzima requerida para liberar 1 µmol de amonio por mL por min a pH 8.6 y 37 °C.
Cuantificación de proteínas. El método del ácido bicincónico fue utilizado para la cuantificación de proteínas. Las muestras fueron incubadas en microplacas de 96 pocillos por 30 min a 37 °C, después se leyó en un lector de microplacas (Infinite M200PRO, TECAN) a 562 nm.
Identificación molecular de la cepa. El ADN genómico se extrajo usando proteinasa K (0.3 µg/µL) y SDS (9 µg/µL), luego del cual fue purificado con una solución de fenol-cloroformo-alcohol isoamílico (25:24:1) y precipitado en etanol absoluto y acetato de sodio 3 M (Calderón-Toledo et al. 2021). El gen ribosómico 16S se amplificó mediante la reacción en cadena de la polimerasa (PCR) utilizando los primers F:5’-AGAGTTTGATCATGGCTCAG-3’ y R:5’-GGTTACCTTGTTACGACTT-3’. Las condiciones de la PCR fueron: desnaturalización inicial a 94 °C por 4 min, seguido de 35 ciclos de desnaturalización a 94 °C por 45 s, hibridación a 55 °C por 1 min, extensión a 72 °C por 45 s; finalizando con una extensión a 72 °C por 7 min (Canales et al. 2014). Los productos fueron separados por electroforesis en gel de agarosa 1% con buffer TBE 1X. Las secuencias obtenidas fueron analizadas mediante los programas Bioedit y Cap3, luego se identificó la similitud con las secuencias nucleotídicas del NCBI mediante el algoritmo BLAST (Huang and Madan 1999; Hall et al. 2011).
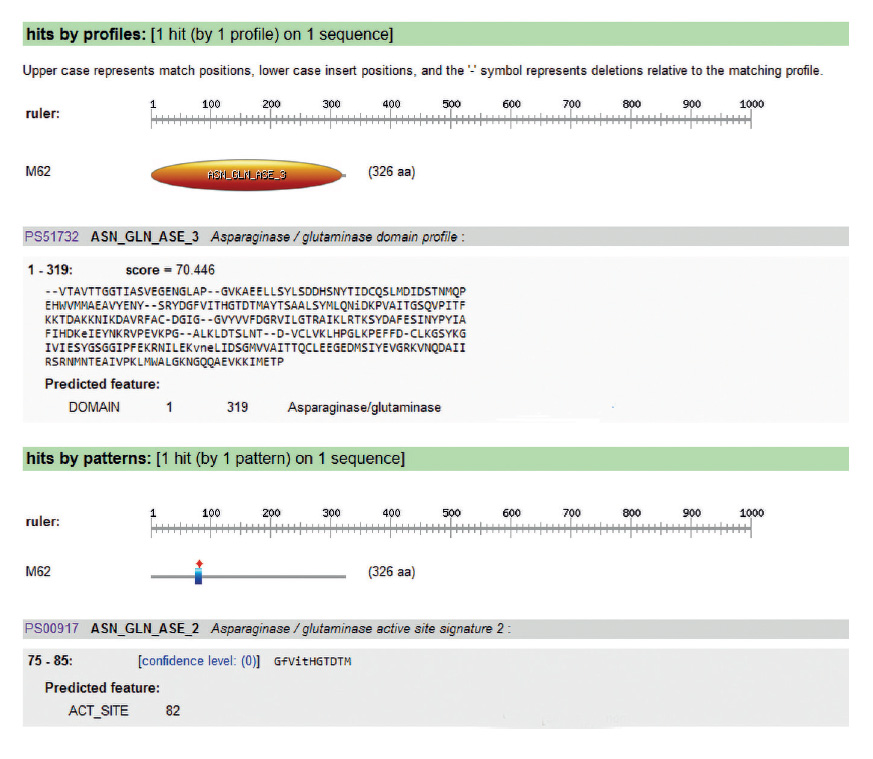
Obtención del gen que codifica para la L-asparaginasa y análisis bioinformático. El gen ansA3 se amplificó usando los primers ansA3 FP (5′-GAA CTC TGG GGA TCC ATG AAA AAG TTA CTG CTG TTG-3′) y ansA3 RP (5′-AGG TTC CAA GAATTC TTATAT GAT GATATC GTC TGC-3′) (Sudhir et al. 2014). Las condiciones de la reacción de PCR fueron similares a lo descrito para la identificación molecular con la diferencia en la etapa de hibridación a 59 °C por 30 s. El producto fue secuenciado y analizado utilizando los programas Bioedit, Cap3, GeneMark para determinar la secuencia aminoacídica, y el algoritmo BLASTP. Se realizó un alineamiento múltiple con las secuencias tipo pertenecientes al género Bacillus y se identificó el modelo evolutivo con mejor ajuste utilizando el programa MEGA (Kumar et al. 2018). Luego, se utilizó el método de inferencia filogenética de máxima verosimilitud utilizando el soporte estadístico Bootstrap con 1000 réplicas. Asimismo, se determinaron las regiones antigénicas mediante el programa predictor ANTIGENIC diseñado por EMBOSS (https://www.bioinformatics.nl/cgi-bin/emboss/antigenic). Además, el programa Expasy Prosite (https://prosite.expasy.org/cgi-bin/prosite) fue usado para identificar el sitio activo de la enzima. La estructura secundaria fue predicha usando el programa PSIPRED.
El gen ansA3 amplificado fue de aproximadamente 1000 pb y depositado en GenBank con el código 2601072.
Optimización de producción de L-asparaginasa. Se analizó el impacto de las concentraciones de: glucosa (1, 2, 3, 4 y 5 g/L), asparagina (5, 10, 15, 20 y 25 g/L), NaCl (10, 20, 30, 40 y 50 g/L) y el efecto del pH (5, 6, 7, 8 y 9). Para ello, se utilizó un inóculo de 2% (v/v) en medio M9 modificado, el cual tuvo una DO:1.0 . Para el diseño experimental se mantuvo un medio salino: Na2HPO4 6 g/L, KH2PO4 2 g/L, MgSO4.7H2O 0.5 g/L y CaCl2.2H2O 0.15 g/L; y las condiciones de cultivo fueron a 37 °C, 180 RPM por 24 h.
Análisis estadístico. Las corridas experimentales fueron realizadas por triplicado, analizados estadísticamente con GraphPad Prism 8.0.1; los gráficos se construyeron con la representación de la media y la desviación estándar.
Resultados y discusión
Producción enzimática. En los ambientes salinos habitan diversos microorganismos caracterizados por la producción de metabolitos que sirven para equilibrar su presión osmótica ante las condiciones salinas extremas y su efecto desnaturalizante (Ebrahiminezhad et al. 2011). En estos ambientes como el Mar Rojo, se ha podido aislar bacterias que logran producir L-asparaginasa entre 1.56 y 8.10 U/mL (Alrumman et al. 2019). En Perú, bacterias con tolerancia hasta de 20% de salinidad y productoras de L-asparaginasa han sido encontradas en las salinas de Maras (Cusco), Pilluana (departamento de San Martín) y Chilca (Lima) (Montes et al. 2021). La cepa Bacillus sp. M62 aislada de la salina de Maras demostró tener actividad L-asparaginasa, como ha sido reportado para otros Bacillus como B. licheniformis, B. alvei, B. subtilis, B. circulans, B. megaterium, B. cereus, adaptados a sobrevivir en condiciones de altas salinidad y producir elevados niveles de L-asparaginasa (Shirazian et al. 2016; Patil & Jadhav 2017).
Algunas bacterias requieren altas condiciones de salinidad, como Bacillus aryabhattai A3, aislado del lago Urmia, que produjo asparaginasa en medio M9 con NaCl 10% (Moghadam & Khadivi 2019). Así, estudios de la microbiota de sedimentos del golfo pérsico han reportado que las bacterias halotolerantes en ambientes salinos y productores de L-asparaginasa pertenecen a los géneros Bacillus, Pseudomonas, Zobellella, Oceanimonas, entre otros (Izadpanah et al. 2014). Por otra parte, Lactobacillus salivarius, aislado del mar de la India, demostró producir L-asparaginasa mediante fermentación sumergida, obteniendo máximos valores (11.02 µmol amonio/mL) a los cinco días de cultivo (Bhargavi & Jayamadhuri 2016). En este trabajo la actividad L-asparaginasa evidenciada mediante el viraje del indicador en el medio M9 se presentó después de 72 h de fermentación, lo que concuerda con lo reportado en otras bacterias endofíticas (War Nongkhlaw & Joshi 2015).
Especies de Bacillus han demostrado que pueden producir L-asparaginasa ya sea a nivel intracelular o extracelular; sin embargo, la producción extracelular presenta ventajas por su facilidad de purificación y escalamiento, además de menor presencia de proteasas (Pradhan et al. 2013). De suelos de Arabia se identificaron Bacillus spp. y Staphylococcus spp. con alta producción de L-asparaginasas extracelulares en fermentación sumergida (El-Dein et al. 2019). En el presente estudio, Bacillus sp. M62 produjo 0.37 U/mL (0.24 U/mg) de L-asparaginasa extracelular a las 96 h de fermentación y 0.22 U/mL (0.03 U/mg) a nivel intracelular a las 48 h con agitación a 120 RPM (Fig. 1).
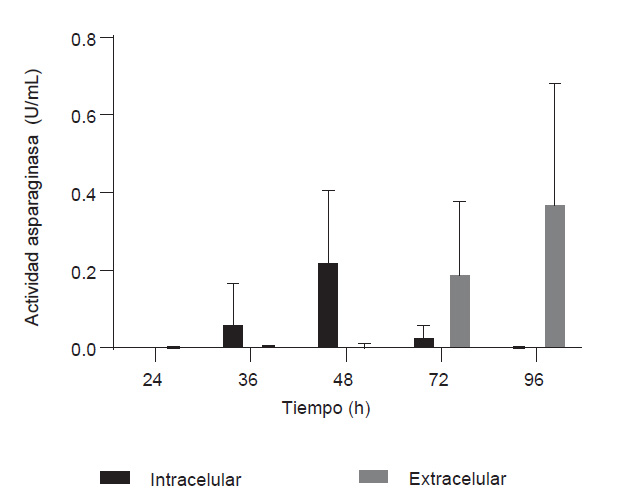
Figura 1 Actividad L-asparaginasa intracelular y extracelular de Bacillus sp. M62 mediante método Nessler en relación con el tiempo de fermentación.
De forma similar, se ha reportado que B. licheniformis MG665995 produjo actividades que superan en 305% a la intracelular, además, esta cepa produce L-asparaginasas de los tipos I y II a nivel intracelular y extracelular respectivamente, el tipo II posee alta afinidad por el sustrato a comparación de la intracelular (Abdelrazek et al. 2019). Además, Staphylococcus sp. MGM1 aislado de suelos; Pseudomonas aeruginosa 50071 y Acinetobacter baumannii S4 obtenidos de muestras clínicas producen L-asparaginasas extracelulares (El-Bessoumy et al. 2004; Muslim et al. 2016; Ebrahimipour et al. 2020).
Identificación molecular y análisis bioinformático. El ensamblado de las secuencias de los genes ribosómicos 16S se realizó a fin de obtener la secuencia consenso, que mediante el algoritmo BLASTN presentó 99.86% de similitud (1413/1415) con Bacillus licheniformis. El gen ansA3 amplificado fue de aproximadamente 1000 pb (Fig. 2), depositado en GenBank con el código 2601072, codifica para una proteína de 326 aa, la cual presentó 99.69% de similitud con otras L-asparaginasas del género Bacillus.
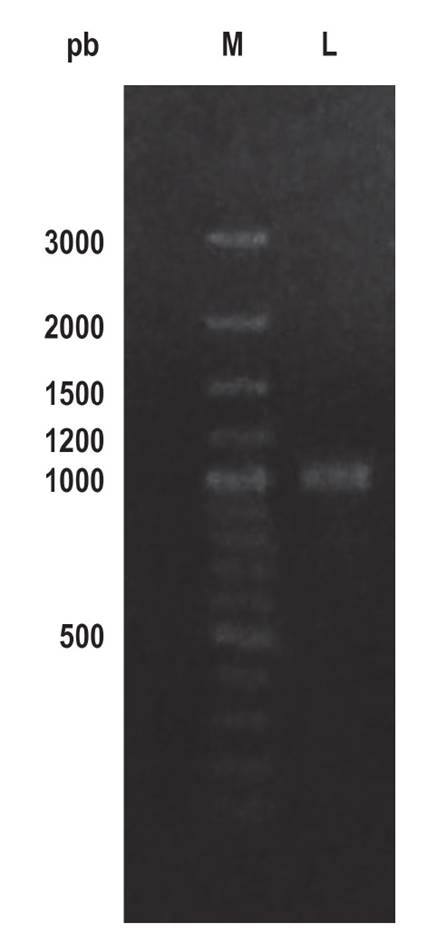
Figura 2 Gen ansA3 amplificado (L) de Bacillus sp. M62 y marcador de peso molecular Ladder 100 pb (M) en gel de agarosa 1%.
El análisis in silico indica que el gen ansA3 codifica una proteína de 35.4 kDa, la cual posee un sitio activo de O-isoaspartil treonina intermedio. Además, se identificó el dominio de 319 aa, así como el centro activo descrito con el patrón PS00917, GFVITHGTDTM, localizado entre los residuos 75 y 85, así como los aminoácidos treonina y ácido aspártico aledaños que se unen al sustrato (Material suplementario 1). La estructura secundaria (Fig. 3) está conformada por alfa hélices (29.75%, 97 aa) y hojas beta (18.40%, 60 aa). El predictor de topología identificó 286 aa pertenecientes a la zona extracelular, 16 residuos de hélice transmembrana, formando el revestimiento del poro y 24 en la región intracelular. Asimismo, se identificaron 15 sitios inmunogénicos (Material suplementario 2), similar a la enzima de Erwinnia chrysanthemi DB08886 y menor número a los identificados para la de E. coli DB00023 (que tiene 18 sitios), por lo cual su aplicación terapéutica podría presentar menor grado de inmunogenicidad y mejores valores de biodisponibilidad plasmática.
Se consideró el mejor modelo evolutivo al de Le y Gascuel (2008) con distribución discreta gamma. El análisis filogenético identificó que la enzima de Bacillus sp. M62 presenta una alta similitud de secuencia aminoacídica con L-asparaginasa de B. licheniformis ATCC 14580 (Fig. 4).

Figura 4 Árbol filogenético con base a los genes ansA3 de Bacillus sp. M62 y otras especies según soporte Bootstrap.
Optimización de la producción de L-asparaginasa. La productividad de L-asparaginasa está relacionada a los factores físicos y químicos que afectan su cultivo, como el pH. En nuestro trabajo, Bacillus sp. M62 produjo 0.91 U/mL de L-asparaginasa a pH 8.0 (Fig. 5). En otros casos, B. subtilis VUVD001 mejoró su producción hasta 2.88 U/mL (3.2 veces) a pH 7.56 (Erva et al. 2018), y B. licheniformis PPD37 halotolerante incrementó seis veces su producción a pH 7.5 en medio M9 (Patel et al. 2021).
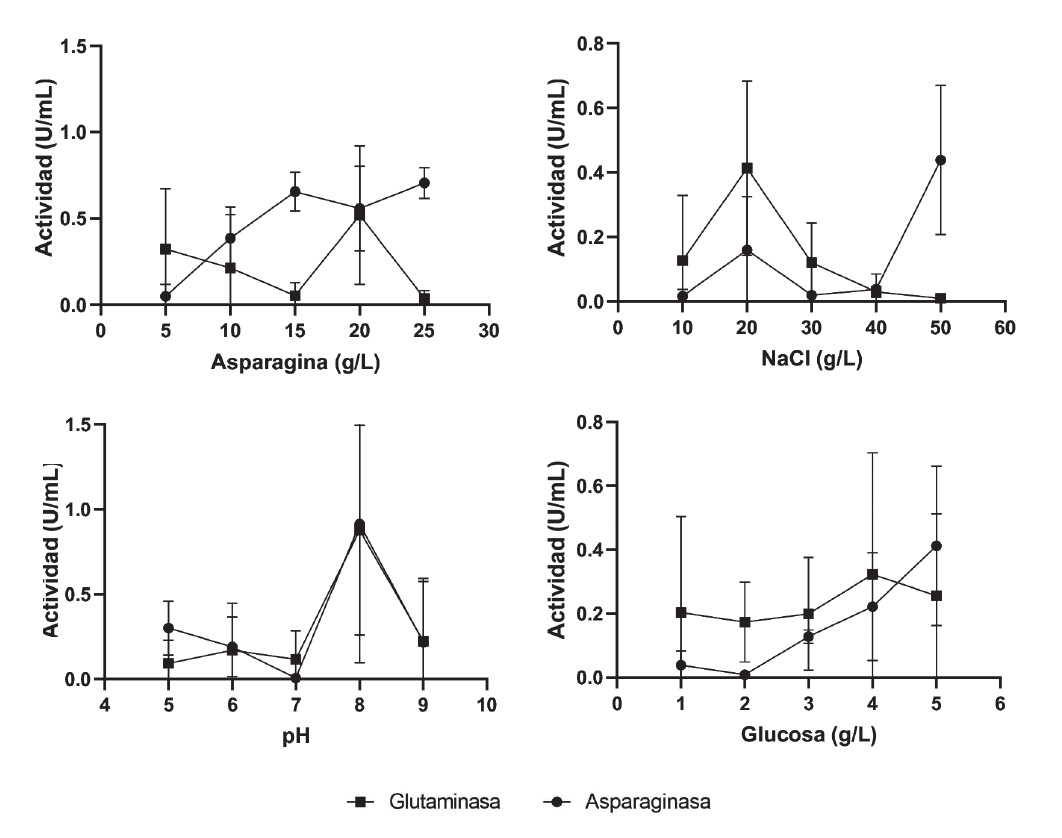
Figura 5 Efectos de L-asparagina, glucosa, NaCl y pH sobre la producción de L- asparaginasa y L-glutaminasa por Bacillus sp. M62.
La mejor producción a niveles alcalinos se explicaría por que el aspartato liberado a partir de L-asparagina posee baja afinidad por el sitio catalítico, mientras que a valores ácidos genera ácido aspártico e impide la unión de la L-asparaginasa a L-asparagina (Abdelrazek et al. 2019). Así, valores de pH 7.0 permitieron la producción óptima de L-asparaginasa en B. megaterium MG1 (Pal et al. 2019). De forma similar, Serratia marcescens NCIM 2919 mejoró su producción a pH 7.0, y bacterias aisladas de suelos de Solapur (India) produjeron hasta 103.70 U/mL optimizando su fermentación a pH 7.0, luego del cual se evidenció decrecimiento y mínima productividad a partir de pH 10 (Ghosh et al. 2013; Shaikh Khaleed & Khobragade 2016). No obstante, B. licheniformis KKU-KH14 y B. velezensis KB-9, aislados del mar Rojo en Arabia Saudita, revelaron máxima producción a pH 6.5, luego del cual disminuyó considerablemente, en tanto B. australimaris NJB19 produjo 37.93 U/mL a pH 6.77 (Alrumman et al. 2019; Mostafa et al. 2019; Chakravarty et al. 2021). Por el contrario, Shakambari et al. (2020) y Venil et al. (2009), demostraron que el pH no es un factor significativo para la producción en Pseudomonas plecoglossicida RS1 y Serratia marcescens SB08.
Otro factor significativo es el nivel de salinidad, así, se ha reportado que B. licheniformis PPD37 produjo hasta 17 U/mL con una concentración de NaCl de 0.8 g/L (Patel et al. 2021). No obstante, con un aumento a 29.20 g/L se incrementa la actividad en 48%, debido a la flexibilidad estructural de L-asparaginasa estimulada por la salinidad (Abdelrazek et al. 2019). También, Bacillus aryabhattai B8W22, obtenido del lago salado de Urmia, produjo 61.70 U/mL con NaCl 30 g/L (Shirazian et al. 2016). De igual forma, Gilavand et al. (2019) reportaron mejores niveles de producción a NaCl 30 g/L usando Rouxiella AF1. Sin embargo, en el presente estudio se obtuvo 0.44 U/mL con mínima actividad L-glutaminasa a NaCl 50 g/L. Por el contrario, Arthrospira platensis NIES-39 mejoró su producción de 0.17 a 0.28 U/mg con 10 g/L (Prihanto & Wakayama 2014). A la vez, Mahajan et al. (2012) y Shakambari et al. (2020), determinaron que este factor no es significativo en la producción de L-asparaginasa de B. licheniformis RAM-8, y Pseudomonas plecoglossicida RS1.
La L-asparagina fue la única fuente de nitrógeno usada para la producción de L-asparaginasa por Bacillus sp. M62, debido a la mayor actividad L-asparaginasa obtenida en comparación con L-arginina, NaNO3 y (NH4)2SO4 en otras cepas de Bacillus (Rahimzadeh et al. 2016). Su efecto como inductor de L-asparaginasa se verificó en P. aeruginosa WCHPA075019 con la obtención de 170.70 U/mg, superior a lo obtenido con extracto de levadura, peptona, glutamina y arginina (Amany et al. 2021). También, Jetti et al. (2017), reportaron como significativo el uso de L-asparagina en Pectibacterium carotovorum MTCC 1428, por la cual se obtuvo hasta 26 U/mL. En el presente estudio se alcanzó la mejor producción (0.71 U/mL) con L-asparagina 25 g/L, menor a lo requerido por B. licheniformis RAM-8, que utilizó L-asparagina 40 g/L para producir 32.26 U/mL (Mahajan et al. 2012). Así mismo, Bacillus altitudinis BITHSP010 aumentó su producción usando L-asparagina 20 g/L, luego del cual se observó disminución en su actividad (Prakash et al. 2020). No obstante, Lactobacillus sp. produjo 131 U/mg usando L-asparagina 17.5 g/L, suplementado con extracto de levadura 12.5 g/L (Dinarvand et al. 2020). De igual forma, Pseudonocardia endophytica VUK-10 registró 7.42 U/mL con L-asparagina 10 g/L y B. australimaris NJB19 37.93 U/mL con 24.4 g/L de este aminoácido, mientras que Lysinobacterium fusiformis B27 produjo 9.47 U/mL con 20 g/L (Kiranmayi et al. 2014; Prihanto et al. 2019; Chakravarty et al. 2021). También, Golbabaie et al. (2020), predijeron utilizando un diseño compuesto central hasta 1.73 U/mL usando L-asparagina 10 g/L en Sarocladium strictum AG90, sin considerarlo significativo, además, Bacillus sp. GH5 incrementó su producción de 9.57 a 45.37 U/mL usando L-asparagina 5 g/L y gelatina 5 g/L como fuentes de nitrógeno (Gholamian et al. 2013).
La glucosa es la fuente de carbono que induce mayor actividad de L-asparaginasa en comparación con maltosa en Bacillus DKMBT10 y sacarosa en coliformes, con máximos valores a las 24 h de fermentación (Moorthy et al. 2010; Shah et al. 2010). Así, la mejor producción (0.41 U/mL) fue obtenida con 5 g/L, ya que mayores concentraciones generan fermentación incompleta al no ser metabolizados todos los azúcares. Su efecto represor se basa en la disminución de AMPc y la inhibición de catabolitos de los componentes de transporte de lactato ligados a la síntesis de L-asparaginasa (Garaev & Golub 1977). En consecuencia, cepas coliformes obtuvieron mejor producción (650 U/mL) con 10 g/L y valores superiores evidenciaron su efecto represor (Shah et al. 2010). Zymomonas mobilis ATCC 35001 registró mejor producción con una relación 1:0.025 para sacarosa y extracto de levadura como fuentes de carbono y nitrógeno, respectivamente, donde la actividad tuvo una relación directamente proporcional a la biomasa producida (Menegat et al. 2016). Así mismo, Pseudomonas aeruginosa SN004 produjo L-asparaginasa 785 U/mL, con glucosa 2 g/L mientras que P. fluorescens NCIM 2100 obtuvo 28.71 U/mL con glucosa 1.5 g/L (Jha et al. 2014; Badoei-Dalfard 2015).
La optimización de la producción de L-asparaginasa ha conllevado la búsqueda de diferentes tipos de métodos como la obtenida por Prihanto et al. (2014), mediante exposición de la cepa a radiación UV por 120 min, de esta forma mejoraron la producción de L-asparaginasa de 3.18 a 8.02 U/mL en B. subtilis RM-1. No obstante, Bacillus sp. M62 demostró mejorar la producción a 2.15 ± 0.39 U/mL (0.63 U/mg) de L-asparaginasa extracelular usando los niveles optimizados de cada uno de los factores, además de evidenciar mínima actividad glutaminasa (0.02 U/mL). Por lo cual, se propone mayores estudios como el clonaje y expresión del gen ansA3 extracelular para su purificación y escalamiento como alternativa promisoria al tratamiento de pacientes con leucemia.












 uBio
uBio