Introducción
En la industria vitivinícola se recurre frecuentemente a la inoculación de levaduras comerciales en la fase de la fermentación alcohólica, aunque no garantizan ser las únicas en el proceso, inhiben el crecimiento de bacterias contaminantes que podrían afectarlo (Capece et al. 2019; Ortiz Navarro 2015). Por lo anterior, se viene empleando de manera más eficiente cultivos puros aislados de zonas vitivinícolas, lo que permite la implementación de levaduras nativas seleccionadas, otorgando propiedades únicas a las bebidas alcohólicas y una mayor eficiencia en la fermentación alcohólica (Querol et al. 2018). La utilización de levaduras nativas seleccionadas en procesos de vinificación presenta ventajas frente a la fermentación espontánea tradicional (Tristezza et al. 2016), por ejemplo, tienen una mayor velocidad de fermentación y reducen el riesgo de contaminación por levaduras salvajes. Por lo tanto, la selección de levaduras propias de cada región permite conservar la tipicidad de los vinos elaborados en ella, asegurando la implantación de estas en los mostos y preservando los recursos genéticos propios de cada región vitivinícola (Combina et al. 2005; Capece et al. 2019; García et al. 2019).
A nivel molecular, la técnica de los Fragmentos de Restricción del ADN Mitocondrial (ADNm-RFLP) (Querol et al. 1992, Barragán-Castillo 2020) ha permitido diferenciar cepas de levaduras con propiedades fisiológicas similares a S. cerevisiae. Así también, se ha empleado en estudios básicos de diversidad y también en el control de calidad de cepas comerciales de levaduras y en el control de fermentaciones vínicas inoculadas (Fernández-Espinar et al. 2001; Capece et al. 2019). Por otro lado, la técnica de Fragmentos de Restricción de Longitud Polimórfica (RFLP) de las regiones ribosomales D1/D2 del gen 26S, es la más utilizada actualmente, como un método rápido y confiable para la identificación de levaduras. (Fernández-Espinar et al. 2006; Capece et al. 2012).
El pisco es un producto de gran demanda y crecimiento en el mercado nacional e internacional y que emplea una fermentación espontánea o natural en su proceso productivo. El departamento de Ica, caracterizado por su clima cálido, sus bodegas de piscos y vinos y sus plantaciones de uva que cubren una superficie de 7662 ha, de las cuales 3164 ha son utilizadas para la elaboración de piscos y vinos (ADEX 2022). La producción pisquera de Ica constituye el 60%-65% de la producción nacional de pisco y además constituye una actividad vitivinícola tradicional (Cáceres & Julca 2018). Quebranta es la variedad de vid originaria de Perú, producto de la hibridación natural entre las variedades Listan Prieto y Mollar Cano (This et al. 2006; Almanza et al. 2021). Esta variedad es la más utilizada en la elaboración de Pisco en la región de Ica y en las demás zonas de denominación de origen, por ello es considerada la variedad de vid emblemática de Perú.
El presente trabajo tiene como objetivo la identificación y la selección de levaduras nativas para la producción de pisco a partir de la uva Quebranta, con la finalidad de obtener una producción homogénea de pisco, con características sensoriales distintivas, genuinas y que conserven la tipicidad de los piscos elaborados en la región. Se aislaron cuatro cepas y se seleccionó a la levadura UNA SC-49, la cual destacó por mostrar aptitudes enológicas de mayor rendimiento alcohólico, siendo además el mejor cultivo iniciador de fermentación de mostos de uva Quebranta y del cual se obtuvo el pisco con características sensoriales relevantes.
Material y métodos
Muestra. Durante la campaña 2018, el 7 de marzo se colectaron mostos frescos de uva Quebranta provenientes de los tanques de fermentación de Agroindustrias Tres Generaciones SAC [14°00'33.5"S, 75°45'17.4"W], ubicado en la zona alta del valle (Ica, Perú). La muestra colectada fue de 5 litros, escala representativa en laboratorio a nivel industrial, la cual fue transportada en condiciones de refrigeración (4 °C) para su procesamiento en el laboratorio del Instituto de Biotecnología de la Universidad Nacional Agraria la Molina.
Aislamiento de levaduras. Se recolecto 500 mL del mosto en un Erlenmeyer de un 1L (con 3 repeticiones) fueron fermentados espontáneamente por espacio de 12 días; las levaduras fueron aisladas a partir del día de haber alcanzado tres cuartos de la fermentación alcohólica (FA) hasta el final de la FA (1.011-1.023 g. cm-3; ≤0.990, respectivamente), la cual se realizó entre los días 7 y 10 de la fermentación. Se tomó un inóculo del mosto (1 mL) y se realizó una dilución seriada (10-1 - 10-3) 0.1 mL de las muestras diluidas fueron sembradas en medio agar Yeast Extract-Peptone-Dextrose ‒YPD (Extracto de levadura 10 g·L-1, peptona 20 g·L-1, glucosa 20 g·L-1 y Agar 15 g·L-1) suplementado con cloranfenicol (0.5 g·L-1) e incubado a 28 °C por 48 h para el aislamiento de 30 colonias por día, desde el séptimo hasta el décimo día de fermentación.
Identificación morfológica y fisiológica. Las colonias aisladas fueron replicadas en agar Wallestein Laboratory - WL (extracto de levadura 4 g·L-1, peptona pancreat caseína 5 g·L-1, glucosa 50 g·L-1, sulfato monopotásico 550 g·L-1, cloruro potásico 425 g·L-1, cloruro cálcico 125 g·L-1, sulfato magnésico 125 g·L-1, cloruro férrico 2.5 g·L-1, sulfato de manganeso 2.5 g·L-1, verde de bromocresol 22 g·L-1 y agar-agar 15 g·L-1) para seleccionar presuntivamente aquellas asociadas con Saccharomyces spp., por sus características morfológicas en placa (colonias elevadas, con borde liso y su coloración blanca o verde pálido en el medio WL) (Pallmann et al. 2001). Además las levaduras elegidas fueron sembradas posteriormente en medio Agar Lisina (6,6 g de medio Lisina (OXOID LTD)/100 mL agua destilada; 1 mL Lactato de potasio al 50%/100 mL; 0.1 mL ácido láctico (Sigma-Aldrich) al 10%/ 100mL y agar 20 g·L-1), donde sólo tenían lisina como única fuente de nitrógeno. Las levaduras del género Saccharomyces no pueden crecer en este medio, debido a que forman un inhibidor intermediario. Por el contrario, la mayoría de las levaduras no-Saccharomyces sí pueden utilizar este aminoácido como única fuente de nitrógeno (Fugelsang & Edwards 2007), por lo que, al no haber desarrollo de los aislados en este medio se confirmó que se trataría de levaduras del género Saccharomyces.
Extracción de DNA. La extracción de DNA se realizó según Hoffman et al. (1987) con algunas modificaciones, brevemente las colonias fueron cultivadas en caldo YPD a 28 °C por 48 h en agitación, las células fueron precipitadas a 3500 RPM por 5 min y transferido a un microtubo de 2 mL, el pellet fue lavado con agua libre de nucleasas para eliminar restos del medio (3500 RPM por 5 min), luego se resuspendió en 200 µL buffer de lisis (1% SDS, 2% Triton X-100 , 100 mM NaCl, 10 mM; pH 8 Tris-HCl, 0.1 mM EDTA) posteriormente se adicionó 200 µl de fenol: cloroformo: isoamílico (25:24:1) para luego proceder a la ruptura de las células a través de un vortex por 5 min. Se adicionó 200 µL de buffer TE (10 mM Tris-HCl, pH 8; EDTA 0.1 mM) y se centrifugó a 13000 RPM por 5 min. El sobrenadante fue colectado en un microtubo de 1.5 mL donde se adicionó etanol puro frío (- 20 °C) mezclados por inversión hasta la precipitación de los ácidos nucleicos, centrifugándose a 13000 RPM por 5 min. El sobrenadante fue descartado y el pellet fue resuspendido en buffer TE. Se realizó un tratamiento enzimático con RNAsa A (10 mg. mL-1) a 37 °C por 2 horas. Los DNAs extraídos tuvieron un tratamiento con acetato de potasio (10 M) y lavado con etanol puro frío los cuales fueron mezclados por inversión. El pellet de DNA fue lavado dos veces en etanol frío (70%) y posteriormente resuspendido en buffer TE.
La integridad del DNA fue realizado a través de una electroforesis de agarosa en buffer TBE a 90 V por 45 min, la tinción se realizó bromuro de etidio (1 mg·mL-1) y la visualización de los DNA se realizó con un fotodocumentador Biometra®.
Identificación molecular de Saccharomyces cerevisiae. La caracterización de la especie se realizó mediante la amplificación del gen ribosomal 5.8S y las regiones intergénicas ITS1 e ITS2. Se emplearon los primers ITS1 (5′-TCCGTAGGTGAACCTGCGG-3′) e ITS4 (5′-TCCTCCGCTTATTGATATGC-3′) que fueron previamente descritos por White et al. (1990) y modificado por Innis et al. (2012). La reacción de amplificación se llevó a cabo en un termociclador PeqStar bajo las siguientes condiciones 95 °C x 5 min, 30 ciclos de 94 °C x 1 min, 55.5 °C x 2 min, 72°C x 2 min y una fase final de 72°C por 10 min. El tamaño de los fragmentos amplificados fue estimado comparando con un marcador de peso molecular de 100 pb (New England BioLabs Inc.) a través de una electroforesis en gel de agarosa.
El análisis del polimorfismo de la longitud de los fragmentos de restricción (RFLP) de la región 5.8S ITS se realizó mediante la digestión enzimática con Hae III, Hpa II y Scrf I (New England BioLabs Inc.). Los patrones de bandas generados por la restricción enzimática permitieron identificar especies dentro de Saccharomyces sensu stricto (Fernández-Espinar et al. 2001, Lopes et al. 2002). La reacción se llevó a cabo en 20 µL de volumen total conteniendo 2 µL del buffer de digestión, 0.5 µL de la enzima de restricción, 2.5 µL de agua libre de nucleasas y 15 µL del producto amplificado. La digestión enzimática se llevó a cabo a 37 °C por 3 horas. La electroforesis se realizó en un gel de agarosa (3%) a 120V por 1hora y los tamaños de los fragmentos obtenidos fueron estimados mediante un marcador de peso molecular de 100 pb. La identificación de la especie se realizó comparando los fragmentos de restricción amplificados según lo reportado por Guillamón (1994, 1998).
Identificación intraespecífica de las cepas de Saccharomyces cerevisiae . Se amplificó la región interdelta y se secuenció parcialmente el dominio D1/D2 del gen ribosomal 26S, para la identificación de levaduras con los primers específicos NL1 y NL4 según lo reportado por Valente et al. (1999). Los análisis de secuencias se realizaron mediante el programa SeqMan (DNAstar), las secuencias consenso se compararon en BLAST del NCBI (National Center for biotechnology Information NCBI (http://blast.ncbi.nlm.nih.gov/Blast.cgi).
Caracterización enológica
Evaluación de la aptitud fermentativa de las cepas nativas seleccionadas. Las cepas aisladas fueron cultivadas para la producción de biomasa (1 x 106 UFC·mL-1) por triplicado según Massera (2010). A nivel de laboratorio, se desarrolló la caracterización enológica en 300 mL de mosto de uva Quebranta a 20 °C (previamente esterilizados). Se evaluó la velocidad de fermentación, capacidad de inicio y culminación de la fermentación con elevado contenido de azúcares del mosto (262.4 g·L-1).
Inoculación de las cepas Saccharomyces cerevisiae aisladas en uva Quebranta para la elaboración del pisco. Se realizó una microvinificación en 5 L de mosto de uva Quebranta previamente esterilizadas con metabisulfito de potasio (50 mg·L-1) para eliminar la flora microbiana natural de las uvas. El proceso se llevó a cabo en tanques de fermentación a escala piloto de acero inoxidable a 20 °C donde se adicionaron las cepas aisladas de S. cerevisiae (2 x 106 UFC mL-1). Una cepa comercial de S. cerevisiae (Zymasil-Saccharomyces cerevisiae) fue considerada como control. Las fermentaciones fueron monitoreadas diariamente a través de su densidad (g·cm-3) hasta el agotamiento del azúcar. Así también se realizó los análisis fisicoquímicos (grado alcohólico, acidez volátil, ésteres, azúcares residuales y glicerol), culminado el proceso de microvinificación se procedió a la microdestilación según la Norma Técnica Peruana para la elaboración del Pisco (INDECOPI 2006). El pisco obtenido fue evaluado según sus caracteres fisicoquímicos, así también sus perfiles organolépticos y sensoriales, los cuales fueron realizados en el Laboratorio para la Certificación del Pisco del Centro de Innovación Productiva y Transferencia Tecnológica Industrial de Ica (CITEagroindustrial). Las pruebas fisicoquímicas fueron realizadas mediante el método de ensayo de la NTP 210.003.2003 para la evaluación de grado alcohólico volumétrico, la NTP 211.041.2003 para la evaluación del extracto seco a 100 °C y la NTP 211.035.2003 para la evaluación de acidez volátil, como ácido acético, esteres, como acetato de etilo, aldehídos, como acetaldehído, furfural, alcoholes superiores, como alcoholes superiores totales y alcohol metílico respectivamente. Posteriormente, se realizó el análisis organoléptico y sensorial con un panel de catadores expertos de la Comisión Nacional del Perú (CONAPISCO 2016) reconocidos por la Organización Intergubernamental de la Vid y el Vino (OIV 2022). Las cepas de levadura aisladas fueron almacenadas en glicerol (40%) a -80 °C.
Análisis estadístico. Los análisis fisicoquímicos de calidad del vino y pisco fueron evaluados mediante un análisis de varianza, la comparación de las medias se realizó a través de una prueba de Tukey (pvalue ≤ 0.05). Los datos fueron analizados con el software R v.4.2.2 (R Core Team. 2023) y RStudio v.7.2.576 576 (Posit team. 2023) con paquetes tales como: ggplot2 (Wickham H. 2016) y fmsb (Nakazawa M. 2023) para realizar estadísticas descriptivas con graficas de líneas en el tiempo y radar. En el análisis multivariado se utilizó un análisis de componentes principales (PCA) utilizando los paquetes de software R: factoextra (Kassambara et al. 2020) y FactoMiner (Le et al. 2008) para discriminar las cepas de levaduras y la relación de las variables en estudio.
Resultados y discusión
Aislamiento de levaduras. Se aislaron 120 cepas de levaduras a partir de mosto Quebranta, con características morfológicas típicas del género Saccharomyces.
Caracterización genética. Utilizando la metodología de Hoffman et al. (1987), se extrajo el ADN de 120 aislados de las cuales 4 cepas fueron identificadas como Saccharomyces sensu stricto, a quienes se sometió a pruebas de fermentación a nivel de laboratorio para validar su aplicación en la producción de pisco. Estos 4 aislados se determinaron como pertenecientes al grupo Saccharomyces sensu stricto luego del análisis de ITS1-5.8S-ITS2 PCR-RFLP. El producto amplificado mostró un tamaño de 850 pb, similar a lo reportado por el grupo de Fernández-Espinar et al. (2001, 2006) (Figura 1).
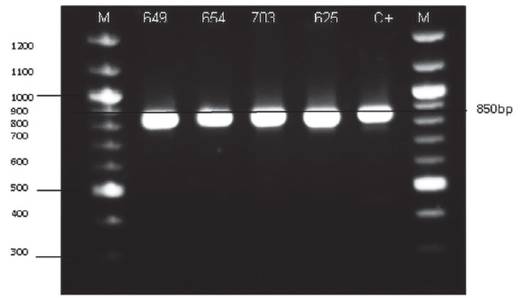
Figura 1 Amplificación del gen ITS1-5.8S-ITS2 de las cepas identificadas como Saccharomyces sp. Electroforesis en agarosa (1.4 %). M= marcador 100pb. C= cepa comercial de Saccharomyces sp.
Identificación de especie de Saccharomyces cerevisiae. El análisis polimórfico de los fragmentos de restricción obtenidos como resultado de la digestión de productos amplificados con las endonucleasas (Scrf I, Hae III, Hpa II, Hinf I) mostró similitud en los patrones de restricción en los 4 aislados (Figura 2 y Tabla 1). Los fragmentos de digestión obtenidos no muestran coincidencias con las cepas registradas en la base de datos presentada (Tabla 1) para identificación a nivel de especie. Esto sugiere que las cepas identificadas pertenecen a un grupo distinto y propio de la región de Ica, por ello la incompatibilidad con trabajos semejantes en otras zonas geográficas.
Tabla 1 Pesos de bandas producto de la digestión con endonucleasas.
| Amplificado | Enzimas de restricción | |||
|---|---|---|---|---|
| Scrf I | Hae III | Hpa II | Hinf I | |
| 850 pb | 400 | 340 | 800 | 370 |
| 310 | 280 | 150 | ||
| 140 | 230 | |||
Identificación intraespecífica de las cepas de Saccharomyces cerevisiae. Los cuatro aislamientos (UNA SC-25, UNA SC-49, UNA SC-54, y UNA SC-03) amplificaron el dominio D1/D2 del gen ribosomal 26S, presentó un producto entre 600-700 pb, el cual corresponde al patrón reportado para especies de Saccharomyces cerevisiae (Valente et al. 1999) (Figura 3). Luego del análisis de ensamblaje de secuencias con el programa SeqMan (DNAstar) se obtuvo una secuencia consenso de 180 nucleótidos donde la secuencia consenso (TTTGTAAAGTGCCTTCGAAGAGTCGAGTTGTTTGGGAATGCAGCTCTAAGTGGGTGGTAAATTCCATCTAAAGCTAAATATTGGCGAGAGACCGATAGCGAACAAGTACAGTGATGGAAAGATGAAAAGAACTTTGAAAAGAGAGTGAAAAAGTACGTGAAATTGTTGAAAGGGAAGGGCA). El alineamiento a través del BLAST permitió confirmar que la secuencia consenso pertenece a la secuencia parcial del gen del RNA ribosomal 26S de Sacharomyces cerevisiae.
De acuerdo con Belloch et al. (1998), la técnica de RFLP ha sido considerada una de las más empleadas para la identificación y filogenia de levaduras, a partir de la región ITS (regiones no codificadas e hipervariables) han permitido el reconocimiento a nivel interespecífico, así por ejemplo para la identificación de especies del género Kluyveromyces, pero contrariamente no permite a nivel específico para el género Saccharomyces. Sin embargo, Segura et al. (2010) indica que si bien la técnica de RFLP de las regiones ITS-5.8S es factible como una prueba de identificación rápida y fácil de interpretar, muestra desventaja al realizar el análisis y no encontrar un patrón de bandas igual al que se obtuvo, ya que aún no se han reportado todos los patrones de bandas para todas las especies, como ocurre en el presente trabajo.
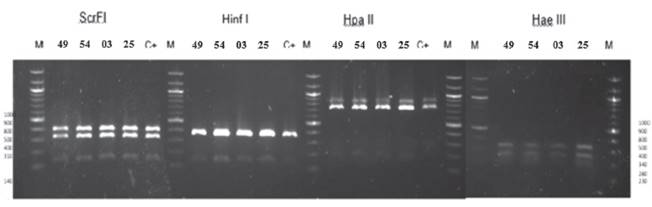
Figura 2 Identificación de levaduras aisladas al final de la fermentación espontanea por PCR-RFLP de la región ITS1-5.8S-ITS2. Patrones de restricción con Scrf I, Hinf I, Hpa II, Hae III. Los números a los extremos de la figura se refieren al tamaño de fragmentos de ADN en pares de bases y se usó el marcador de peso moléculas de “100 pb DNA ladder” (M). Los números en la parte superior indican la codificación de los aislados y la cepa control Saccharomyces comercial (C+).
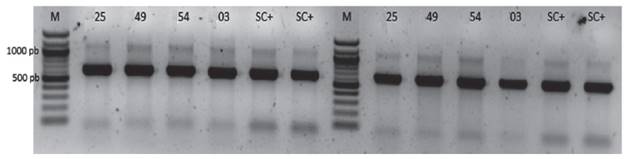
Figura 3 Amplificación del gen ribosomal 26S (Dominio D1/D2) con los primers NL1 y NL4 genera un amplicon de 600-700 pb. M: marcadores de peso de 100 pb, 25, 49, 54 y 03: Código de los aislados de levaduras. SC+: control positivo de Sacharomyces cerevisiae.
Por otro lado, empleando otras técnicas como cariotipo, amplificación de regiones δ o análisis de restricción del ADN mitocondrial las cuales han permitido identificar cepas autóctonas de S. cerevisiae representativas a diferentes ambientes geográficos (Vezinhet et al. 1992; Schvarczová et al. 2017; Castillo et al. 2020), confirmando que las cepas de levaduras nativas se encuentran adaptadas a determinadas característica climáticas y substratos. Así, algunos enólogos corroboraron que la aplicación de levaduras seleccionadas procedentes de la microzona donde fueron utilizadas dieron buenos resultados (Garofalo et al. 2018; Capece et al. 2019; García et al. 2019; Dumitrache et al. 2020), además señalaron que la diversidad, composición y evolución de la microbiota de levaduras presente en el mosto depende de varios factores como la localización geográfica y las condiciones climáticas (temperatura, lluvias, etc.) (Querol et al. 1990; Barata et al. 2012; Belda et al. 2016), esto explicaría la gran diversidad y la poca o ninguna coincidencia con la base de datos YeastID (https://www.yeast-id.org/) para la identificación de la subespecie en el presente estudio.
Caracterización enológica. Tres cepas de levaduras nativas (UNA SC-25, UNA SC-49 y UNA SC-54) presentaron buena adaptación al entorno y con un perfil organoléptico distintivo; este resultado fue contrastado con lo reportado por García et al. (2019) y Arroyo-López et al. (2010) quienes concluyen que la selección de levaduras locales asegura la conservación de las propiedades sensoriales de vinos producidos en una región determinada. Las pruebas enológicas comprobaron la superioridad organoléptica de al menos una cepa aquí identificada.
El seguimiento de la fermentación alcohólica (FA) tuvo una duración máxima de 12 días, para cada una de las cepas en monocultivo. De las 4 cepas analizadas, UNA SC-03 fue descartada en esta etapa debido a su bajo crecimiento en biomasa para la inoculación. Las fermentaciones UNA SC-25, UNA SC-49 y UNA SC-54 fueron realizadas en laboratorio simulando las condiciones de bodegas artesanales, con mosto de uva Quebranta (pasteurizado) a temperatura controlada (20 °C) durante un periodo de 8 a 12 días (Figura 4). En todos los mostos evaluados, las levaduras nativas fueron capaces de llevar a cabo la FA hasta del gasto total del azúcar, mostrando diferentes cinéticas. Estas diferencias podrían atribuirse a la variación genética de cada inóculo de levaduras nativas en el mosto y a la capacidad de estas para llevar a cabo la FA a 20 °C. La velocidad de la FA con la cepa UNA SC-54 fue más rápida en relación con las demás cepas, mientras que la cepa UNA-25 fue la más lenta. Comportamientos similares fueron reportados en la investigación de Yamamoto et al. (2011), quienes aislaron 7 cepas de levaduras nativas para la producción de Shochu, un destilado típico de Japón análogo al Sake, en la cual observaron también diferencias en la actividad fermentativa, seleccionando una cepa en particular que genéticamente era una S. cerevisiae, pero con características distintivas a las cepas industriales ya que tenía una alta actividad fermentativa, alto rendimiento de alcohol y aromas particulares, características buscadas para la diversificación de este tipo de destilados, al igual que el pisco para rescatar la tipicidad, objetivo de esta investigación.
Se determinaron las propiedades enológicas de cada cepa inoculada, permitiendo conocer y comparar la evolución de estos caracteres a partir de cada fermentación. Particularmente, la cepa UNA SC-49 presentó un alto rendimiento alcohólico al igual que la cepa control SC (S. cerevisiae comercial-Zymasil) en referencia a las cepas testadas, alrededor del 4% (Tabla 2). En Italia, Garofalo et al. (2018), reportaron resultados similares, encontrando que el 90% de las cepas nativas estudiadas tuvieron un alto rendimiento en alcohol (entre 10-12 v/v) comparado con la cepa control, lo cual también es observado en la cepa UNA SC-49 del presente trabajo. Por otro lado, la relación del grado alcohólico y el contenido de azúcar residual estuvieron de acuerdo con los resultados reportados por Martínez-Lapuente et al. (2016) y Vigentini et al. (2017). Es decir, la cepa UNA SC-49 fue capaz de concluir la fermentación hasta dejar trazas de azúcares (2.40 g·L) al igual que la cepa UNA SC-25 y la cepa control SC. Contrariamente, UNA SC-54 dejó azúcares residuales en mayor cantidad (4.84 g·L) (Tabla 2).
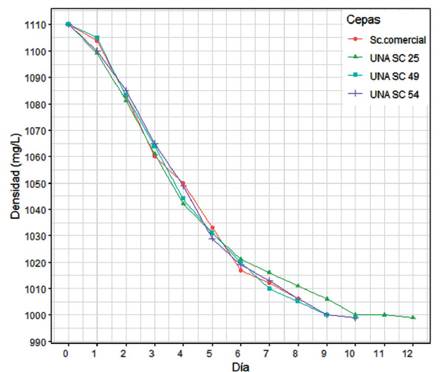
Figura 4 Proceso fermentativo y evaluación del consumo de azúcares (Densidad gr/L) de tres cepas nativas seleccionadas y una cepa control (Sc. Comercial) en mosto de uva Quebranta durante 12 días.
Tabla 2 Evaluación de los parámetros de calidad del mosto fermentado a partir de uva Quebranta con las 3 cepas nativas de levaduras. La evaluación se realizó con tres repeticiones. Los valores representan la media ± SD. Se comparó mediante un Análisis de Varianza (pvalue ≤0.05) así mismo también se comparó mediante la prueba de Tukey para evaluar las diferencias de producción entre las cepas aisladas y Sc. Comercial: control.
| Pisco | Grado alcoholico | Acidez volatil | Azucares residuales | Esteres | Glicerol |
|---|---|---|---|---|---|
| UNA_SC-49 | 14.3 ± 0.09b | 0.37 ± 0.01ab | 2.3 ± 0.005b | 2.68 ± 0.005a | 10.4 ± 0.01a |
| Sc.comercial | 15.02 ± 0.03a | 0.42 ± 0.01a | 2.4 ± 0.025b | 2.63 ± 0.015b | 8.31 ± 0.005b |
| UNA_SC-25 | 9.64 ± 0.05c | 0.33 ± 0.02b | 2.4 ± 0.015b | 0.44 ± 0.02c | 7.62 ± 0.01c |
| UNA_SC-54 | 12.38 ± 0.05d | 0.24 ± 0.01c | 4.85 ± 0.047a | 0.44 ± 0.005c | 7.39 ± 0.01d |
Por otro lado, los análisis químicos al final de la FA muestran una producción de acidez volátil expresada en ácido acético, dentro de los parámetros establecidos por la OIV, para todas las cepas evaluadas, la cepa UNA SC-49 tuvo una notable reducida producción (0.23 g·L) (Tabla 2), a pesar del elevado grado alcohólico creciente durante la fermentación, comparable a los resultados obtenidos por Vigentini et al. (2017), con un valor medio de 0.39 g/L de ácido acético. En ese sentido, la Comunidad Europea (CE) indicó el límite legal de acidez volátil, alrededor de 1.5 g/L (Bely et al. 2008), por lo que concentraciones superiores a 1.2 - 1.3 g/L podrían resultar desagradables y restar calidad al producto final. Asimismo, también en el estudio de Garofalo et al. (2018) la mayoría de las cepas nativas investigadas presentaron valores de acidez volátil inferiores al umbral de sensibilidad entre 0.6 y 0.9 g/L.
En los destilados como el pisco, el glicerol aporta suavidad y una sensación de aterciopelado en boca, atenuando las sensaciones “ardientes” debidas al alcohol y, eventualmente, a la excesiva aspereza por la tanicidad dependiendo de la variedad de vid. Otro aporte del glicerol es la dulzura y sensación de llenado en boca. Altos valores de glicerol (10 g/L) fueron registrados por la cepa UNA SC-49 (Tabla 2), carácter que dio un efecto favorable en la calidad del vino y por tanto en el pisco (Figura 5), impactando de manera positiva a nivel sensorial, con el alto grado alcohólico (38-48 °GL) de este destilado. Estos resultados apoyan diferentes investigaciones de levaduras nativas en bebidas fermentadas a partir de uva en la cual reportaron que generan grandes cantidades de glicerol y tienen un considerable impacto para mejorar la calidad de los vinos (Zhao et al. 2015; Mančić et al. 2021).
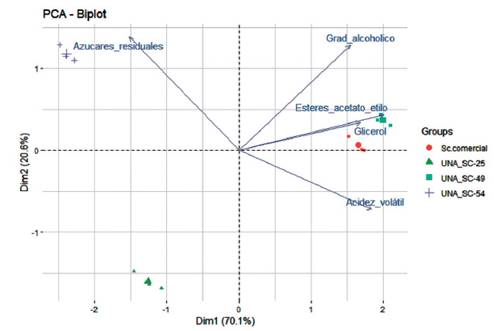
Figura 5: Análisis de componentes principales de los parámetros de calidad del vino antes de ser destilado (azúcar residual, grado alcohólico, esteres, gliceroles y ac. Volátiles) producido durante el proceso de fermentación de tres cepas nativas de levaduras seleccionadas para la producción de pisco en uva Quebranta y una cepa control (Sc. Comercial). UNA SC-49 presenta cualidades semejantes o superiores a la cepa empleada como como control.
Se observaron diferencias entre las cepas nativas evaluadas en cuanto a la producción de ésteres expresado como acetato de etilo, otro parámetro importante en el pisco. En particular la cepa UNA SC-49 presentó el más alto valor (2.69 g·L), seguido por la cepa control con (2.60 g·L), mientras que la cepa UNA SC-54 y UNA SC-25 ambas tuvieron igual y bajo rendimiento (0.44 g·L.) (Tabla 2). En efecto, la composición del etilo de los ácidos grasos demostró que era un carácter dependiente de la cepa como lo han sugerido Kemp et al. (2015), y que además pueden contribuir en el aroma de los vinos ya que los ésteres son el principal grupo químico de los compuestos volátiles. El contenido de ésteres, las concentraciones de alcoholes superiores y la disponibilidad de los precursores son los principales factores limitantes para la producción de ésteres (Capozzi et al. 2016). En general, luego de la validación de las aptitudes enológicas, la cepa UNA SC-49 es el mejor cultivo iniciador de fermentaciones de mostos de uva Quebranta para obtener el pisco de mejor calidad (Figura 5).
Pasado tres meses de reposo de los destilados, a partir de los vinos obtenidos con cada una de las cepas seleccionadas, de acuerdo con las indicaciones de la norma técnica (INDECOPI 2006), fueron confirmadas las aptitudes de la cepa UNA SC-49 como mono inóculo por sus óptimas características fisiológicas y fermentativas para la elaboración del pisco. Todos los piscos elaborados con las cepas nativas seleccionadas estuvieron dentro de los parámetros establecidos por la Norma Técnica Peruana para la elaboración del pisco (Tabla 3) (INDECOPI 2006) sin embargo en la evaluación sensorial se pudo evidenciar una diferenciación de las cualidades organolépticas de la cepa UNA SC-49, en todos los descriptores relacionados al pisco: limpidez, tipicidad, color, calidad 6-20 (gustativo), calidad 7-15 (olfativo), intensidad positiva, persistencia armoniosa y armonía juicio global (Figura 6), destacando frente a las demás cepas incluso al elaborado con la cepa control (S. cerevisiae comercial-Zymasil), desarrollando un perfil organoléptico positivo, reproducible y fácilmente reconocible. Contrariamente el pisco obtenido con la cepa UNA SC-25, tuvo una menor valoración para todos los descriptores excepto para calidad 6-20 (gustativo) (Figura 6).
Tabla 3 Determinación de variables de calidad del Pisco elaborado con 3 cepas nativas de levaduras. La evaluación se realizó con tres repeticiones. Los valores representan la media ± SD. Se comparó mediante un análisis de Varianza (pvalue ≤0.05) así mismo también se comparó mediante la prueba de Tukey para evaluar las diferencias de producción entre las cepas aisladas. Sc. Comercial: control.
| Pisco | GL | Ex_seco | Ac_Vol | Esteres | Aldehido | Furfural | Alcoh_Sup | Alcoh_Met |
|---|---|---|---|---|---|---|---|---|
| Sc. comercial | 42.8±0.15a | 0.2±0.004c | 40.033±1.39a | 40.5±0.84ab | 4.9±0.02b | 0.9c | 305.1±13.60a | 28.3±0.38a |
| UNA_SC-25 | 40.6±0.15b | 0.3±0.006a | 26.533±1.92b | 12.9±1.19c | 3±0.04c | 1.1a | 285.7±19.23a | 25.6±0.53a |
| UNA_SC-49 | 42.9±0.15a | 0.1±0.006d | 31.1±1.92b | 41.8±1.19a | 4.9±0.04b | 0.8d | 308.6±19.23a | 27.3±0.53a |
| UNA_SC-54 | 42.6±0.15a | 0.24±0.006b | 38.066±1.92a | 38.2±1.19b | 7.3±0.04a | 1b | 315.5±19.23a | 28±0.53a |
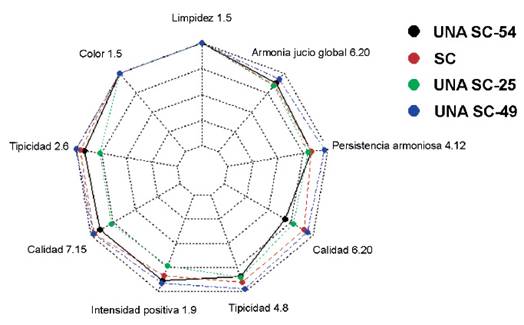
Figura 6 Análisis de las características organolépticas de los piscos Quebranta obtenidos de los vinos fermentados con tres cepas de levaduras nativas seleccionadas (UNA SC-49, UNA SC-25 y UNA SC-54) y una cepa control (Sc. Comercial).
Finalmente, los piscos evaluados a nivel organoléptico fueron clasificados de acuerdo con el artículo 44 del Reglamento del Concurso Nacional del Pisco (CONAPISCO 2016), entre buenos y excelentes, siendo el que obtuvo el mayor puntaje el pisco elaborado con la cepa UNA SC-49. La caracterización enológica ha permitido evidenciar en las condiciones experimentales descritas, cómo la cepa UNA SC-49 tiene la capacidad de conducir la fermentación alcohólica de manera eficiente, con alto rendimiento alcohólico, así como también aporte de características organolépticas peculiares al mosto fermentado y luego al destilado.












 uBio
uBio 


