Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Acta Médica Peruana
versión On-line ISSN 1728-5917
Acta méd. peruana v.23 n.3 Lima sep./dic. 2006
Tema de revisión
Genómica y proteómica: Un paso más
Genomics and proteomics: one further step
Franklin Aldecoa Bedoya1 y Carlos Battilana Guanilo2
1. Médico Oncólogo Clínico. Gerente Médico Laboratorios Roche.
2.Médico Internista Nefrólogo. Profesor de medicina UNMSM y Director médico de Laboratorios Roche.
RESUMEN
El desarrollo de la genómica y proteómica descansan sobre los descubrimientos fundamentales que constituyen hitos históricos sin los cuales no hubiera sido posible el hallazgo de nuevos paradigmas, teorías, tecnología, que han cambiado drásticamente el enfoque de la ciencia médica. El descubrimiento de la estructura del ADN y sus funciones básicas: replicación, transcripción y traducción, asociadas a la manipulación del material genético celular a través de las enzimas de restricción, ligasas, polimerasas, secuenciación de las bases nucleotídicas, nos llevaron a la ingeniería genética, el develamiento del genoma humano, la creación de nuevas herramientas para el estudio y diagnostico de las enfermedades, como el PCR (reacción en cadena de la polimerasa), microarray, y al mejor entendimiento acerca del comportamiento de nuestro organismo frente a los medicamentos (farmacogenómica). Ahora nos toca un reto superior: entender mejor el comportamiento de las proteínas como elementos básicos de vida, el alfabeto a través del cual el DNA genera vida. Estos nuevos conceptos traen consigo nuevas responsabilidades, problemas éticos y morales y obviamente la necesidad de una nueva legislación para este desarrollo.
Palabras clave: proteómica, genómica, geneterapia, biología molecular, clonación.
ABSRACT
The development of genomics and proteomics depends on fundamental discoveries that resulted in new paradigms, theories and technologies, that have drastically changed the focus of medical sciences. The discovery of the structure of DNA and the basic functions of replication, transcription and translation, associated to the manipulation of cellular genetic material through restriction enzymes, ligases, polymerases, nucleotide base sequences, etc., lead us to genetic engineering, the disclosure of the human genome, the creation of new tools for the study of diseases and diagnosis, such as PCR (Polymerase Chain Reaction), microarray technology, and the better understanding of the behavior of our organism in relation to medication (pharmacogenomics). Now we have a greater challenge: to improve our understanding of the behavior of proteins as basic elements, the machinery through which DNA generates life. These new concepts carry new responsibilities, bring up ethical and moral problems and the need for appropriate legislation for their development.
Key words: porteomic, genomic, genotherapy. molecular biology, Cloning.
INTRODUCCIÓN
En el año 2001 la humanidad fue testigo de un acontecimiento que acaparó la atención no solamente de todos los medios de comunicación, sino de las organizaciones científicas, gremios profesionales, instituciones, etc. La noticia se esparció rápidamente hasta el último confín del planeta: se había completado la secuenciación del genoma humano1. Desde que se inició el desarrollo de la genética con Mendel, la influencia de esta área fue grande, pero contribuyó en los cuidados de la salud de pocas personas, la mayoría no tuvo el beneficio directo de la genética2. La percepción que actualmente se tiene de la genómica es mucho más ambicioso, ya que ésta descansa en el acceso experimental directo del genoma total y su aplicación a condiciones comunes que incluyen muchos millones de personas en el mundo: cáncer, SIDA, tuberculosis, enfermedad de Parkinson y de Alzheimer entre otras patologías.
En la presente publicación pretendemos dar una mirada muy rápida y superficial a los aspectos que se relacionan directamente con el desarrollo de la genómica y proteómica; no podemos abarcar cada uno de los tópicos en forma completa, porque la cantidad de información en cada una de sus partes es compleja y desbordante. Iniciamos con una visión histórica de los eventos que condicionaron la evolución del conocimiento de los genes hacia el descubrimiento de la estructura del ADN, acontecimiento que sin duda es una de las piedras angulares en la rápida y sorprendente secuenciación de la información genética de una serie de organismos, hasta llegar al ser humano. Inmediatamente, incidimos en los procesos básicos para generar la información genética y que durante muchos años fue el dogma central de la biología molecular; la simplificación de estos procesos no es sencilla y muchos de los aspectos finos no son tomados en cuenta, por lo cual resultan decididamente incompletos. Laderivación del conocimiento científico a la forma aplicada podrá evidenciarse a través de una somera revisión de algunas de las principales tecnologías sobre las cuales se basan actualmente la genómica y proteómica, las cuales apuntan a ser de notable utilidad, cuando al final, la experiencia y simplificación lleven estas tecnologías al consultorio médico.
El otro aspecto que enfocamos, es el contexto terapéutico: ¿Cómo influirán los nuevos conocimientos sobre las posibilidades de búsqueda de nuevos lineamientos y blancos terapéuticos?; existen ya algunos nuevos medicamentos que han roto los moldes tradicionales de enfocar el tratamiento y a futuro esperamos mayor progreso; pero además, debemos entender mejor, qué sucede con la variación farmacocinética y farmacodinámica de muchos de los medicamentos que actualmente disponemos, en el contexto de la variabilidad genética de cada persona.
Posteriormente, abrimos una pequeña ventana para explorar algunos nuevos conceptos de la proteómica, área más reciente y con mayores dificultades por diferentes razones, muy rápidamente esbozadas aquí, que constituyen un verdadero reto para los investigadores actuales y para aquellas nuevas generaciones que se introduzcan en esta importante arista de la biología. La funcionalidad de las proteínas es ampliamente conocida por los médicos, sin embargo, muchas funciones no puede ser previsibles por los actuales sistemas de información, debido a las diferentes variables que condicionan este comportamiento. Finalmente, se registra una lista de las posibilidades del alcance de la investigación genómica a futuro: ¿Qué debemos esperar en los siguientes años en relación al conocimiento genómico?, ¿Cuál es la orientación de la investigación científica?, ¿Cómo influenciará sobre los aspectos biológicos, en la salud y en la sociedad?; preguntas que obviamente solo el tiempo podrá determinar.
BASES MOLECULARES DE LA GENÉTICA
1. Los hitos históricos más importantes del ADN Juan Gregorio Mendel, un monje agustino alemán (1822- 1884) es el padre de la genética. Utilizando el Pisum sativum o guisante de color hizo probablemente la contribución más grande de la iglesia a la ciencia. La planta elegida por Mendel se autofecunda obligatoriamente, y su polen, en condiciones normales, no puede alcanzar el estigma de otra flor. Sin embargo, abriendo la flor inmadura y retirando la antera que proporcionaría el polen y cubriendo posteriormente el estigma con polen de otra planta con distintas características, se produce una fecundación absolutamente experimental, cruzada, y controlada. Las conclusiones finales de sus estudios se conocen como las leyes de la genética de Mendel, son el modelo teórico sobre las cuales se basa aún mucho de lo que sabemos actualmente en la transmisión de los caracteres por herencia3,4. En 1902 Walter Sutton, correlacionó la asociación de los cromosomas maternos y paternos en pares y su posterior separación durante la división celular, con las leyes mendelianas de la herencia5. Posteriormente Thomas Morgan encontró en 1910 que el rasgo del color blanco de los ojos podía transmitirse junto a un factor que determinaba el sexo, en el cromosomaXde la mosca de la fruta (Drosophila)6, demostrando más tarde, que durante la meiosis existe un intercambio de material entre cromosomas homólogos (crossing-over) y que la probabilidad de un entrecruzamiento entre dos genes asociados, es proporcional a la distancia entre ellos en el cromosoma; por tanto, contando la frecuencia de entrecruzamientos entre alelos de un par de genes es posible mapear aquellos genes en el cromosoma7 (Morgan recibió el premio Nobel en 1933 por este trabajo).

Basados en un experimento de Griffith en 1928, en el cual infectando con Streptococcus pneumoniae a un grupo de ratones en los cuales la bacteria con cápsula (neumococo S) causaba la muerte en menos de 24 horas y que al usar la misma cepa de Streptococcus sin cápsula (neumococo R) no letal, asociada al neumococo virulento previamente muerto por calor, mataba nuevamente los ratones, Avery, Mac Leod y McCarty condujeron en 1944 su clásico experimento del principio transformante en el cual encontraron que el ADN purificado de las bacterias letales S transformaban a las bacterias no virulentas R a la forma letal8. Hasta ese entonces la opinión mayoritaria era que el ADN, aun siendo un importante constituyente de los cromosomas, era una molécula monótona y más bien estructural, y que de ninguna manera podía ser portadora de la información hereditaria. Sin embargo, a pesar de esta nueva evidencia, algunos suponían que dicha información posiblemente venía de las proteínas asociadas a los cromosomas y se tuvo que esperar casi diez años, hasta que Hershey y Chase confirmaron el rol deADN en su clásico experimento con bacteriófagos de virus9.
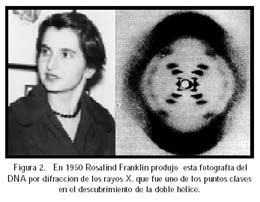
En 1950 el bioquímico Erwin Chargaff demostró que aunque la composición del ADN varia de especie en especie (sobre todo en lo relacionado a la cantidad de cada nucleótido), el número de bases adenina (A) era igual al de timinas (T) y el de citosinas (C) al de guaninas (G)10. Poco tiempo después Rosalind Franklin trabajando en el grupo deWilkins en Londres obtuvo una excelente fotografía por medio de la difracción de rayos X de las fibras deLADN. Sobre estos hallazgosWatson y Crick dedujeron que el ADN estaba formado por una doble hélice regular constituida por azucares (desoxirribosa) y fosfatos con las bases nucleotídicas en forma de peldaños formando enlaces A-T y C-G11.
Lamentablemente, Rosalind Franklin murió de una enfermedad leucémica y no pudo compartir el premio Nobel conWatson y Crick en 1962.
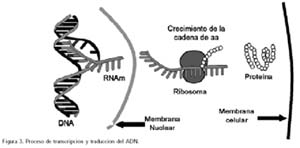
2. Dogma central de la biología molecular
La teoría de Watson y Crick fue conocida posteriormente como el Dogma Central de la Biología Molecular, basado en que elADN codifica los genes de un modo lineal estricto a través de un mediador: elARN, el cual es una plantilla para la síntesis de proteínas.
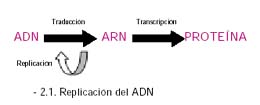
En las células procariotas la replicación comienza en un punto definido y avanza a la misma velocidad en ambas direcciones hasta finalizar con la duplicación del material genético. En los eucariotas en fase S del ciclo celular se inicia la duplicación en distintos puntos (replicones), en ambas direcciones. Sin embargo, en cada replicón la inserción de nucleótidos es continua en la dirección 5-3, pero en la cadena antiparalela (3-5) se forma por segmentos pequeños (fragmentos de Okasaki) de 1 000 a 2 000 bases; este proceso complicado implica muchos complejos enzimáticos, que permiten el desenrollar elADN, regular procesos, como las ciclinas entre otros. La replicación semiconservativa fue demostrada por Meselsonn y Stalh en 195812. Este mecanismo ha sido explotado en el proceso de secuenciación del ADN actual, usando complejos enzimáticos bacterianos.
- 2.2. Transcripción
Para que la célula pueda usar las instrucciones de la información genética, ésta debe trasladarse del núcleo al citoplasma, donde las proteínas serán construidas. El ARN de tipo mensajero (ARNm) es el intermediario, que realiza esta labor. ElARNm esta conformado por una cadena única que no forma doble hélice, con mucha menos estabilidad que el ADN (vida media en minutos). El proceso de transcripción es similar a la replicación del ADN, pero en la incorporación de nucleótidos se reemplaza la timina por el uracilo. El proceso es comandado por la enzimaARN polimerasa y solamente se copian sectores que codifican genes, dependiendo del tipo celular en la cual se lleve a cabo este proceso en el tiempo. Esta fase es muy compleja y al igual que la transcripción tiene varias enzimas que regulan su funcionamiento. La existencia en los eucariotas de los exones e intrones complica aun más este proceso; ahora sabemos que pueden existir varias formas de recorte a este nivel, de tal forma que un gen conformado por diversos exones e intrones tiene formas alternativas de cortado a nivel RNAm, de tal forma que un gen puede generar más de un tipo de proteína13.
- 2.3 Traducción
Para que un gen sea expresado el ARNm debe ser traducido a proteína. El problema era codificar 20 diferentes aminoácidos, contando para ello con solo 4 nucleótidos; no podía haber una correspondencia uno a uno, ni siquiera uno a dos, el número mínimo debiera ser al menos 3 nucleótidos por aminoácido (codón), pero las posibilidades de combinación eran (4 x 4 x 4) 64, de lo que se dedujo que había secuencias de codones redundantes o que existían codones sin sentido. En 1961 Niremberg y Matthaei demostraron que un polímero sintético de bases uracilo, añadido a una mezcla de ribosomas, ARNt (ARN de transferencia), aminoácidos, ATP (como fuente de energía) y otras enzimas, daban origen a una proteína monótona de fenilalanina, confirmando que el triplete UUU generaba dicho aminoácido14, a partir de entonces comenzaron a describirse nuevos tripletes ligados a sus aminoácidos respectivos, constituyendose finalmente en 1964 la tabla de la clave genética. El proceso de traducción es catalizado por un elemento molecular complejo: el ribosoma, el cual tiene un ARN ribosomal (RNAr) y proteínas.
Tecnología en biología molecular y genómica A partir de las observaciones de Watson y Crick, los cuales dedujeron brillantemente que ambas cadenas de la doble hélice eran complementarias, una cadena sirve de plantilla para la síntesis de una nueva copia, reteniendo por tanto la información original. Esto explica como la información genética es guardada en los cromosomas y como pasa a una célula que está en división.
Por tanto, si ambas cadenas complementarias son separadas en el laboratorio por calentamiento (desnaturalización), ellas pueden volver a unirse por la complementariedad de sus nucleótidos. Este proceso de separación y nueva unión es conocido como hibridización y es la piedra angular de un gran sector de la tecnología de la biología molecular.
La tecnología genómica es la aplicación de la automatización de la tecnología molecular básica a un sistema paralelo masivo. Metafóricamente, si la biología molecular provee las operaciones de corte, copiado y pegado para un determinado texto (gen), la tecnología genómica opera sobre múltiples genes al mismo tiempo13. Por tanto, para entender bien esta nueva tecnología de alto rendimiento, es necesario conocer sobre las técnicas de la biología molecular básica.
3. Técnicas de la biología molecular
3.1. Enzimas de restricción (cortar)
Son enzimas que cortan elADN. Son obtenidas de algunas bacterias que las usan como protección a invasión de ADN foráneo. El primero en obtener y purificar estas enzimas fue Hamilton Smith a principios de los 7015. Estas enzimas reconocen secuencias específicas de 4 a 8 nucleótidos lugar donde producen el corte. Se conocen actualmente más de 400 diferentes tipos.
3.2. Clonaje del ADN (copiar y pegar) Es un proceso mediante el cual se usan bacterias como hospederos, para insertar en su material genético o en los llamados plásmidos un segmento de ADN de interés y hacer que dicha bacteria se replique ilimitadamente. Fue Stanley Cohen quien logró insertar plásmidos que transportaban genes de resistencia a antibióticos de una bacteria a otra y posteriormente incluir material genético de otras especies en bacterias16.
El proceso comienza con el aislamiento de un fragmento deADN de interés mediante las enzimas de restricción; al mismo tiempo se aísla y abre el ADN circular de un plásmido con la misma enzima de restricción usada; inmediatamente se junta ambos materiales permitiendo que los fragmentos se peguen usando una ADN ligasa y se tenga un nuevo círculo de material plasmídico (plásmido quimérico). Las bacterias se dividirán rápidamente con el consiguiente aumento del gen insertado y la producción de la proteína de interés dentro de la bacteria, la cual será purificada para su uso posterior. Toda este proceso tiene el nombre de Tecnología del ADN recombinante o Ingeniería Genética.
3.3. PCR: Clonación sin bacterias
Mediante esta técnica, múltiples copias de un segmento de ADN específico pueden producirse por amplificación, usando un sistema de síntesis consecutiva que no requiere células vivas. Esta incluye una serie de reacciones a partir de la enzimaADNpolimerasa, por lo cual toma el nombre de Reacción de la polimerasa en cadena o PCR (iniciales en inglés). Mediante un segmento iniciador o primer de nucleótidos complementarios a un extremo de la secuencia deseada en una de las cadenas, y otro iniciador complementario en el otro extremo de la otra cadena, lograremos insertar los nucleótidos complementarios mediante la polimerasa y obtener millones de copias del segmento deseado en aproximadamente 20 ciclos. Mullis logró el premio Nobel por esta técnica en 199317.
La tecnología moderna de secuenciación del ADN está basada en el método de interrupción controlada de la replicación delADNdesarrollada por Fred Sanger en 1977, por el cual obtuvo el Nobel en 1980. Sanger combinó la maquinaria de replicación natural de las bacterias con algo de tecnología ADN recombinante y bioquímica inteligente para crear un sistema de clonación in vitro.
1.4. Método de Sanger: Secuenciación del genoma Usó nucleótidos especiales (dideoxinucleótidos) para cortar la replicación una vez que aleatoriamente una de estas bases se asociaba al proceso. Para visualizar los diferentes segmentos obtenidos eran radiomarcados y luego separados por electroforesis obteniéndose una placa radiográfica con la posición de acuerdo al tamaño molecular de los distintos segmentos deADN generados se podía colegir los nucleótidos en forma secuencial18.
4. Tecnología genómica
4.1. Secuenciación automatizada del ADN.
La primera gran innovación del método de Sanger la hizo Leroy Hood, al desarrollar nucleótidos fluorescentes que reemplazaron a los radioactivos en 1985. Estos podían ser medidos directamente en el gel de acrilamida por un detector laser; adicionalmente, cada nucleótido fue marcado con un color distinto, lo cual facilitó su identificación. Luego Hood conectó el proceso a una computadora y fundó Applied Biosystems, Inc. (ABI)19. En esencia todo el Proyecto Genoma Humano fue realizado en máquinas de ABI. Sin embargo, uno de las principales limitaciones del metodo de Sanger/ABI, fue que solo podía secuenciar fragmentos de 500 a 800 bases.
4.2. Subclonación shotgun.
El proceso de secuenciación de fragmentos grandes de ADN requería cortar con enzimas de restricción, ingresar la información a plásmidos (subclonación) y luego secuenciar.
Para aumentar la fidelidad, el proceso era duplicado. Sin embargo, aparte del tiempo consumido en tales tareas, habían muchos errores en los extremos de los fragmentos que deberían empatar con su complemento. Teóricamente, si un fragmento de ADN es copiado muchas veces, luego fragmentando en forma aleatoria (subclonación shotgun) y posteriormente secuenciados, si se tiene una suficiente cantidad de muestras, es posible reestablecer nuevamente el fragmento original a través de los extremos coincidentes de cada minifragmento.
A mediados de los 90 el Institute for Genomic Research (TIGR) logró automatizar todo el proceso de subclonación shotgun y secuenció en un tiempo realmente corto para la tarea, la secuencia completa del genoma del Haemophilus influenzae20. Celera Genomics Corporation más tarde, desarrolló un sistema de secuenciación basado en este principio, con lo cual impulsó la secuenciación del genoma humano y lograr objetivos más tempranos de los esperados21.
GENETERAPIA
La aplicación más evidente de este tipo de terapia es la corrección de algún defecto único de un gen, que corresponda a una enfermedad heredada. Lo racional es el reemplazo del gen dañado o alterado, por un gen normal trasladado a través de una serie de vectores hacia el contexto celular donde debiera funcionar. La introducción del material genético de reemplazo debe cumplir varios pasos: atravesar la membrana celular, cruzar el citoplasma y llegar al núcleo donde debe generar transcripción. De hecho cada paso implica muchos obstáculos que incluyen los mecanismos naturales de protección del la célula contra los elementos extraños, sobre todo a nivel nuclear. Los vectores pueden ser clasificados como de tipo viral o no viral.
Se han hecho algunos tratamientos experimentales en enfermedades como: la deficiencia de deaminasa de adenosina (ADA), en el cual existe una disfunción de los linfocitos T y B por acumulación de productos tóxicos derivados de la ausencia de la enzima ADA; fibrosis quística (FQ), una enfermedad hereditaria recesiva en la cual existe una mutación del gen regulador de conductancia transmembrana de la FQ, el cual expresa un canal de cloro alterado y cuya principal manifestación es la continua infección a nivel bronquial; hipercolesterolemia familiar, la cual es una enfermedad hereditaria dominante caracterizada por un defecto en el gen que codifica el receptor para lipoproteínas de baja densidad a nivel hepático.
Los resultados son aun irrelevantes y la toxicidad producida en un par de casos, en uno de los cuales hubo muerte del paciente, han generado opiniones adversas22-24. Sin embargo hay algunas publicaciones más recientes que muestran interesantes resultados en estudios experimentales en perros con hemofilia B25, amaurosis congénita de Leber y en cáncer26.
TECNOLOGÍA MICROARRAY
La base de la tecnología gene array radica en que un gran número de ADN de genes conocidos son pegados en la superficie de una matriz de pequeños microespacios (generalmente una laminilla de vidrio), sobre la cual se pone secuencias de ARN marcado, extraídas de algunas células de interés. Dicho RNA se ligará por hibridaciónRNA-DNAen la matriz. La cantidad de RNA marcado que se liga al DNA fijado a la matriz puede luego medirse13. Los experimentos array son siempre conducidos comparando dos o más muestras y miden la expresión genética, es decir la cantidad relativa de RNAm al momento del estudio y puede o no correlacionar con los niveles de transcripción y los niveles de proteína en la célula (pero, generalmente correlaciona bien).
El ensamblado robótico para utilización masiva del array toma el nombre de tecnología microarray; en un solo proceso se pueden evaluar miles de genes y es una herramienta flexible para medir la expresión genética en cualquier tejido del cual pueda aislarle el ARN. Las aplicaciones inmediatas de esta tecnología han sido básicamente en investigación y desarrollo, enfermedad y respuesta a drogas. Uno de los campos donde esta técnica puede colaborar es la patología; existen muchas neoplasias que difícilmente pueden tener un diagnóstico patológico seguro, mediante el uso del microarray podría hacerse un diagnóstico más fino, incluyendo la posibilidad de agrupar subtipos de cáncer dentro de una misma neoplasia (predicción de clase).Así por ejemplo Golub y colaboradores usaron esta técnica para distinguir entre leucemias agudas de tipo linfático y mieloide27, Alizadeh estableció grupos pronósticos en linfomas de células grande B, basados en el perfil de expresión genética28.
Asimismo, el pronóstico de otras enfermedades en el campo oncológico podrían variar, como en el cáncer de próstata, en el cual muchas veces existen incrementos del antígeno prostático específico asociado a un proceso inflamatorio al que pueden diagnosticar como neoplasia sin serlo, e incluso, la evaluación de la agresividad en caso de neoplasia y la correlación con el tratamiento respectivo. Sin embargo, antes de que esta tecnología llegue a ser ampliamente usada en el campo clínico, aun se requieren algunas consideraciones:
- Debemos estar seguros que lo que estamos midiendo es el material genético de las células que nos interesan y no de las células vecinas de soporte u otra estirpe.
- En los casos de tumor, lo más probable es que estemos midiendo solamente una de las tantas variaciones celulares del mismo tumor.
- La medida del contenido de RNAm es indirecta y pueden haber varios factores de error en la prueba.
- Existen genes muy similares en su conformación que comparten parte de su composición nucleotídica (homología) y podrían haber respuestas cruzadas.
- Los genes individuales pueden tener caminos alternativos de corte, en el momento en que el RNAm conserva aun exones e intrones.

FARMACOGENÓMICA
La fármacogenética es el estudio de cómo los diversos genes afectan la respuesta de las personas a las drogas; la farmacogenómica es definida como el uso de la información secuenciada delADN para medir, predecir la respuesta de un individuo a las drogas13.
Existen varios ejemplos en relación a interacciones de las drogas por problemas genéticos: durante la II guerra mundial se descubrió que cerca del 10% de población afroamericana tenía alelos polimórficos de la glucosa 6 fosfato deshidrogenasa que condicionaba anemia hemolítica cuando tomaban el antimalárico primaquina; aproximadamente 0,04% de la población general son homocigotes para alelos de la pseudocolinesterasa, los cuales no pueden inactivar el relajante muscular succinilcolina, lo cual termina en parálisis respiratoria; cerca del 10% de personas caucásicas son homocigotes para los alelos del gen CYP2D6 del citocromo P450 por lo cual no metabolizan la droga antihipertensiva debrisoquina; existen muchos alelos polimórficos del gen N-acetiltransferasa, que pueden reducir o aumentar la actividad de la isoniazida; finalmente, pacientes homocigotes para los alelos del gen de la enzima convertidora de angiotensina con una deleción del intrón 16 no muestran beneficio cuando usan enalapril. En los casos anteriores, conociendo el lugar de variación del nucleótido en el gen, podemos establecer pruebas simples de marcadores tipo SNP (single nucleotide polymorphism) para determinar que personas pueden tener estos problemas; pero además, podemos incluso cubrir una buena parte del genoma buscando SNPs que podrían asociarse con respuesta a drogas tanto en el aspecto de seguridad como de eficacia. En algunos casos, las diferencias entre las respuestas a una misma droga en un grupo de pacientes identificado por screening farmacogenómico podría indicar incluso, un mecanismo diferente de enfermedad13. Por tanto, la farmacogenómica puede proveernos con información confiable que puede ayudar al médico en la decisión terapéutica apropiada, con una dosificación justa de acuerdo ya no al peso, edad o sexo, sino basados en las características genéticas de la persona.
PROTEÓMICA
Es el estudio sistemático de las diversas propiedades de las proteínas para proveer detallada descripción de la estructura, función y control de los sistemas biológicos, esto incluye: secuencia, cantidad, modificaciones, interacciones con otras proteínas, etc. tanto en salud como en la enfermedad29. Dentro de la versatilidad de las proteínas encontramos que:
- Numerosas proteínas cambian su forma deliberadamente mientras realizan su funcion.
- Pueden asociarse selectivamente con colaboradores (cofactores) para llevar a cabo su tarea de forma más eficiente.
- Numerosas transformaciones postraducción afectan las propiedades de numerosas proteínas.
- Tienen la capacidad de diversificarse en conglomerados desde simples dímeros a cientos de subunidades30.
Cuantificación de las proteínas.
La medición de la cantidad de diferentes proteínas en un extracto celular es técnicamente más difícil que medir el ARNm con tecnología microarray, debido a la diversidad de las proteínas. El método más importante para cuantificar proteínas ha sido la electroforesis en gel de poliacrilamida en 2 dimensiones (2-D PAGE), el cual ha sido el caballito de batalla de la química proteíca por varias décadas: inicialmente se separan las proteínas por pH en una dirección, usando su punto isoeléctrico, posteriormente se separan por tamaño en la otra dirección en el mismo gel y por electroforesis, finalmente se visualizan en gel con tinciones especiales o por autoradiografía. Este método solo puede proveer un estimado aproximado. Sin embargo, la proteómica necesita un método de alto rendimiento que permita identificar y cuantificar un alto número de proteínas a la vez; nuevas tecnologías como la espectrometría de masa asociada a procedimientos que ionizan las moléculas proteícas con laser en un campo eléctrico, para luego medir la cantidad de tiempo hasta que los iones alcancen el detector son actualmente las más usadas; sin embargo, aún no es posible un mismo procedimiento para todo este proceso.
Interacciones proteína-proteína.
Algunas proteínas interactúan con otras para formar complejos moleculares multi-subunidades, para regular la función de otras proteínas o para ser reguladas. La extensión y naturaleza de estas interacciones son importantes para entender las vías regulatorias y metabólicas y para la caracterización funcional de muchas otras que están siendo descubiertas. Varias tecnologías están disponibles para evaluar estas interacciones, sin embargo, ninguna tiene capacidad para analizar el conjunto completo de una célula u organismo. Proteómica estructural Se espera conocer más acerca de las funciones biológicas de las proteínas a partir de su estructura tridemensional (3D), más que de su secuencia aminoacídica o su peso molecular. Actualmente, no es posible predecir la estructura tridimensional, basándonos solamente en su secuencia primaria o por los datos de la espectrometría de masa. Este trabajo pasa por la purificación y cristalización de la proteína, la cual será posteriormente sometida a cristalografía por rayos X o resonancia magnética nuclear. Sin embargo, es posible correlacionar la información conocida y acopiada en bases de datos especiales, para predecir la conformación 3D de proteínas con secuencias similares (hasta en 30%), proceso llamado threading.
Blancos de drogas
Hasta hace poco tiempo, las compañías farmacéuticas habían creado cerca de 500 blancos terapéuticos dirigidos a proteínas. Con la aplicación de la genómica al desarrollo de nuevas drogas se espera que este número crecerá a cerca de 4 000 en los próximos años. Particularmente, la tecnología microarray será fundamental para este crecimiento.
Visión de la genómica
Sobre la base del Proyecto Genoma Humano y contando con pilares como: recursos, desarrollo de tecnología, biología computacional, entrenamiento, educación y aspectos éticos y sociales, se configuran tres grandes áreas dentro de las cuales existen grandes retos a llevar a cabo en los siguientes años31.
I. La genómica hacía la biología: elucidar la estructura y función de los genomas.
- Identificar comprensivamente los componentes estructurales y funcionales codificados en el genoma humano.
- Dilucidar la organización de la red genética y las vías de las proteínas y como ellas contribuyen al fenotipo celular y del organismo. (c) Entender las variaciones hereditarias en el genoma humano.
- Entender las variaciones evolucionarias entre las especies y los mecanismos subyacentes.
- Desarrollo de opciones que faciliten la diseminación de la información genómica tanto en el contexto clínico como investigacional.
II. La genómica hacia la salud: Trasladar el conocimiento basado en el genoma hacia los beneficios de la salud.
- Desarrollar fuertes estrategias para identificar contribuciones genéticas a la enfermedad y la respuesta a las drogas.
- Desarrollar estrategias para identificar las variaciones genéticas que contribuyen a la buena salud o a la resistencia a la enfermedad.
- Desarrollar herramientas basadas en el genoma que predigan la susceptibilidad a la enfermedad y la respuesta a las drogas, detección temprana y taxonomía molecular de la enfemedad.
- Usar el conocimiento de los genes y sus vías para desarrollar nuevos y poderosos enfoques terapéuticos contra la enfermedad.
- Investigar cómo es comunicado el riesgo genético en el contexto clínico, cómo influencia esa información en las estrategias de la salud y en el comportamiento, asimismo, cómo influyen estos en los resultados y los costos.
- Desarrollar herramientas basados en el genoma, para mejorar la salud de todos.
III. La genómica hacia la sociedad: Promover el uso de la genómica para maximizar beneficios y minimizar daños.
- Desarrollar opciones para el uso de la genómica en el contexto médico y no médico.
- Entender la relación entre genómica, raza, etnicidad y las consecuencias de no establecer estas relaciones.
- Entender las consecuencias de no desarrollar las contribuciones genéticas a los rasgos humanos y el comportamiento.
- Politicas efectivas para definir lo asociado a la ética en el uso del genoma
REFERENCIAS BIBLIOGRÁFICAS
1. Venter JC, Adams MD, Myers EW, et al. The secuence of the human genome. Science 2001;291:1304-1351. [ Links ]
2. Guttmacher AE, Collins FS. Genomic Medicine -A primer- .New Engl J Med.2002;347:1512-1520. [ Links ]
3. Mendel, G. Bersuche ubre Pflanzen-Hybriden. Berhandlungen des naturforschenden Vereines, Abhandlungen, Brünn 1866;4:3-47. [ Links ]
4. Hernández Bronchud M. La Industria del ADN: Principios básicos y potencial futuro. Editorial MCR S.A. Barcelona 1992. [ Links ]
5. Sutton W. The chromosomes in heredity. Biol Bull 1903;4:231-251. [ Links ]
6. Morgan T. Sex-limited inheritance in Drosophila. Science 1910;32:120-122. [ Links ]
7. Morgan T. The physical basis of heredity. Philadelphia: Lippincott, 1919. [ Links ]
8. Avery OT, McLeod CM, McCarty M. Studies on the chemical nature of the substance inducing transformation of pneumococcal types. J Exp Med 1944;79:137-158. [ Links ]
9. Hershey AD, Chase M. Independent functions of viral proteins and nucleic acid in growth of bacteriophage. J Gen Physiology 1952;36:39-56. [ Links ]
10. Chargaff E. Chemical specificity of nucleic acids and mechanisms of their enzymatic degradation. Experientia 1950;6:201-209. [ Links ]
11. Watson JD, Crick FHC. A structure for deoxyribose nucleic acid. Nature 1953;171-173. [ Links ]
12. Meselson M, Stahl FW. The replications of DNA in Escherichia coli. Proc Natl Acad Sci USA 1958;44:671-682. [ Links ] 13. Brown S. Essential of Medical Genomics. Wiley-Liss Inc, New Yersey. 2003. [ Links ]
14. Niremberg et al. RNA code words and protein synthesis, VII. On the general nature of the RNA code. Proc Natl Acad Sci USA 1965;53:1161-1168. [ Links ]
15. Smith HO, Wilcox KW.A restriction enzyme from haemophilus influenzae. I.Purification and general properties. J Mol Biol 1970;51:379-391. [ Links ]
16. Cohen SN, Chang AC, Boyer HW, Helling RB. Construction of biologically functional bacterial plasmids in vitro. Proc Natl Acad Sci USA 1973;70:3240-3244. [ Links ]
17. Saiki RK et al. Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science 1988;239:487-491. [ Links ]
18. Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci USA 1977;74:5463-5467. [ Links ]
19. Smith LM, Sanders JZ, Kaiser RJ, et al. Fluorescence detection in automated DNA sequence analysis. Nature 1986;321:674-679. [ Links ]
20. Fleischmann RD, Adams MD, White O, et al. Wholegenome random sequencing and assembly of Haemophilus influenzae Rd. Science 1995;269:496-512. [ Links ]
21. Venter JC, Adams MD, Myer EW, et al. The sequence of the human genome. Science 2001; 291:1304-1351. [ Links ]
22. Blaese RM et al. T lymphocyte-directed gene therapy for ADA-SCID: Initial trial results after 4 years. Science 1995;270:475-480. [ Links ]
23. Crystal RG et al. Administration of an adenovirus containing the human CFTR cDNA to the respiratory tract of individuals with cystic fibrosis. Nature Genet 1994;8:42- 51. [ Links ]
24. Grossman M et al. A pilot study of ex vivo gene therapy for homozygous familial hiper-cholesterolemia. Nature Med 1995;1:1148-1154. [ Links ]
25. High KA. Gene transfer as an approach to treating hemophilia. Circ Res 2001;88:137-144. [ Links ]
26. Khuri FR, Nemunaitis J, Ganly I, et al. A controlled trial of intratumoral ONYX-015, a selectively-replicating adenovirus, in combination with cisplatin and 5-fluorouracil in patients with recurrent head and neck cancer. Nature Med 2000;6:879-885. [ Links ]
27. Golub TR, Slonim DK, Tamayo P, et al. Molecular classification of cancer: Class discovery and class prediction by gene expression monitoring. Science 1999;286:531- 537. [ Links ]
28. Alizadeh AA, Eisen MB, Davis RE, et al. Distinct types of diffuse large B-cell lymphoma identified by gene expression profiling. Nature 2000;403:503-511. [ Links ]
29. Patterson SD, Aebersold RH. Proteonomics: the first decade and beyond. Nature genetics supplement. 2003;33:311- 323. [ Links ]
30. Hol WG. Perspectives: Structural genomics for science and society. Nature structural biology. Structural genomics supplement. Nov. 2000. [ Links ]
31. Collins FS, Green ED et al. A vision for the future of genomics research. Nature.2003;422:835-847. [ Links ]
Correspondencia:
Dr Carlos Battilana Guanilo
carlos.battilana@roche.com