Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Acta Médica Peruana
versión On-line ISSN 1728-5917
Acta méd. peruana v.26 n.2 Lima abr./jun. 2009
ARTÍCULO DE REVISIÓN
Un nuevo virus A/H1N1, una nueva pandemia: Influenza un riesgo permanente para una humanidad globalizada
A new virus A/H1N1, a new pandemic: a continuing risk to a globalized humanity
Fernando Osores Plenge1,7 ; Jorge Gómez Benavides2,7 ; Luis Suarez Ognio3,7,8 ; César Cabezas Sánchez4,7 ; Jorge Alave Rosas5 ; Ciro Maguiña Vargas6,7,8
1. Médico Investigador. Infectologo Tropicalista Magister. Profesor de la Facultad de Medicina Humana de la Universidad Ricardo Palma, Lima, Perú. Coordinador del Instituto de Investigaciones en Ciencias Biomédicas INICIB- FAMURP, Lima, Perú.
2. Médico Epidemiólogo. Profesor de la Universidad Mayor de San Marcos. Dirección General de Epedimeologia, Ministerio de Salud, Lima, Perú.
3. Médico Epidemiólogo de la Dirección Regional de Salud de Ica, Profesor de Epidemiología y Salud Publica de la Universidad Nacional San Luis Gonzaga de Ica y de la Universidad Peruana de Ciencias Aplicadas UPC, Lima Perú.
4. Médico, Infectologo Tropialista. Profesor de la Universidad mayor de san Marcos. Sub Jefe del Instituto Nacional de Salud, Ministerio de Salud, Lima Perú.
5. Médico Infectólogo Tropicalista, Sub Jefe del Instituto Nacional de Salud, Ministerio de Salud, Lima Perú.
6. Médico Infectólogo Tropicalista, Dermatólogo. Profesor Principal y Director Adjunto del Instituto de Medicina Tropical "Alexander von Humboldt" de la Universidad Peruana Cayetano Heredia, Lima, Perú.
7. Grupo de análisis y seguimiento de la influenza estacional y riesgos pandémicos, Lima Perú.
RESUMEN
La influenza es una enfermedad altamente infectocontagiosa de la cual se tienen registros históricos descriptivos desde la época griega y de certeza etiológica tan solo hace casi ocho décadas atrás. Su agente causal es el virus del influenza de los que se conoce existen tres grandes tipos: A B y C. El tipo A tiene la propiedad de circular por diversos reservorios biológicos, tales como el hombre, los cerdos y las aves, entre otros. Representa además una elevada variabilidad genética lo que le permite continuos cambios o derivas antigénicas menores responsables de la influenza humanas epidémicas y a veces reordenamientos amplios con cambios antigénicos mayores los que originan la temida influenza pandémica.
En un mundo cada vez mas globalizado, con una población superior a los seis mil millones de personas, marcado por grandes inequidades sociales y con cambios climáticos evidentes, los virus de la influenza serán un riesgo permanente para la seguridad de la humanidad.
La clínica de los diversos subtipos virales pueden balancearce desde las formas inaparentes hasta las formas graves de gripe o influenza, dependiendo de la virulencia del subtipo viral infectante y del huesped. Aunque nuestra tecnología diagnóstica y de inmunización ha avanzado sorprendentemente, la preparación y disposición de los nuevos kits diagnósticos suelen tardar al principio y en el caso de las vacunas estas no están disponibles para el un nuevo subtipo viral pandémico en el momento que se las necesita. El desarrollo de antivirales contra la gripe no es notorio, contándose aprobados para uso humano los amadantanes como la amantadina y la rimantadina y los inhibidores de la neuraminidasa como oseltamivir y sanamivir principalmente. Claramente los más vulnerables en este contexto son los países en vías de desarrollo y en especial aquellos mas pobres, hecho que nos debería llamar a una profunda reflexión.
Palabras Clave: influenza, gripe, variabilidad antigénica, neuraminidasa, hemoaglutinina, nueva influenza A/H1N1, pandemia.
ABSTRACT
Influenza is a highly contagious disease. There are some historical descriptions of this condition by ancient Greek physicians, and the etiological agents have been known only for the last eight decades. The causative agent is the influenza virus, which has three main types: A, B, and C. Type A is capable of circulating within many different biological reservoirs, including humans, swine, and birds. It also has high genetic variability, which allows it to have minor antigenic drifts or mutations which are responsible of epidemics in humans. Sometimes changes are quite marked, leading to pandemics.
In a globalized world, with more than 6 billion inhabitants, with many social inequities and evident climate changes, influenza viruses are a permanent risk for mankind.
Clinical features for the different viral subtypes may vary from subtle infections to full blown and severe, life-threatening forms. Event with the great advances in diagnostics and immunization, the manufacture and distribution of new diagnostic kits may take some time, and new vaccines are not always readily available. Specific therapies against influenza are not well developed. There are two groups of drugs, the so called adamantane derivatives, such as amantadine and rimantadine, and the neuraminidase inhibitors, such as oseltamivir and zanamivir. Most vulnerable areas for the new flu pandemics include developing countries, particularly the poorest ones, so that the greatest effort must be made for helping these areas.
Keywords: influenza, antigenic drift, neuraminidase, hemaglutinin, novel influenza A H1N1, pandemic.
INTRODUCCIÓN
Los virus de la influenza o gripe (VI) son diversos, perteneciendo a la familia de los Ortomixovirus, un grupo de virus ARN de sentido negativo agrupados en cinco géneros, tres de ellos conocidos como virus de la influenza tipo A, B y C (VI-A, VI-B y VI-C)1, y los dos restantes denóminados isavirus y thogovirus2. Sin embargo son los tres primeros géneros de esta familia los que llaman nuestra atención en este artículo de actualización, y aunque tanto los VI-A, VI-B y VI-C mantiene una dinámica ecopidemiológica constante, son preferentemente los VI-A pertenecientes a los linajes humano(VIAH), porcino (VIAP) y aviar (VIAA) y sus subtipos derivados los causantes de las grandes pandemias y epidemias estacionales humanas, epizootias, endozootias y zoonosis, tendiendo a variar constantemente en el tiempo3-,13, circulando por todo el globo terráqueo principalmente a través de las migraciones humanas y animales favorecidas por la globalización, la pobreza, el hacinamiento, los conflictos sociales y el cambio climático14-20.
Los VI, primordialmente los subtipos de VI-A, tienen el potencial de infectar eficientemente a las células del epitelio ciliado del tracto respiratorio humano a través del contacto persona a persona, por aereosoles producidos durante el estornudo, la tos y la respiración misma, generando enfermedad respiratoria aguda alta y baja, la mayoría de ellas altamente transmisibles entre humanos con un espectro epidémico y clínico que oscila desde las formas inaparentes-leves a las altamente graves y mortales21-,25.
Normalmente, año a año los VI-C, VI-B y con especial impacto e importancia para la salud pública los VI-A circulantes van mutando26,27 generando cambios sutiles en sus hemaglutininas, neuraminidasas y otros componentes de sus estructuras – el VI-C no posee hemaglutininas-. Esto es lo que se conoce como deriva antigénica28, fenómeno que genera nuevos subtipos virales parecidos a los del año anterior por lo que nuestro sistema inmune mantiene un grado relativamente elevado de reconocimiento y respuesta a estos mutantes. Es este, el origen de las influenzas estacionales o epidémicas, que afectan entre el 5 a 20% de la población mundial, produciendo una gran morbimortalidad global, estimándose de uno a tres millones de casos graves y entre 250 000 y 500 000 ( 694,4 -1 388,8 muertes por día al año) la cifra de muertos anualmente por estos virus, concentrándose la letalidad principalmente en los extremos etarios de la población, es decir niños y ancianos29-30.
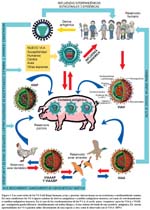
Tanto aves, cerdos, caballos, humanos entre otros seres del reino animal tienen su propio linaje de VI-A. Estos distintos linajes VI-A y sus diversos subtipos evolutivos poseen la particularidad de poder infectar no sólo al hombre (gripe humana)31 sino a diversas especies entre las que se encuentran primates, aves (gripe aviar), porcinos (gripe suina), equinos (gripe equina), entre otras33-3,4, y si bien cada especie tiene su linaje y subtipos propios, de tanto en tanto estos pueden saltar de una especie a otra directamente35-39 o pueden recombinarse en un en un hospedero común40 para generar nuevos subtipos desconocidos para la misma u otra especie afectada41. En el caso específico de la especie humana los porcinos parecen tener un rol primordial en las recombinaciones42 ya que poseen receptores tanto para los subtipos de VIAA así como para los subtipos de VIAH.
Desde esta perspectiva, en el último siglo la humanidad vivió tres pandemias y un sinnúmero de epidemias estacionales, del mismo modo registró en su devenir temporal la reaparición del linaje del VI/H1N1(influenza rusa), descendiente directo del letal VI/H1N1 de 1918, del cual la evidencia científica indica un origen a partir de un ancestro viral parecido al aviar43, que se estima pudo estar previamente circulando de manera ineficiente entre 1915 y 191744, pero que no se distribuyó ampliamente entre humanos y cerdos hasta que en 1918 finalmente infectó a ambas especies dando origen a la primera y peor pandemia del siglo XX conocida como gripe española42,45,46 –se dice de ella oficialmente que cobró la vida de 40 a 50 millones de personas en todo el mundo47-50– además vió la aparición -por reordenamiento de virus aviares y humanos que tuvieron como hospedero al cerdo- de dos nuevos virus pandémicos en ese mismo siglo, el H2N2 o influenza asiática y H3N2 o influenza de Hong Kong51-53, así como de nuevos subtipos netamente aviares y porcinos capaces de infectar –aunque ineficientemente- al ser humano de manera directa.
Estas influenzas estacionales y pandémicas no han sido ajenas al Perú. Así por ejemplo la mal llamada influenza española, se estima llegó entre enero y marzo de 1919; no contándose con datos precisos sobre la mortalidad y letalidad causada en la población de nuestro país en esa época. Esta pandemia fue estudiada por el Dr. Aníbal Corvetto Bisagno54-56, y por el Dr. Edmundo Escomel quien refiere: En el perú por felicidad ha sucedido lo mismo, habiendo sido la epidemia general pero poco grave y si bien en Arequipa se afirma haber existido 5 000 casos, muy pocos han sido fatales, limitándosé a los tarados o insuficientes orgánicos.En el Cuzco y Caylloma parece haber sido la mortalidad mayor57.
La segunda pandemia o gripe asiática, llegó al Perú en la segunda quincena de julio de 1957, siendo documentada por los Drs. Roberto J. Cornejo Ubilluz y Jorge Villena Pierola. Los primeros casos se reportaron en las ciudades fronterizas con Chile como Tarata, Locumba y Tacna, en el departamento de Tacna, o con Bolivia, como Yunguyo y Desaguadero, en el departamento de Puno, se diseminó de sur a norte hasta llegar a Lima en la primera semana de agosto, las últimas ciudades afectadas fueron Iquitos, Tumbes y Chachapoyas a donde llegó la tercera semana de diciembre. Las tasas de ataque observadas fluctuaron entre 20 y 40% en la costa, mientras que en la sierra, fluctuaron en alrededor del 70%, se notificaron al Departamento de Epidemiología del Ministerio de Salud Publica y Asistencia Social, nombre con el que se conocía el Ministerio de Salud, 135 292 casos con una tasa de ataque de 15 por cada 1000 habitantes y 1 081 defunciones (letalidad = 0,8%)58.
Luego de esto, diversos subtipos del linaje humano A H3N2, H1N1 y VI-B han seguido circulando en el Perú59-65 y aunque los estudios al respecto son escasos, se ha logrado demostrar múltiples subtipos virales circulantes en el Perú66,67.
Al 2000 se estimó que los años de vida y la tasa de años de vida pérdida por enfermedad transmisible atribuible a la influenza y neumonía fue de 424 602,0 y 16,3768
Por último, en esta constelación de sucesos, ya en pleno siglo XXI desde marzo-abril estamos siendo oficialmente testigos activos de la aparición de un nuevo subtipo viral A/ H1N1 humano –inicialmente denominado virus de la influenza porcina- que ha llegado a convertirse en un fenómeno pandémico, y del cual se afirma es totalmente desconocido para la especie humana. Este y otros subtipos virales humanos, aviares y porcinos cocirculantes podrían estar marcando la pauta para la gestación de un nuevo virus de la influenza altamente transmisble, virulento y mortal entre humanos en un mundo globalizado con aproximadamente de 6 500 000 millones de habitantes69.
INFLUENZA: UNA HISTORIA COMPLEJA DE DERIVAS, REORDENAMIENTOS Y SALTO DE ESPECIES
El término influenza fue introducido en el siglo XV durante la edad media y se le atribuye al Papa Benedicto XIV70 para describir epidemias que eran atribuidas a las influencias astrales, derivando de la palabra latina influentia, aunque también se sostiene que otro posible origen es la expresión influenza di freddo, o sea, por la influencia del frío71. Este término fue adoptado por los ingleses en el siglo XVIII y durante el mismo periodo los franceses denominaron la enfermedad como la grippe72.
Sin embargo desde el siglo 400 a.C., en los escritos médicos griegos El Libro IV de la Epidemias, ya es posible encontrar referencias hechas por Hipocrátes de lo que podría ser una enfermedad similar a la IH48,54,73,74. También existen registros que datan de 1173 d.C que han sido tomados como las primeras descripciones probables de influenza epidémica72.
Del mismo modo, datos históricos posteriores indican que el 9 de diciembre de 1493, la segunda expedición de Cristóbal Colón con 17 barcos y 1 500 hombres aproximadamente, un día después de llegar al poblado La Isabela en la isla La Española sufrieron una enfermedad caracterizada por fiebre, síntomas respiratorios y malestar que duraban en su mayoría de cuatro a cinco días de manera muy similar a la gripe, denominada romadizo y que causó gran mortalidad entre los españoles y estragos entre los indios. Junto a esta expedición llegaron cerdos, caballos y gallinas comprados en la isla Gomera. Estos encontrarían en el archipiélago Caribeño una abundante biodiversidad de aves acuáticas no conocidas previamente75 y a los indios precolombinos, quienes carecían de cerdos y caballos76
A pesar de ello, la primera pandemia claramente registrada se originó en Asia en el año 158072En los siguientes siglos, se reportaron importantes pandemias de influenza en los años 1729, 1789, 1830 y 189872,77,78. Robert Johnson en 1793 describió con claridad el brote de 1789 ocurrido en los Estados Unidos de Norteamérica como una enfermedad que causa postración con un repentino cuadro febril, que dura de cuatro a cinco días y que es seguido de un período de recuperación que dura varias semanas con tos y debilidad y que ataca a personas de mediana edad79.
Así en los últimos 300 años han ocurrido 10 pandemias causadas por virus de influenza, con un promedio de 33 años cada una72,80.
En 1892, Richard Pfeiffer identificó el Haemophilus influenzae, él y otros eminentes microbiológos erróneamente llegaron a la conclusión de que esta bacteria gram negativa de forma variable era la causa de la gripe81. Así, la etiología de la influenza permanecería desconocida hasta 1930 -1933, en que se logró aislar por primera vez a nivel de laboratorio el agente etiológico de esta enfermedad, primero en cerdos por Richard Schope y luego en humanos por un grupo de investigación dirigido por Patrick Laidlaw y el Medical Research Council del Reino Unido57,82,83
Durante el siglo XX la primera gran pandemia de IH ocurrió entre 1918 y 1919, y fue denominada, durante la Primera Guerra Mundial, como gripe o influenza española. Aunque el virus no se originó en España, este fue el primer país afectado del continente europeo, donde se le dio una gran cobertura de prensa a los primeros estallidos tempranos de la enfermedad, motivo por el cual se le otorgó equivocadamente esta triste denominación de origen47,72.
Aunque el punto de origen de la pandemia de 1918 es en la actualidad controversial, es aceptado que la primera evidencia fehacientemente registrada ocurrió el 11 de marzo de 1918 en el Fuerte Riley, estado de Kansas, en Estados Unidos (EE UU)84.
La segunda y tercera pandemias, con un estimado de 4 millones y 1,5 millones de muertes respectivamente, ocurrieron en 1957 y en 1968 y se denominaron influenza asiática e influenza de Hong Kong, fueron producidas por dos subtipos virales emergentes denominados H2N2 y el H3N270,72
En este contexto histórico de la influenza o gripe, se conoce como deriva viral antigénica ó variación antígénica menor al proceso por el cual los VI-A y VI-B pueden con facilidad generar mutaciones espontáneamente produciendo y acumulando sutiles cambios en alguno de sus componentes antigénicos de membrana en especial a nivel de las glicoproteínas conocidas como hemoaglutininas y neuraminidasas28,41,85.
Mientras tanto, el reordenamiento o reemplazo viral antigénico (variación antígénica mayor), sólo ocurre en VI-A y es hasta a la fecha un proceso esporádico que se caracteriza por el cambio total de hemoaglutininas y en menor medida neuroaminidasas, originando un nuevo subtipo viral para el cual el sistema inmunitario humano no tiene ningún tipo de defensa, pues simplemente le es totalmente desconocido84,86. Ver Figura 1.
Mucho se ha discutido acerca de quién y cuando se causará la próxima pandemia de influenza humana (PIH). Hay consenso de que lo más probable es que esta sea causada por un subtipo aviar reordenado con otro humano utilizando al cerdo como hospedero intermediario –lo que de hecho ya ocurrió en las pandemias de 1957 y 1968-, o en su defecto evolucione saltando directamente a la especie humana con una tasa de trasmisibilidad óptima para desencadenar un escenario pandémico crítico, parecido o peor al ocurrido en 191887,88.
El origen del VI-A H1N1 de 1918 es aún materia de controversia89,90. El recocimiento de su ARN fue posible a partir de muestras parafinadas de pulmón almacenadas y directamente de tejido pulmonar congelado de víctimas de la influenza de 1918, que fueron entrerradas y conservadas naturalmente en el suelo congelado (permafrost) de la gélida Alaska43,91,92.
Aunque en la actualidad la secuencia viral reconstruida del VI de 1918 indica que prácticamente todo el virus era desconocido para la especie humana de aquel entonces, lo que descartaría la posibilidad de reordenamientos previos a partir de subtipos aviares circulantes, el análisis filogenético del virus deja abierta la alta probabilidad de que un virus aviar desconocido finalmente salto de especie de manera directa transmitiéndose efectivamente entre humanos41,93,94, a su vez que casi simultáneamente en el tiempo, también lo hacia en cerdos45,95-98.
Hoy se sabe que los descendientes posteriores del subtipo H1N1 de 1918 siguieron circulando por deriva antigénica entre humanos hasta la década de los cincuenta, para luego desaparecer con la entrada en escena del VIAH H2N2 que dio origen a la influenza asiática; sin embargo a nivel de los cerdos la circulación de subtipos porcinos H1N1de manera enzoótica persistió. En 1977 un subtipo del linaje humano H1N1 descendiente del VI-A de 1918 volvió a reaparecer99. Desde aquella época hasta el presente solo tres (H1, H2 y H3) de los 16 subtipos de hemaglutininas y dos (N1, N2) de los 9 subtipos de neuroaminidasa han circulado establemente en el humano y desde 1968 al 2009 la H2 no es aislada27,100.
Ninguno de los descendientes directos del VI-A H1N1 de 1918, tanto a nivel del linaje humano como del linaje enzootico porcino o influenza porcina clásica ha mostrado la patogenicidad de su ancestro. En adición el VI H2N2 ha dejado de circular, no siendo detectado a la fecha, mientras que la aparición por reordenamiento del un nuevo VI-A H3N2 o influenza de Hong Kong en 1968 a dado origen por deriva antigénica a un linaje humano y a otro enzoótico porcino H3N286. Todos estos subtipos virales cocirculan simultáneamente desde hace 32 años, incrementando la probabilidad de reordenamientos genéticos27,100. De hecho hay casos de redistribución de segmentos de genes entre VIH, VIA y VIP que ocurren en los cerdos de manera natural101-103
En los últimos 20 años el subtipo predominante del linaje humano que ha circulado ha sido el H3N2, salvo en los períodos 1998-1999 y 2000-2001 en el que circuló preferentemente el H1N1104. De hecho la deriva antigénica del virus H3 ocurren más a menudo, y sus nuevas variantes tienden a sustituir a las antiguas rápidamente, de modo que la evolución de la hemaglutinina H3 es más lineal que en los otros subtipos de VI104,105, presentando períodos relativos de calma de entre tres a cinco años seguido de periodos de rápido cambio antigénico105. El significado epidemiológico de estas variaciones antigénicas mas rápidas en el virus H3 es que suelen ir acompañadas de epidemias de influenza mucho más graves105.
En el 2001 un nuevo subtipo VIAH denóminado H1N2 se generó posiblemente por el reordenamiento entre los subtipos H3N2 y H1N1 cocirculantes106-107, luego en el 2002, otro nuevo subtipo parecido al AH3N2/ Fujian/411/02 surgió por deriva antigénica en Asia y causó brotes en todos los continentes108,109.
En el hemisferio norte durante el invierno de 2003-2004 este subtipo viral antigénicamente mutado produjo una severa ola epidémica de gripe estacional, recordándonos la potencialidad patógena de la deriva antigénica86.
Del 2004 al 2009 VIH subtipos H1N1, H3N2 e influenza B han cocirculado, predominando el H3N2. En simultáneo a estos eventos cepas aviares (H5N1, H7N7, H9N2) y porcinas capaces de enfermar al hombre directamente también han estado presentes110,111.
El salto de especies es un aspecto crítico en la peligrosidad que afecta solo a los VI-A. Subtipos aviares principalmente han demostrado en las últimas décadas la capacidad de poder pasar a la especie humana de manera directa produciendo enfermedad grave, pero con un grado de transmisibilidad deficiente, impidiendo su diseminación masiva hasta el momento entre humanos112. La circulación de diferentes cepas virales de linaje aviar con fenómenos de derivas antigénicas y reordenamientos semejantes a los ocurridos en 1918 parecen estar ocurriendo nuevamente, lo que nos indicaría que los procesos microecológicos a nivel viral podrían estar marcando al igual que en 1918 la pauta previa a la aparición de un subtipo viral no solamente adaptado para transmitirse con relativa eficiencia como el nuevo subtipo A/H1N1, sino que a diferencia de este último, poseería una gran virulencia con aterradoras tasas de ataque y mortalidad109. Independientemente de en donde aparezca por primera vez este VI-A, el mismo no será recordado por la desenfrenada información mediática que lo seguirá, sino por las miles de muertes humanas, el colapso social y de la actividad económica a nivel mundial. Así, con solo una tasa de ataque del 10% y una letalidad del 50%, menor a la del VIAA H5N1 estaríamos hablando de 300 millones de muertes a nivel global.
En retrospectiva histórica y epidemiológica ya en 1983, una infección del VIAA H5N2 apareció en pollos y pavos en el estado de Pennsylvania y se convirtió en altamente patógeno para las aves de corral. Virológica y serologicamente no hubo evidencia de transmisión a seres humanos113. Luego en 1994 se aisló un nuevo subtipo de VIAABP H5N2 relacionado a aves marinas, el que infectó a aves causando enfermedad subclínica o leve en las mismas, sin embargo este mismo virus mutó en el transcurso de un año de un VIAABP a otro VIAAAP H5N2 en el que se identificó una nueva inserción de dos aminoácidos en su HA produciendo una elevada mortalidad en aves de corral114. Estos brotes demostraron que los mercados de ventas de aves vivas desempeñaban un papel fundamental en la propagación viral115. Senne et al. describe a los mercados de aves vivas como el eslabón perdido en la epidemiología de la gripe aviar 116.
A pesar de que hay reportes previos de transmisión de VIAA a humanos117-119, es en 1997 que un VIAAP H5N1 fue detectado por primera vez en humanos en Hong Kong120. A partir de enero del 2003 hasta el presente se han venido reportando sucesivas epizootias en aves de corral y aves silvestres que han afectado a por lo menos 60 países121, del mismo modo se han producido zoonosis limitadas en humanos en una cantidad acumulada de 433 casos con una mortalidad de 262 es decir un tasa de letalidad de 60,50 %, afectando 15 países122. En la actualidad se viene observando un incremento de la virulencia de los subtipos H5N1 aislados en casos humanos123. Además se ha podido observar en el caso del VIAA H5N1 su transmisión a gatos124-125 y del VIAA H3N2 a perros126.
Desde 1999 en Hong Kong, han sido aisladas cepas de virus de la gripe H9N2 en niños con enfermedad leve y autolimitada. El virus contiene genes homólogos a los virus H5N1 de 1997. Este virus H9N2 es ahora endémico en poblaciones de pollos y aves vivas en Asia y ha sido aislado en cerdos127-129.
En el 2003, el Centro Nacional de Influenza en los Países Bajos confirmó 83 casos de infección por el virus de influenza H7N7 en trabajadores de granjas de aves de corral y sus familias ocurrido posterior al brote de H7N7 en pollos que comenzó a finales de febrero de 2003130-134. El subtipo H7N3 y H10N7 también han sido reportados135-136.
Con respecto a la influenza porcina es claro que la transmisión a humanos ha y puede ocurrir137,138, un ejemplo de ello es el brote epidémico de 1977 de influenza porcina conocida como A/New Jersey/76 (Hsw1N1) que causo fuertes estragos en población militar joven en el fuerte Dix, New Yersey, EUA. Este virus cocirculó por breve tiempo con el VIH A/Victoria/75 (H3N2)139. Es más desde 1974 la información serológica acumulada demuestra que el riesgo de que a partir de VIAP se produzcan epidemias humanas no debe ser subestimado140. Concordantemente, ese mismo año, en un estudio posmorten en un adolescente granjero de 16 años -que había fallecido de neumonía complicada y tenía como antecedente el padecer de enfermedad de Hodking- se aisló en sus pulmones un subtipo de origen porcino denominado A/MayoClinic/103/74(Hsw1N1) además de anticuerpos positivos para VIAP141. En 1975 un niño de cinco años también procedente de una granja de porcinos presentó síndrome febril y respiratorio de origen desconocido con seroconversión demostrada a VIAP, cinco de los siete miembros de la familia también tenían anticuerpos contra VIAP142. Desde 1973 ya se pronosticaba que un virus de influenza A muy probablemente de origen porcino podría aparecer dentro de un período no muy lejano a finales del siglo pasado143. En general, las infecciones humanas con virus de la influenza porcina, hasta la fecha, han sido diferentes y mucho más leves que las observadas con la gripe aviar A (H5N1)144, y más similares a las infecciones de baja patogenicidad del virus de la gripe aviar145
Para 1976, diversos subtipos de A/H1N1 del linaje del virus de la influenza porcina clásico (VIAPc), también conocido como peste porcina clásica (PPc) circulaban desde que en 1918 tanto porcinos y humanos enfermaron durante la gran pandemia de 1918. Del mismo modo, la aparición de la pandemia H3N2 humana de 1967 coincidió también con la aparición y cocirculación de un nuevo subtipo de VIAPc H3N2146. A lo largo de los siguientes años diversos subtipos de VIAP H3N2 surgieron principalmente como producto de derivas y reordenamientos antigénicos múltiples en los que coparticiparon componentes humanos, porcinos y aviares147-153.
Así, alrededor del año 1998 como producto de estos procesos de deriva y recombinación antigénica asociadas a enzootias se detectó a partir de un triple reordernamiento de un VIAPc con un VIAH contemporáneo H3N2 y un subtipo desconocido de VIAA americano, a un nuevo subtipo de VIAPr denominado H3N2r en Norteamerica 99,144-147,154-156. El VIAP H3N2r subsecuentemente sufrió cambios antigénicos menores y se cree se reordenó con un VIAPc H1N1 originando un nuevo subtipo de VIAP H1N2 paralelo al humano157. Nuevos subtipos de VIAP A/H1N1 también han seguido generándose y circulando. Desde el 2005 al 2009 12 casos humanos infectados con estos virus H3N2r se encuentran debidamente documentados en los EUA158-162
Por otra parte y en concordancia temporal con la reaparición en 1977 del VIAH H1N1, un nuevo VIAP H1N1 asociado en sus ocho porciones genéticamente constitutivas a un virus euroasiático aviar apareció en 1979 en Europa del Este extendiéndose por el continente europeo, sutituyendo al VIAPc H1N1150-163-165.
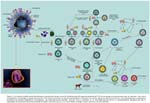
Los VIAPr H3N2r siguieron circulando predominantemente en Norteamérica, mientras que el VIAP H1N1 euroasiatico (H1N1EuAs) lo hicieron en Europa dándose origen a dos subtipos antigénicamente distintas de VIAP tanto en Norteamérica como en Europa156,166 .
El análisis filogenético a partir de genes de hemoaglutinina A del VIAP H3N2 recolectados desde 1970 hasta el 2006 en China han demostrado que estos pueden ser disgregados en tres grandes linajes: aviar, humano y porcino europeo142. Un análisis filogenético reveló que A/Swine/Guangdong/96/06 se deriva directamente del VIH H1N1 de alredor del año 2000, mientras que A/swine/Tianjin/01/04 y A/swine/Henan/01/06 parecen ser descendientes de los VIH H1N1 circulantes en 1980167. Últimamente otros subtipos virales porcinos han hecho su debut en China, Canadá y Estados Unidos, con componentes del VIAPc, VIAH y VIAA. La cocirculación y reordenamiento enzootico entre los VIAPr H3N2r, y el H1N1EuAs ha sido reportada143,168-177. De hecho los genomas de los subtipos VIAP H1N1 muestran una mayor tendencia hacia una evolución divergente que convergente y/o paralela178
En términos generales podemos afirmar que desde la aparición del VIAH H1N1 en 1918, el genoma de los diferentes VI circulantes han tenido patrones congruentes pudiéndose realizar líneas filogenéticas179. Ver Figura 2. El 15 y 17 de abril del 2009 se detectó un nuevo virus de origen porcino capaz de infectar a humanos. Este virus fue denominado tardíamente nuevo virus de la influenza A/ H1N1, que de hecho no es más que un subtipo reordenado que ha pasado ha ser parte del linaje humano4,180. Es este virus el que ha desencadenado el terror pandémico de la influenza una vez más sobre la humanidad, y es en este contexto en donde la realidad y la historia han sido distorsionadas pues si bien el virus tiene cuatro componentes suinos, el mismo muestra un reordenamiento en el que también participaron subtipos virales de VIA y VIH180, por lo que su denominación de virus de la influenza porcina ha sido incorrecta.
Del mismo modo si bien en marzo del 2009 se reportaron en México, Veracruz, comunidad La Gloria, Valle de Perote, próximo a una granja de cerdos en donde se crían anualmente alrededor de un millón de cerdos, brotes de enfermedades respiratorias que culminaban en neumonía y bronconeumonía181, no fue posible detectar una epidemia o de influenza porcina en dicha granja182-183 . Lo cierto es que es sumamente probable que el nuevo virus de influenza A/H1N1 estuviera circulando desde marzo del 2009 tanto en Estados Unidos como en México. De hecho, el 25 de abril del 2009 la Organización Mundial de la Salud (OMS) informa al mundo sobre la epidemia causada por el virus de influenza A H1N1 de origen suino, notificado primero por Estados Unidos en dos niños al sur de California el 28 y 30 de marzo4,184-185 y casi paralelamente por México5,calificándolo de emergencia de salud pública de importancia internacional (ESPII)178 bajo el reglamento sanitario internacional187.
El 27 de abril, el nivel de alerta pandémica se eleva a fase 4 y el 29 de abril a fase 5188, después de encontrar evidencias de transmisión sostenida a nivel de comunidad en al menos dos países de la misma región de OMS189, indicio claro de la inminencia de una pandemia. Finalmente, la OMS decreta la fase 6 o fase pandémica un 11 de junio del 2009 con 46 737 infectados por el virus en todo el mundo y 74 países afectados con 141 fallecidos190. Esta última fase se caracteriza por los criterios que definen la fase 5, acompañados de la aparición de brotes comunitarios en al menos un tercer país de una región distinta, al 29 de mayo del 2009191.
En el Perú este nuevo subtipo humano A H1N1 llegó posiblemente días antes al viernes 15 de mayo del 2009, fecha en que se confirma el primer caso oficialmente por el Ministerio de Salud del Perú (MINSA)192. A la fecha del cierre de este artículo el número de casos confirmados oficialmente en Perú es de 4 104 casos con 29 fallecidos y 2 661casos dados de alta. En el mundo oficialmente 131 países y 18 territorios dependientes han sido afectados con 172 855 y 1 039 fallecidos193.
Modelos computacionales de proyección cuantitativa indican que a medida que la pandemia se ha desarrollado y entrado en una fase sostenida y generalizada, el número oficial de casos confirmados por el Centro de Control de Enfermedades de los Estados Unidos de Norteamérica sólo representan una fracción de los casos reales que ocurren y que pasan como un cuadro gripal o desapercibidos194,195.
RESERVORIO
La evolución de los subtipos de VI-A H1N1 y H3N2 es compleja sugiriéndose un modelo de almacenamiento y preservación ecológica, que permitiría la aparición de nuevos subtipos a partir de reservorios persistentes localizados en zonas tropicales para dirigirse a las zonas Templadas196.
Un análisis antigénico de la hemoaglutinina de aproximadamente 13 000 humanos con gripe A (H3N2) procedentes de los seis continentes durante el 2002-2007 mostró que había una circulación temporal continua entre el Este y el Sudeste de Asia (E-SE) a través de una red de oleadas en toda la región con superposición de epidemias y que de aquí se dispersaron a las regiones templadas a partir de esta red durante cada año, llegando primero a Oceanía, América del Norte y Europa, y más tarde América del Sur. Esto implicaría que las nuevas variantes H3N2 emergen primero E-SE para luego propagarse al resto del mundo197. Muchas de la nuevas variantes H3N2 han sido detectadas primigeniamente en China198-199.
Del mismo modo, Alonso et al muestra como en Brasil las influenzas interpandemicas inician y viajan en ondas desde los centro menos poblados del norte hacia las zonas mas templadas al sur en las grandes ciudades200.
Otro estudio en China con respecto al VIAA H5N1 muestra como la distancia mínima a humedales, el rango de precipitaciones anuales y la presencia de mercados de aves vivos en las proximidades con respecto a la población de aves migratorias son las principales variables que contribuyen a la diseminación de este virus201, del mismo modo se ha observado un incremento de la circulación de VIAAH en una patron de estacionalidad paralelo al reservorio humano durante la estación lluviosa en los tropicos109,,202
Laguna –Torres et al en un importante trabajo en el Perú remarcan de que a pesar de ser el tiempo de medición muy corto en su estudio para establecer metodológicamente tendencias estacionales/clima, se pueden encontrar la presencia de factores ambientales como humedad y temperatura asociados a la aparición de VI-A y VI-B66
Desde hace mucho tiempo se conoce que las aves, especialmente las acuáticas, son reservorios naturales de los VI-A que conforman el linaje aviar, existiendo dos tipos de virus de influenza aviar: la altamente patógena (VIAAAP) y los de baja patogenicidad (VIAABP), contituyendo un reservorio permanente para su perpetuación, debido a que en ellas la infección suele ser asintomática y el VI-A establece un estado de equilibrio evolutivo203-206. Alaska es tal vez el punto de intersección migratoria de aves más importante del mundo que incluyen Asia, Australia, Europa, Norteamérica y América del Sur207. Aquí se han encontrado en aves, fuentes de agua y sedimentos, diversos subtipos de VI aviar tales como H3N6, H3N8, H4N6, H8N4 y H12N5 y hemaglutininas como la H11 y H12208.
El VIAA infecta con avidez las células del tracto intestinal de las aves, en especial patos, gansos y cisnes, por lo que el virus es excretado en altas concentraciones en las deyecciones de estas aves. La transmisión de los VI-A entre aves ocurre primariamente por la vía fecal-oral. Desde que estas aves cumplen ciclos migratorios anuales con destinos predeterminados que incluyen escalas para descanso y alimentación, principalmente en cuerpos de agua, humedales y bosques209-210, la destrucción de estos, debido al aumento de las actividades humanas, especialmente las prácticas de utilización de la tierra, esta causando que las aves migratorias se vean obligadas a buscar refugio y alimentos en otros lugares tales como explotaciones agrícolas. Esto puede dar lugar a aumentos de contacto entre los salvajes y las aves domésticas, lo que facilita la transmisión del virus a las poblaciones de aves domésticas.
Debido a que los VIAA son depositados en las rutas de migración de las aves, diversos mamíferos pueden infectarse, por el contacto directo con las deyecciones aviares depositadas en sus trayectos de migratorios especialmente en fuentes hídricas como lagos, humedales pantanales, entre otros, por el consumo directo de aves o huevos infectados por depredadores naturales.
En el humano el proceso de infección se da esencialmente por el contacto directo con estas aves, aereolización y/o fomites contaminados con partículas virales83,137,197,211, iniciando una circulación endémica en el nuevo hospedero, independiente al reservorio madre212-217. La supervivencia de los VIAA fuera de sus anfitriones aviares depende de los distintos subtipos virales, el pH, la salinidad, temperatura y el tipo de medio en el cual el virus queda dispersado218-220.
Se sabe por ejemplo que después de ser excretados VIAA estos pueden permanecer viables en fuentes de agua dulce, sedimentos y excretas humedas por períodos prolongados de tiempo221,222. En términos generales estos virus pueden sobrevivir un rango de 4 a 6 hasta más de 30 dias en temperaturas que oscilan entre 37 ºC y 0 ºC y su persitencia es inversamente proporcional al pH, temperatura y salinidad del medio acuoso207-223.
Aunque el rol de las fuentes abióticas en la perpetuación y transmisión de los VIAA es todavía poco entendido, se estima que son un reservorio para el contagio de futuras aves224-227,además de una fuente para múltiples y constantes reordenamientos228
Del mismo modo los VI- A y B del linaje humano puede permanecer viables en superficies porosas desde horas a días, dependiendo de factores ambientales y humanos tales como cobertura mucosa protectora, temperatura, humedad relativa e higiene229-231
En la costa de la Región de Lima en Perú se han detectado varios subtipos de VIABP (H3N8, H4N5, H10N9, y H13N2)232, lo que demuestra el rol dispersos de las aves acuáticas en el sistema ecodinámico de estos virus aviares.
Los porcinos, por otro lado, tienen un importante y complejo rol como reservorios de los VI-A. Tanto los VI-A del subtipo aviar como los del subtipo humano pueden infectar y causar enfermedad en los cerdos.
Una coinfección en cerdos con dos o más virus de diferente orígen o linajes pueden dar lugar a la presentación de un nuevo virus por reordenamiento con ocho segmentos de ARN de su genoma totalmente distintos; los que pueden originar nuevas epidemias en las manadas de cerdos y tiene el potencial de iniciar una pandemia humana156,. Asimismo, los porcinos pueden servir de reservorios de cepas humanas antigénicamente viejas y preservarlas en el tiempo160,233-,234. De hecho una redistribución de VIA y VIH en cerdos en 1979, condujo a un brote en los niños en los Países Bajos en 1993235.
Los VI-A también afectan a los caballos236, en la actualidad hay dos diferentes subtipos de VI-A propios de los equinos, el H3N8 y el H7N7, el primero fue aislado por primera vez en Praga y se ha mantenido estable estimándose que sería uno de los linajes más antiguos de todos los virus de la influenza en mamíferos. Estudios serológicos han demostrado que estos virus infectan a los humanos83,237. Así mismo, en varios tipos de mustélidos, en especial los hurones, también se ha comprobado que estos son altamente sensibles a los VI-A que atacan a los humanos238-241.
En el caso de los humanos, la circulación de VIAH se consideró limitada hasta hace poco a los subtipos H1N1, H1N2, H2N2 y H3N2242. Sin embargo, en los años recientes se ha comprobado que otros subtipos del VIAA, como el H5N1, H7N7, H9N2 y H10N7, propios del reservorio aviar han infectando al hombre243,244
TRANSMISIBILIDAD
La transmisión de virus de la gripe en los seres humanos se produce por contacto directo con el individuo infectado en ambientes cerrados o a una distancia aproximada de 1 metro245,246 por inhalación-contacto de gotitas infecciosas o gotitas de Flügge de 5 μm o menor tamaño en conjuntiva y mucosa respiratoria a partir del estornudo o tos del individuo infectado. Estas mismas gotitas de Flügge pueden permanecer suspendidas en el aire y viajar distancias importantes247,248. También se han descrito núcleos de gotas mayores a los 10μm que por su peso se precipitarían sobre fomites generando su contaminación con las superficies libre y que a su vez servirían de vía de incoculación indirecta del virus en la parte superior de las vías respiratorias o de la mucosa conjuntival. La importancia relativa de estas rutas es todavía materia de debate249-253.
Los niños juegan un papel especial en la transmsibilidad de los VI. La carga de enfermedad en esta población es relevante. La tasas de infección en los niños preescolares y escolares puede superar el 30 % en las influenzas interpandemicas246 ademas estos son agentes de transmisión del virus por más días que los adultos. El comportamiento de nuevos virus pandémicos en esta y otras poblaciones es desconocido254.
Se sugiere que en las primeras etapas de un brote epidémico o pandémico, las mutaciones que ocurren durante las sucesivas replicaciones del virus no necesariamente permanecen en la cepa circulante de manera que la circulación de mutantes no es homogénea durante algún tiempo254.
Los VIAH pueden sobrevivir durante 24-48 horas en superficies no porosas y 8-12 horas en tela, papel y tejidos, tambien se ha demostrado que es transferible a partir de superficies no porosas a las manos durante 24 horas y de tejido a mano por 15 minuto. La sobrevida del virus en la mano no fue mayor a 5 minutos luego de la transferencia viral255. Así por ejemplo virus de la influenza han sido detectado como viables por varios días en la superficie de los billetes221.
El transporte comercial aéreo, si bien contribuye a la propagación de las epidemias de influenza, la magnitud de su impacto no está claro en comparación con otros factores256-258 e inclusive algunos estudios indican que su impacto sería menor259-260.
Algunos estudios muestran que las fuerzas ambientales tales como la temperatura y humedad desempeñan un papel muy importante en la direccionalidad y transmisibilidad de la expansión de la influenza interpandemica en relación a los factores de densidad poblacional y viajes192-194 H1N2, H2N2 y H3N2242. Sin embargo, en los años recientes se ha comprobado que otros subtipos del VIAA, como el H5N1, H7N7, H9N2 y H10N7, propios del reservorio aviar han infectando al hombre243,244
TRANSMISIBILIDAD
La transmisión de virus de la gripe en los seres humanos se produce por contacto directo con el individuo infectado en ambientes cerrados o a una distancia aproximada de 1 metro245,246 por inhalación-contacto de gotitas infecciosas o gotitas de Flügge de 5 μm o menor tamaño en conjuntiva y mucosa respiratoria a partir del estornudo o tos del individuo infectado. Estas mismas gotitas de Flügge pueden permanecer suspendidas en el aire y viajar distancias importantes247,248. También se han descrito núcleos de gotas mayores a los 10μm que por su peso se precipitarían sobre fomites generando su contaminación con las superficies libre y que a su vez servirían de via de incoculacion indirecta del virus en la parte superior de las vías respiratorias o de la mucosa conjuntival. La importancia relativa de estas rutas es todavía materia de debate249-253.
Los niños juegan un papel especial en la transmsibilidad de los VI. La carga de enfermedad en esta población es relevante. La tasas de infección en los niños preescolares y escolares puede superar el 30 % en las influenzas interpandemicas246 ademas estos son agentes de transmisión del virus por mas dias que los adultos. El comportamiento de nuevos virus pandemicos en esta y otras poblaciones es desconocido254.
Se sugiere que en las primeras etapas de un brote epidemico o pandemia, las mutaciones que ocurren durante las sucesivas replicaciones del virus no necesariamente permanecen en la cepa circulante de manera que la circulación de mutantes no es homogénea durante algún tiempo254.
Los VIAH pueden sobrevivir durante 24-48 horas en superficies no porosas y 8-12 horas en tela, papel y tejidos, tambien se ha demostrado que es transferible a partir de superficies no porosas a las manos durante 24 horas y de tejido a mano por 15 minuto. La sobrevida del virus en la mano no fue mayor a 5 minutos luego de la transferencia viral255. Asi por ejemplo virus de la Influenza han sido detectado como viables por varios días en la superficie de los billetes221.
El transporte comercial aéreo, si bien contribuye a la propagación de las epidemias de influenza, la magnitud de su impacto no está claro en comparación con otros factores256-258 e inclusive algunos estudios indican que su impacto seria menor259-260
Algunos estudios muestran que las fuerzas ambientales tales como la temperatura y humedad desempeñan un papel muy importante en la direccionalidad y transmisibilidad de la expansión de la influenza interpandemica en relación a los factores de densidad poblacional y viajes192-194
Debido a que los VI-A son genéticamente variables, su tasa de casos secundarios a partir de uno primario o transmisibilidad (RO) es dificil de predecir. Con un nuevo VI-A el R0 comenzará bajo, probablemente un poco por encima de 1 y, a continuación, con cada generación aumentará a medida que el virus se adapta a la población humana. La rapidez con que los VI-A pueden mejorar su transmisibilidad depende desgraciadamente en gran parte del carácter impredecible del mismo virus262.
En centros de atención de salud, estudios de evaluación de las medidas para reducir la propagación de virus respiratorios sugieren que el uso de máscaras podría reducir la transmisión de la gripe263. En la comunidad, sin embargo, los beneficios del uso de máscaras no se ha establecido, especialmente en zonas abiertas, en contraposición a los espacios cerrados con contacto estrecho con una persona con síntomas gripales237,264
Para los fines de esta revisión tomaremos arbitrariamente ciertos conceptos de importancia estandarizados a la fecha para la nueva influenza A/H1N1 y que es necesario tener en cuenta en este acápite de transmisibilidad1:
Contacto cercano
Aquel que ha cuidado, convivido o tenido contacto directo con secreciones respiratorias o secreciones de un caso sospechoso, probable o confirmado de la nueva influenza A H1N1265.
Contacto cercano tambien ha sido definido preventiva y provisionalmente como la interacción a menos de 1 metro con una persona que es un caso confirmado o probable de infección por el nuevo subtipo de influenza A (H1N1), durante el periodo infeccioso (desde un día antes, hasta 7 días después del inicio de los síntomas)266. Recomendación provisional para el tratamiento con antivirales de los pacientes confirmados o sospechosos de estar infectados por el nuevo subtipo del virus influenza A(H1N1). 30 de abril 2009. Se ha sugerido un distanciamiento Social mínimo de 2 metros267
Caso sospechoso
A quien cumpla alguna de las siguientes condiciones:
Persona con fiebre mayor de 38 °C acompañada de al menos uno de los siguientes signos o síntomas tales como rinorrea, tos, o dolor de garganta y que, estuvo en los 7 días previos al inicio de su enfermedad: en una zona con casos confirmados de infección por influenza A(H1N1), o en contacto cercano con un caso confirmado de infección con el virus Influenza A(H1N1) 265.
Caso probable
Cualquier caso sospechoso con resultado de prueba positiva para influenza A, pero no subtipificado por los reactivos comúnmente utilizados para detectar la infección por el virus de la influenza estacional, o persona que murió a causa de una infección respiratoria aguda inexplicada y con nexo epidemiológico con un caso probable o un caso confirmado265.
Caso confirmado
Persona con una prueba de laboratorio confirmatoria de infección con el virus influenza A(H1N1) que permita su subtipificación confirmatoria y que sea realizada en los establecimientos autorizados por el Ministerio de Salud265,268. En este caso el Instituto Nacional de Salud (INS) y el U.S. Army Medical Research Institute of Infectious Diseases Naval Military Research of Infectious
Diseases de la Armada de los Estados Unidos. Cualquier laboratorio que tenga alguna prueba para confirmar la Nueva Influenza AH1N1 debe someterla al INS para su validación y la correspondiente autorización193.
Es importante observar que en el contexto actual de epidemiológico de la pandemia por A H1N1 las definiciones de caso probable y caso sospechoso son eliminadas268.
PREVENCIÓN Y ESCENARIOS EPIDEMIOLÓGICOS
La Influenza afecta a la humanidad en diferentes escenarios epidemiológicos, se presenta como una enfermedad endémica estacional, como enfermedad zoonótica esporádica (influenza aviar o porcina) como epidemias locales o regionales y ocasionalmente como grandes pandemias que afectan a gran parte de la población del mundo. Para la prevención y el control de la Influenza pueden ser tomadas medidas llamadas farmacológicas entre las que se encuentran las vacunas y los antivirales y las llamadas medidas no farmacológicas las que considera medidas de distanciamiento social, la protección personal el aislamiento y la cuarentena.
Influenza estacional:
Frente a la influenza estacional la principal medida es la vacunación, cada año las vacunas son actualizadas para las cepas de virus de influenza que hasta la fecha han sido conocidas. Cada país ha tomado diferentes medidas para implementar la vacunación para la influenza estacional algunos países recomienda la vacunación para toda la población, otros vacunan a los considerados grupos de riesgo o grupos de mayor vulnerabilidad como los menores de 2 años y los ancianos. En nuestro país en el presente año 2009 se ha iniciado la vacunación gratuita para la influenza estacional considerando a los niños menores de 1 año (a los 7 y 8 meses), a los mayores de 60 años, a los trabajadores de salud y a la población en riesgo269.
La vacuna anti-influenza es una vacuna trivalente, compuesta por tres virus inactivados, fraccionados y desprovistos de lípidos reactogénicos, lo que la hace una vacuna de buena calidad inmunógena y con muy pocos efectos secundarios a su administración. La composición recomendada por la OMS para la vacuna del hemisferio sur de este año es con 3 cepas de influenza: Esta vacuna es recomendada para ser usada en la estación 2009 (invierno hemisferio sur) debe contener las siguientes cepas:
-
A/Brisbane/59/2007 (H1N1) –virus similares,
-
A/Brisbane/10/2007 (H3N2) –virus similares,
-
B/Florida/4/2006 – virus similares
Indicación del uso de vacuna influenza
-
Adultos mayores de 60 años
-
Mujeres en segundo y tercer trimestre de embarazo
-
Niños entre 6 meses y 2 años
-
Personal de los establecimientos de salud y cuidadores de hogares de niños y ancianos
-
Personas, adulto y niño, en riesgo de complicaciones por influenza: Enfermedades pulmonares crónicas, cardiopatías congénitas o adquiridas, terapia crónica con corticoides, terapia crónica con aspirina, pacientes con trasplantes de órganos sólidos o médula ósea, pacientes inmunosuprimidos con terapia inmunosupresora actual. En estos pacientes la respuesta humoral está reducida por lo que se recomienda vacunar también a todos los contactos intradomiciliarios de estos pacientes de riesgo. (Padres, hermanos, cuidadores, personal de salud)
Influenza aviar y porcina
Se han descrito casos humanos por diferentes virus de influenza aviar, que fueron contagiados principalmente por manipular o estar en contacto con aves domésticas, la vigilancia y control de brotes del virus aviar AH5N1 sigue siendo un importante candidato a convertirse en un virus pandémico. Frente a los brotes epizoóticos por virus aviares son eliminados las aves domésticas en un área extensa. El control debe ser dirigido hacia la detección oportuna de los brotes epizoóticos y a la protección de las personas que trabajan en contacto con las aves. La crianza de aves de manera informal, llamada crianza de traspatio es otro de los aspectos que deben ser controlados por medidas regulatorias como un mecanismo de prevención. En el Perú la Vigilancia y control de la influenza en aves, está a cargo del Servicio Nacional de Salud Animal SENASA, organismo del Ministerio de Agricultura. Recientemente ha sido descrito la presencia de virus aviares en aves silvestres en el Perú270. En nuestro país nunca se ha hecho vigilancia de virus de influenza porcina por considerarse una enfermedad exótica.
Brotes y epidemias locales o regionales
Muy pocos brotes o epidemias locales de influenza han sido descritos en el Perú, no se conoce exactamente cuál es el porcentaje atribuible a la influenza en la morbilidad y mortalidad por enfermedades respiratorias que resenta el Perú y que continua siendo la principal causa de muerte para toda la población y en especial para los niños pequeños y los ancianos271. La vacunación para la influenza estacional y la vigilancia epidemiológica son las principales medidas para la prevención y detección precoz de brotes para su control.
Algunos brotes han sido descritos en población indígena amazónica con consecuencias severas como los ocurridos en la población Nanti de la selva del Cusco. Frente a estos brotes fue necesario desplazar brigadas de emergencia en helicópteros para poder atender a esta población que vive en aislamiento voluntario y en zonas de difícil acceso272. Se requiere de una política especial para la protección de las poblaciones indígenas en quienes las cepas de los virus de influenza se comporta de manera epidémica
Influenza pandémica
Las actividades de prevención y control frente a la influenza pandémica requieren que los países planifiquen con anterioridad todas las acciones pertinentes y que una vez que se inicia una nueva pandemia requiere de gran capacidad de organización y conducción de las medidas de Salud Pública. La Organización Mundial de la Salud desde hace varios años ha exhortado a los países a que tengan un Plan de Preparación y Respuesta frente a una eventual Pandemia de Influenza, ahora que el mundo vive el inicio de la pandemia de influenza por el virus AH1N1 los países que así lo hicieron están cosechando las ventajas de un trabajo planificado en salud pública.
Los planes deben de considerar las diferentes actividades que deberán realizarse durante los periodos y fases de la evolución de la generación de la pandemia:
Período interpandémico
-
Fase 1: Durante esta fase no se ha detectado la circulación de un nuevo subtipo del virus de influenza en humanos. En animales puede estar presente un subtipo de virus de influenza que ha ocasionado infección en humanos o un nuevo virus que se considera tiene bajo riesgo de producir enfermedad en humanos.
-
Fase 2: No se han detectado nuevos subtipos del virus de influenza en humanos. Sin embargo, un subtipo del virus de influenza animal identificado es considerado una amenaza de producir enfermedad humana.
Período de alerta de pandemia
-
Fase 3: Se detectan casos de infección humana por un nuevo subtipo, sin contagio de persona a persona, o algunos casos aislados de contagio de persona a persona en contacto cercano.
-
Fase 4: Se detecta uno o varios grupos pequeños donde se ha producido transmisión limitada de persona a persona y con una distribución geográfica muy localizada, lo cual indica que el virus aun no está bien adaptado a los seres humanos.
-
Fase 5: Se detecta uno o varios grupos más grandes pero la diseminación de persona a persona sigue siendo localizada, lo que sugiere que el virus mejora progresivamente su adaptación a los seres humanos, pero tal vez no es completamente transmisible aún (riesgo de pandemia considerable).
Período pandémico
-
Fase 6: Fase pandémica: Durante esta fase, la transmisión esta incrementada y permanente en la población general. La OMS declara esta fase cuando se demuestra transmisión sostenida en al menos 2 continentes (Dos regiones diferentes de la OMS)
Durante los 2 primeros periodos es muy importante el fortalecimiento de las capacidades del país en el desarrollo de infraestructura sanitaria, capacitación, organización, funcionabilidad y equipamiento para la implementación de la respuesta en el caso de una pandemia de influenza. En el Perú desde el año 2005 contamos con un Plan Nacional de Preparación y Respuesta frente a la pandemia de Influenza273 y un red nacional de epidemiología comandada por la Dirección General de Epidemiología desde fines de los años 80, que mantienen la vigilancia epidemiológica y juega un rol sumamente importante en el control epidemiológico.
Los planes de preparación y respuesta no solo deben ser Planes Nacionales o Regionales, cada Institución Pública o privada, cada establecimiento de salud y hasta cada familia debe de tener su propio plan y estar preparado para actuar durante la pandemia. La OMS, la OPS, los CDC de los estados unidos desde hace algunos años y otras agencias últimamente has proveído de propuestas, planes modelo, protocolos genéricos, listas de chequeo y otras herramientas para facilitar el proceso de planificación274,275
Las fases 4 y 5 son muy breves y son una corta oportunidad para tratar de realizar una contención del foco de la epidemia, y se había considerado la posibilidad de realizar un bloqueo farmacológico utilizando masivamente antivirales en la población alrededor de los primeros casos276, en esta pandemia del 2009 esta contención no fue posible y rápidamente se disemino desde su foco inicial, lo que motivo que la OMS declaré la fase 6. El 11 de junio de 2009, la Dra. Margaret Chang Directora de la Organización Mundial de la Salud elevó a fase 6277.
..A finales de abril la OMS anunció la emergencia de un novedoso virus de la gripe A. Se trata de una cepa de H1N1 que no había circulado anteriormente en la especie humana, un virus completamente nuevo. El virus es contagioso y se propaga fácilmente entre las personas, y de un país a otro. A día de hoy se han notificado casi 30 000 casos confirmados en 74 países. .Sobre la base de los datos disponibles, y de la evaluación de los datos por esos expertos, cabe concluir que se cumplen los criterios científicos que definen las pandemias de gripe. Por consiguiente, he decidido elevar el nivel de alerta de pandemia de gripe de la fase 5 a la fase 6. El mundo se encuentra ahora en el inicio de la pandemia de gripe de 2009
Medidas farmacológicas
Generalmente frente a un nuevo virus pandémico no se dispone de una vacuna, aunque la ciencia y la tecnología están acortando el tiempo que se requiere, aun el tiempo que demora la producción de una vacuna especifica es por lo menos de 6 meses y cuando se disponga probablemente ya se habrán infectado una gran cantidad de personas. Respecto al uso de fármacos antivirales, los que se cuentan en la actualidad pertenecen a dos grupos, los adamantanes y los inhibidores de la neuroaminidasa, los antivirales pueden ser utilizados para el tratamiento de los pacientes para reducir la gravedad y la letalidad de la enfermedad y también como quimioprofilaxis, para prevenir la infección en quienes tienen un alto riesgo de exposición, como el personal médico y de enfermería que atenderá a los pacientes, las fuerzas públicas que mantendrán la seguridad y el orden como la policía los bomberos etc.
Las posibilidades del uso de los antivirales dependerá si la cepa del virus es sensible o resistente y de la disponibilidad de ellos; tanto el virus de la influenza aviar AH5N1 y el virus AH1N1 de la pandemia actual han demostrado ser resistentes a los adamantanes y sensibles a los inhibidores de la neuroaminidasa, de estos últimos son 2 los que se disponen actualmente, el oseltamivir y el zanamivir. Frente a la actual pandemia la mayoría de los países siguiendo las recomendaciones de OPS/OMS no están utilizando los antivirales como quimioprofilaxis y han dispuesto que el oseltamivir sea solo para el tratamiento de pacientes, reservando su uso para los pacientes con un cuadro clínico severo y para los pacientes que presentan alguna condición de riesgo o comorbilidad que les incrementa la probabilidad de hacer cuadros clínicos mas graves, como es el caso de las gestantes, los niños menores de 5 años, los mayores de 65 años, los que presentan alguna inmunodeficiencia como los pacientes infectados con el VIH, los diabéticos y en general los que presenten alguna enfermedad que reduce su capacidad respiratoria, cardio-vascular o metabólica, como los que padecen de enfermedad pulmonar obstructiva (EPOC), Insuficiencia cardiaca, Insuficiencia renal crónica etc.
Restricción de viajes internacionales y tamizaje en aeropuertos y otros puntos de entrada al país.
La OMS no recomienda ninguna medida que se contraponga al Reglamento Sanitario Internacional278. Durante la Fase 4 o la Fase 5, los países pueden recomendar a sus habitantes que eviten los viajes no esenciales a países donde se ha demostrado la actividad epidémica pero no debe suspenderse ni el comercio ni el tránsito de personas. El tamizaje de febriles con scanner térmico o la búsqueda de personas con síntomas gripales que ingresan al país no ha demostrado que tengan efectividad alguna para detener el ingreso de la influenza a un país. La experiencia de varios países durante el SARS en el año 2003 demostró la ineficiencia de este tipo de medidas; por el contrario consumen recursos humanos y financieros muy importantes los que se agotan pronto279. Lo que sí es recomendable es la información que pueda entregarse a os viajeros indicando como reconocer la enfermedad y los lugares donde puede acudir buscando atención si la necesita o teléfonos donde pueda llamar.
Medidas no farmacológicas:
Los objetivos de las medidas no farmacológicas para mitigar una epidemia de influenza en la comunidad son:
-
Retrasar el crecimiento exponencial de los nuevos casos y entonces desviar a la curva epidémica a la derecha, buscando ganar tiempo para una mejor organización de la respuesta y si es posible para la fabricación y distribución de una vacuna para la cepa pandémica.
-
Reducir el pico máximo de la epidemia aplanar la curva epidémica, y así evitar el colapso de los servicios de salud por una sobresaturación de la demanda de atención.
-
Disminuir la cantidad total de casos incidentes, buscando reducir la cantidad total de personas que enferman o mueren por la epidemia en la población, buscando reducir la curva epidémica.
Estos objetivos pueden lograrse mediante la implementación de intervenciones no farmacológicas (INF) y deben tener la única meta de salvar vidas humanas. Las INF disminuyen el contacto entre personas enfermas y sanas, disminuyendo entonces la posibilidad del contagio, las personas infectadas y entonces se reducirá también la necesidad de atención médica en los servicios de salud y se reducirá el impacto económico y social263,280.
Frente a la pandemia de influenza hemos escuchamos no solo a políticos sino incluso a médicos decir cosas como es imposible detener el avance de la epidemia y solo nos queda atender a los pacientes. En las siguientes líneas tratamos de sostener que existen evidencias que nos alientan a realizar todos los esfuerzos por mitigar el impacto de esta pandemia de influenza.
Para poder entender el sustento de las medidas no farmacológicas que deben de tomarse frente a una pandemia, analicemos los factores que intervienen en la generación de una epidemia. Las epidemias se reproducen, cada caso de un afectado puede contagiar a uno o a más personas, y así se van extendiendo, si cada persona infectada contagia a uno o a mas, entonces las epidemias crecerán, si se contagia cada vez a menos entonces las epidemias se mitigaran. El número reproductivo básico, R0, es el número promedio de infecciones nuevas que una persona infectada provocará en promedio durante el curso de su infección en una población completamente susceptible en la ausencia de intervenciones281-283. Ver Figura 3.
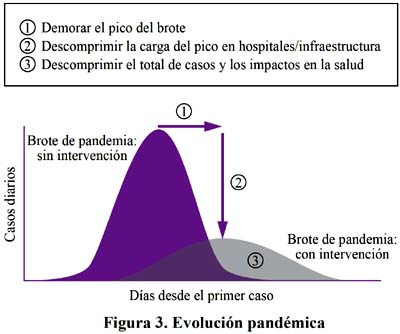
Si R0 es igual a 1 esto quiere decir que cada caso contagiara a una persona más, si R0 es mayor que 1 la epidemia crecerá, si R0 es igual a 2 la epidemia en cada periodo se duplicara y crecerá rápidamente, si R0 es menor que 1 entonces la epidemia se irá reduciendo.
El R0 proporciona una medida de la transmisibilidad del agente infeccioso. El R0 puede ser modificado por cambios en el agente patógeno, en el hospederos o en las redes sociales que facilitan los contactos donde se da a transmisión, por lo tanto intervenciones en alguno de los componentes del Ro producirán cambios en la curva epidémica269,270,271.
El primer componente del Ro es la Tasa de Contacto Infeccioso, si una persona está infectada ¿Qué posibilidad tiene de contagiar a otra?, esto dependerá de con cuantas personas estará en un contacto estrechos durante el periodo que es contagioso alrededor de 7 días y probablemente más de 10 días en el caso de los niños; el segundo factor es la eficiencia de la transmisión, es decir ¿A cuántos de los expuestos como contactos contagiara?, este factor depende principalmente del virus; y el tercer factor Duración de la infecciosidad, corresponde al tiempo en que la persona infectada permanece como fuente de infección284. Ver Figura 4 y 5.
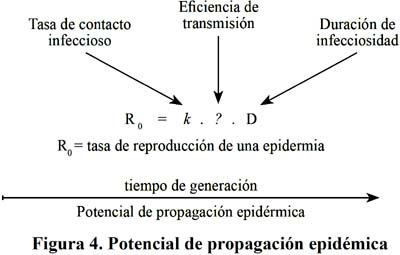
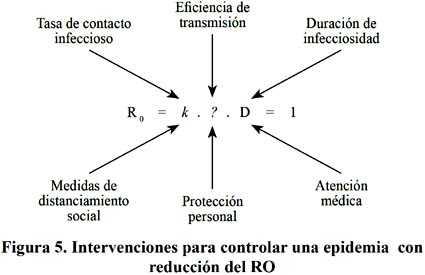
La reducción de la Tasa de Contacto Infeccioso, se logrará si se reduce drásticamente el número de personas con la que se pone en contacto un primer caso infectado, este es el principal sustento por ejemplo de las suspensión de las clases escolares; el segundo componente, Eficiencia de la transmisión se reduce con las medidas de protección personal, como el uso de mascarillas o respiradores cuando una persona se encuentra muy cerca de personas infectadas, como es el caso de los trabajadores de salud que atienden los pacientes; y el tercer factor Duración de la Infecciosidad dependerá de que tan rápido es detectado una persona infectada reciba atención medica, se le administren antivirales y se observen las medidas de aislamiento domiciliario o en el establecimiento de salud según sea el caso272. Ver Figura 6.
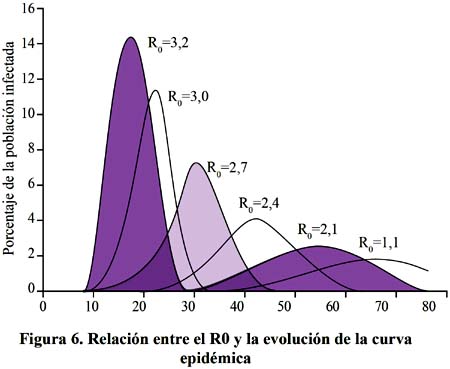
Las acciones para mitigación de una pandemia deben de ser implementadas de manera temprana, selectiva y estratificada y considerando múltiples medidas no farmacológicas. Las medidas deben de ser implementadas lo antes posible antes del crecimiento abrupto de la pandemia. Las principales medidas no farmacológicas son las siguientes
-
Educación sanitaria, dirigida a fomentar el autocuidado, higiene de la tos y lavado frecuente de manos. También brindar información de cuando y donde acudir si se presenta síntomas gripales.
-
Aislamiento de todas las personas con influenza confirmada o que se sospeche clínicamente de la misma. El aislamiento puede realizarse en el hogar o en un establecimiento de salud, dependiendo de la gravedad de la enfermedad del individuo.
-
Cuarentena domiciliaria voluntaria de todos los familiares de los casos de influenza que se han expuesto y que podrían estar incubando la enfermedad, probablemente los que requieran necesariamente salir a la comunidad por razones de trabajo los seguirán haciendo, quienes al presentar los primeros síntomas deberán de buscar atención médica y observar aislamiento.
-
El cierre de escuelas (incluyendo escuelas públicas y privadas así como academias y universidades), así como la suspensión de todas las actividades relacionadas con la escuela como fiestas, eventos deportivos, excursiones etc. El cierre de las escuelas no debe verse solo como una medida de protección a los escolares, sino como una medida de Salud Pública que busca cortar la transmisión hacia las personas con mayor vulnerabilidad para enfermar gravemente y morir ya que son los niños pequeños que diseminan la enfermedad a los adultos mayores, gestantes y otras personas vulnerables. Es en las escuelas donde se da el mayor contacto social entre personas285,286, existen evidencias históricas de las pandemias anteriores y modelamientos matemáticos actuales287-292 que fundamentan el cierre de las escuelas.
-
La medidas de distanciamiento social, buscan reducir el contacto entre adultos en la comunidad y en el lugar de trabajo, incluyendo, por ejemplo, la cancelación de grandes reuniones públicas y modificación de los entornos y horarios laborales para disminuir la densidad social y mantener un lugar de trabajo saludable en la mayor medida posible sin interrumpir los servicios esenciales. No existe una clara evidencia del impacto del cierre de locales públicos como los restaurantes, cines, estadios y otros. La experiencia reciente de las drásticas medidas tomadas en México están siendo evaluadas. Los centros laborales que puedan permitir que los trabajadores realicen tareas desde su casa deben hacerlo, o dividir turnos de trabajo para disminuir el hacinamiento y dar facilidades para la higiene personal293.
-
Licencia laborales, debe de darse las facilidades de descanso médico por razones laborales a las personas que tengan un cuadro gripal por una semana después del inicio de la enfermedad independientemente de la gravedad del caso, también debe considerarse una licencia especial paraquienes tienen mayor riesgo de enfermar gravemente como las gestantes y los que tienen algún tipo de inmunodepresión. Las gestantes que por razones laborales tienen contacto con niños como las maestras deberán tener una licencia de mayor duración, independiente del tiempo de suspensión de las clases escolares. En la presente pandemia las embarazadas han tenido un riesgo elevado de formas graves habiendo reportado muertes en varios países294.
-
El rol de la Comunicación Social es sumamente importante, las autoridades sanitarias en el nivel nacional y local deben de mantener permanente información a través de los medios de comunicación masiva de manera oportuna y transparente, deberán de prepararse comunicados oficiales o notas de prensa lo más frecuente posible sobre el número de casos sin dejar de dar las recomendaciones adecuadas de manera permanente. La estrategia de comunicación debe ser preparada por especialistas en técnicas de comunicación y con sustentos epidemiológicos. La falta de una comunicación oportuna o el ocultamiento de información generará que los medios de comunicación especulen y puedan generar confusión a la población haciendo perder la credibilidad en los servicios de salud que muchas veces tienen consecuencias serias en el manejo de la emergencia.
Podrían existir algunas controversias sobre algunas de las medidas no farmacológicas dado que las recomendaciones se basan más en la experiencia histórica y en modelos matemáticos que en ensayos de campo controlados, sin embargo todas ellas tienen plausibilidad biológica y epidemiológica y su implementación requiere de una evaluación juiciosa en cada lugar y en cada situación donde se apliquen y nunca poner razones económicas o estudiantiles por encima del riesgo de la salud y la vida de las personas.
En los Estados Unidos se ha propuesto un índice de gravedad de la pandemia basado en la tasa de letalidad (porcentaje de víctimas fatales) y una guía que establece las intervenciones que se recomiendan según el nivel de severidad de la pandemia263. Sin embargo el esperar que la pandemia llegue a tal índice de gravedad podría hacer que las medidas se tomen tardíamente, por lo que un equipo de epidemiólogos, infecto logos y otros profesionales con experiencia en el control de epidemias deben asesorar a las autoridades sanitarias y orientar las decisiones que se toman en el curso de la pandemia. En nuestro país además de los equipos técnicos del MINSA se cuenta con la asesoría de la OPS y del Comité de Control de Enfermedades Transmisibles del Consejo Nacional de Salud
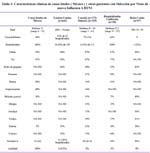
La letalidad promedio observada en el mundo es de 0,5%, en nuestro país la letalidad al 10 de julio de 2009, es de 0,3% por lo que el nivel de Gravedad se encontraría entre 2 y 3 a la fecha295. Ver Tabla 1.

Todas esas estrategias basadas en la comunidad se deben realizarse de manera conjunta y muy tempranamente cuando empieza la actividad epidémica. No debe esperarse a que la epidemia se esté expandiendo, por ejemplo, si se espera que el ausentismo escolar o laboral sea manifiesto, probablemente ya será muy tarde para tomar alguna medida de prevención.
Debe considerarse la situación especial de las poblaciones cerradas como las cárceles, los orfanatos, los cuarteles. En las pandemias anteriores y durante la pandemia actual se han descrito brotes en estas poblaciones. En primer lugar debe de darse todas las facilidades para la higiene en especial del lavado de manos con agua y jabón, si se decide limitarse el ingreso de visitas a las cárceles debe de hacerse en coordinación con las autoridades y los mismos internos explicando que es una medida transitoria para prevenir la transmisión de la enfermedad y facilitar que sigan recibiendo alimentos o comunicarse de otra manera con sus familiares, teléfono o cartas en el mismo centro penitenciario. En todos los casos deberá disponerse de un lugar para aislamiento de posibles pacientes y desarrollar una vigilancia estricta de síntomas.
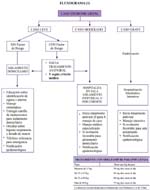
La capacidad de un país para implementar las medidas de prevención y dada ya la situación de actividad pandemica, la capacidad de contención y mitigación, dependerá principalmente de la capacidad social de respuesta en Salud Publica que el país y los niveles subnacionales hayan construidos. Una organización en salud pública si no cuenta con suficientes recursos humanos, principalmente epidemiólogos y salubristas organizados en una estructura adecuada y sostenidos en una carrera que permita que los que adquieren mayor experiencia y conocimientos sean los que toman las decisiones estratégicas, difícilmente podrá poner en práctica las intervenciones adecuadas y podrá ser influenciado a poner en práctica decisiones políticas sin fundamento técnico. Ver Figura 7.

LOS VIRUS: VIRULENCIA Y FISIOPATOLOGÍA
Tres son los grades tipos de virus ARN negativos del genero Ortomixovirus que causan influenza. Estos son conocidos como virus de la influenza A, B y C1. Poseen una morfología pleomórfica, con viriones generalmente esféricos u ovoides, con un diámetro de 80 a 200 nm234. El genoma viral se encuentra comprimido en ocho segmentos fragmentados de ARN que codifican hasta 10 tipos de proteínas conocidas, ocho de ellas estructurales296-298. El VI-C es una excepción ya que solo son siete fragmentos299-300. Cuentan con una envoltura bilipídica derivada de la membrana de la célula hospedera en la que se insertan dos glicoproteínas o antígenos principales de superficie externa denominados hemaglutininas (HA) y neuraminidasas (NA). Sin embargo, el VI-C carece de NA301.
El reservorio genético de VIA presente en las aves acuáticas proporciona una inmensa fuente de diversidad genetica para que otros hospederos como los porcinos puedan interaccionar a través de sus cadenas de receptores sacáridos con residuos de ácido siálico (RSAS) -generalmente vinculados a la galactosa- una familia de monosacaridos asociadas a glicoproteínas de superficie celular y glicolípidos en el glicocalix302. Esto es el inicio en la generación de múltiples caminos aletorios de mutación a partir de subtipos individuales estructurados espacialmente en vías ecosistémicas entre el hemisferio norte y sur, con una respuesta inmune variables entre sus hospederos303.
Los VI-A se caracterizan por presentar una polimerasa trimérica PA, PB1, PB2 responsable de diversos procesos involucrados con la replicación viral y su interacción específica con su hospedero304,305. Cambios sutíles en su estructura pueden marcar la diferencia para la formación de un virus altamente citotóxico y letal137,306, como Taubenberger, J. K. et al. lo han propuesto para el virus H1N1 de 191866. De hecho la recreación experimental de la infección del mortal virus H1N1 en macacos comparada con un virus H1N1 contemporáneo ha probado ser letal307,308, produciendo serio daño en el tejido pulmonar con una extensión que varió entre 60 a 90%, asociada a lesión alveolar con abundante edema y exudado sanguinoliento así como una respuesta inmune sobre magnificada140
En la actualidad, se conocen 16 HA y 9 NA y su combinación permite diferenciar subtipos virales de influenza95,97,309. Asimismo, la mutación frecuente de los genes que codifican las glicoproteínas HA y NA determinan variantes que son clasificadas por el lugar geográfico, el número del cultivo y el año de identificación. Por ejemplo, las cepas A/Texas/36/91 (H1N1); A/Japón/305/57 (H2N2); A/Pekín/32/92 (H3N2); y B/Panamá/45/90, entre otras32.
Todos los tipos de hemoaglutinina de los linajes aviares, humanos, porcinos y equinos reconocen (RSAS) en los glucolípidos y gluproteínas de sus células diana. Los VIAH del subtipo humano se unen con predilección a las células diana mediante el enlace de la HA a los RSAS α -2-6- Gal, que son abundantes en el tracto respiratorio humano superior, en especial en la mucosa nasal. Los VI-A del subtipo aviar, como el H5N1, y los VI-A del linaje equino tienen predilección por los RSAS α-2-3-Gal, especialmente abundantes en los intestinos de las aves.
Mientras tanto los porcinos poseen RSAS α -2-6- Gal y RSAS α-2-3-Gal, es decir sus células diana puede fijar hemaglutininas de linajes animales diferentes310-317. En el caso de los VIAA los cambios de posición de aminoácidos en las proteínas secuenciadas por su genoma, presentan una mayor variabilidad en su PB1-F2, HA, NA, NS1, que las proteínas homologas de sus pares no aviares285. La HA y NA se consideran sumamente variables, debido a la presión inmune de los hospederos, en el caso de la NS1 se desconoce que es lo que induce su alta variabilidad pero se sabe que la misma juega un rol importante en el ciclo de vida viral285
Sin embargo, hoy se sabe que diversas células del tracto respiratorio humano, como las células epiteliales de la tráquea, los neumocitos tipo II, los macrófagos alveolares, las células caliciformis de las glándulas submucosas traqueales productoras de mucina y las células epiteliales cuboidales no ciliadas de los bronquiolos terminales, presentan aunque en menor proporción RSAS α-2-3-Gal, lo que puede permitir a los subtipos de IABP e IAAP como la H5N1 tener capacidad de union con estas células, lo que contribuiría a la lesión pulmonar318-325.
Es más, las hemoaglutininas (HA) no solo juegan un rol relevante en el reconocimiento sino también en la trasmisibilidad viral intraespecies, por lo que mutaciones mínimas en la distribución de aminoácidos de las HA podrían variar la preferencia por un RSAS determinado mejorando o aboliendo la transmisión326,327. Nuevamente el cerdo encuentra un rol protagonico328 como una coctelera en los que subtipos VIAA y VIAH podrian generar cambios completos en las hemoaglutininas. Tampoco debemos olvidar otras aves como el pollo y la codornis en que la presencia de RSAS α -2-6- Gal y RSAS α-2-3-Gal a sido intestinal es comprobada316,329
A pesar de que los VIAA están adaptados para crecer a una temperatura promedio de 40 ºC, es decir la temperatura del tracto entérico de las aves, la temperatura de la corriente de aire de las vías aéreas humanas es en su porción proximal es inferior para llegar a 37ºC en las porciones distales, lo que podría sugerir una transmisión zoonótica limitada por la temperatura entre hospederos humano y el VIAA37, sin embargo la transmisibilidad y capacidad de infectividad de las células hospederas por los VIAA parece obedecer a un patron multifactorial, con multiples hospederos intermediarios y escenarios multiecosistémicos que en esencia podrian favorecer el salto de especies o el reodenamiento viral.
También se ha propuesto que células caliciformes a nivel del aparato respiratorio secretoras de mucinas expresan ácido sialico α-2-3 siálico , y por lo tanto, pueden actuan como una barrera para la gripe zoonótica infecciones virales299,330 Como es de esperar los VIAH son ávidos por el aparato respiratorio alto humano, mientras que los VIAA lo son por el bajo.331,332
La fisiopatología de la infección de los VIAH es producto de una relación directa entre el grado de replicación viral en las células epiteliales respiratorias con RSAS α -2-6- Gal que son abundantes en el tracto respiratorio humano superior290, en especial en la mucosa nasal y traqueobronquial, sus efectos citopáticos directos sobre los epitelios nasal y traqueobronquial y de la activación de la cascada de citoquinas (CQ)298,333,334.
La CQ produce una respuesta proinflamatoria a nivel local en el fluido nasofaríngeo y traqueobronquial como a nivel sistémico, a través del torrente sanguíneo335,336. Esta respuesta, se encuentra caracterizada por el aumento significativo en las concentraciones de diversas CQ, como interleuquinas 6 (IL-6)323,324, 8 (IL-8)337, factor de necrosis tumoral alfa (TNF-α)323, interferones alfa (INF-α)324 y gama (IFN-α)325.
También se sabe que la NA de los VIAH induce a los macrófagos locales a secretar interleuquina 1 (IL-1), TNF-α, INF-β y quimioquinas338,339 y que los viriones completos de los VI-A hacen lo propio con las células mononucleares sanguíneas estimulándolos a producir IL- 1, IL-6, TNF-α340. Por tanto, está completamente claro que los VI-A tienen el potencial directo para inducir la producción de varias CQ324.
Otro importante efecto del virus sobre el huésped es la disfunción fagocitaria temporal que produce sobre neutrófilos y macrófagos. Esta depresión funcional se produce como resultado de la unión de la HA de los VIAH sobre las superficies de los fagocitos en donde se encuentran receptores que contienen sialoglicoproteínas341. El grupo de las glicoproteínas de origen no inmune conocidas como colectinas (colágeno-lectinas) del huésped presentes en la sangre y en el surfactante pulmonar tiene un rol inicial en la defensa no inmune del huésped contra el virus: favorece la opsonización, por agregación de las partículas virales gracias a su alto grado de multimerización, y protege, a su vez, a los neutrófilos y macrófagos del proceso de desactivación por el VI-A infectante341.
En cuadro severos por VIAH se han descritos procesos de hipercitoquinemia asociado a incrementos de IL-6, IL-8, proteína interferon inducible de citoquinas (IP-10), Monoquinas inducidas por IFN-γ (MIG), asi como hiperactivación de la proteína kinasa activadora del mitógeno fosfo-p38 en células T de ayuda o hellpers. La inhibición de la vía de señalización de las proteínquinasas fue bloqueada y una respuesta Th1 amplificada fue observada durante el estadío temprano de la replicación viral325. Esto concuerda con otros estudios previos323,324,342. La IP-10 y la MIG asi como y proteína 1 de hiperactivación del monocito (MCP-1) podrían explicar la coomorbilidad elevada en personas mayores de 60 años325. Estos perfíles de citoquinas observados en pacientes con VIAH con cuadros severos es parecido al de aquellos pacientes con VIAA H5N1325. Asi por ejemplo en los VIAA por H5N1 los perfiles de hipercitoquinemia se caracterizan por concentraciones plasmáticas elevadas de IL-6, IL-8, PI-10, monoquinas inducidas por IFN-γ (MIG), MCP-1, IL-10, e IFN-γ236,298,343,345. Se aprecia además que en los VIAA contemporanéos la NS1 juega un papel importante en la virulencia inhibiendo intracelularmente la maquinaria de defensa antiviral, como la producción de factor nuclear kappa (FN- k) y proteinquinasas activadoras de ARN346,347. Esto resulta importante cuando se tiene en cuenta que las pandemias de 1957 y 1967 los nuevos VIAH H2N2 y H3N2 respectivamente sólo tuvieron cambios en los antígenos de superficie en el la polimerasa de la proteína PB1 a partir de componentes aviares, mientras el resto continuó teniendo una procedencia humana. En cambio los recientes brotes H5, H7 y H9 han sido causados por VIAA en las que todos los genes se han obtenido de componentes aviares, incluido el NS285.
Aunque los VIAA, como el H5N1, tienen predilección por los sialosacáridos RSAS-2-3-Gal, abundantes en los intestinos de las aves298,348 el tracto respiratorio bajo humano, las células epiteliales de la tráquea, los neumocitos tipo II, los macrófagos alveoláres y las células epiteliales cuboidales no ciliadas de los bronquiolos terminales, presentan el sialosacárido RSSA-2-3-Gal, lo que permite al subtipo de IAAP H5N1 tener predilección por estas células, lo que contribuye a la severidad de la lesión pulmonar298,336.
Actualmente el número de pacientes fallecidos por H5N1 con necropsia efectuada sigue siendo bajo, los hallazgos en las mismas muestran a nivel pulmonar una extensa consolidación y un grado variable de hemorrágia; los neumocitos son el blanco primario de la infección, lo que genera un daño alveolar difuso (DAD) intenso y fulminante asociado con un infiltrado instersticial linfocitario y fibrosis122,236. En los casos de corta duración menor a los diez días se observó inflamación exudativa difusa con daño alveolar caracterizado por edema, exudado fibroso y membranas hialinas323,308,8, en los casos de mayor duracion el curso a sido hacia una fase proliferativa fibrosa alveolar y fibrótica insterticial.337,342,349
Hiperplasia de neumocitos II, hemofagocitosis, descamación epiteliar intraalveolar, bronquiolítis, dilatación cistica de los espacios aereos, hemorragia, pleurítis, neumonítis y apoptosis del epitelio alveolar y macrofagos también ha sido observada.338,342,350-351 Una depleción linfoide en el bazo y en los nódulos linfáticos, así como necrosis tubular aguda, también han sido descritas340. Así, los pacientes infectados por el subtipo VI-A H5N1 presentan primariamente una neumonía viral, la cual se complica con síndrome de distrés respiratorio agudo (SDRA), síndrome de disfunción multiorgánica (SDM) y síndrome hemofagocítico severo (SHF)353,354. En ocasiones, se ha observado afectación cardíaca, síndrome de Reye, síndrome séptico sin bacteriemia documentada, encefalítis, entre otros trastornos.
Con respecto a la nueva influenza A/H1N1 es poco lo que se sabe. Por el momento con los datos preliminares con los que se cuenta se ha observado que el virus viene mostrando un comportamiento benigno. La mayoría de casos reportados han comprendido gente joven menor de 25 años pero el grueso de casos fatales se ha concentrado en la población de 30 a 50 años en donde un grupo significativo de los fallecidos tenían morbilidad sobreagregada, como enfermedad pulmonar previa, desordenes autoinmunes, diabetes, enfermedad cardiovascular y obesidad14.
DIAGNÓSTICO
Para el diagnóstico de la influenza y de los virus respiratorios en general y para el diagnóstico de la infección por la nueva influenza A H1N1, son necesarios algunos pasos esenciales, como a) La correcta obtención de la muestra y el correcto registro de la información clínico epidemiológica356 b) Correcto almacenaje, transporte y envio de la muestra de acuerdo a las normas de IATA357 y c) La aplicación de la técnica de laboratorio adecuada y su interpretación, asi como los cuidados de bioseguridad358
La muestra adecuada para el diagnóstico es la obtenida a través del hisopado nasal y faríngeo, también puede hacerse aspirado nasofaríngeo o lavado bronqueolo alveolar o aspirado traqueal344. El hisopo que se utiliza es el de dacrón o poliéster estéril. Utilizando un hisopo en cada caso se obtiene una muestra de los cornetes nasales y otra de la faringe, ambos hisopos se introducen en un criovial conteniendo un medio de transporte viral. Registrar el nombre y el código en el vial. Para evitar lesiones en la mucosa, se debe embeber el hisopo en el medio de transporte viral, y escurrir el exceso, antes de tomar la muestra. Lo más importante es obtener células, no secreciones.
En el registro de la información es imprescindible consignar además del nombre, la edad, procedencia, la fecha de inicio de síntomas y la fecha de obtención de la muestra, que son críticos para la interpretación. El transporte de la muestra se hace manteniendo una cadena de frio ( 2º C a 8º C)
Es importante destacar la necesidad de la protección personal durante la obtención de la muestra para ello utilizar los equipos de protección personal indicados para agentes infecciosos transmitidos por aerosoles, como mandilón, 2 pares de guantes descartables (un par debe ser desechado después de cada obtención), respirador NIOSH N95, lentes protectores y gorros descartables346.
Entre los métodos utilizados para el diagnostico de la influenza en general tenemos las pruebas rápidas , la inmunoflorescencia directa e indirecta, el aislamiento viral y las técnicas moleculares359. Las pruebas rápidas están basadas en el uso de anticuerpos monoclonales, y siendo nuevo el virus de la influenza A H1N1, no existen aun en el mercado estas pruebas que puedan detectar con una adecuada sensibilidad y especificidad al nuevo virus. Se ha tratado de utilizar estas pruebas para el diagnóstico durante esta pandemia, sin embargo las pruebas rápidas muestras entre 30 y 50% de falsos negativos360. En el Perú, cuando se comparó el diagnóstico del RT-PCR en tiempo real con la prueba de inmunofluorescencia se ha encontrado una elevada proporción de falsos negativos con esta última prueba, llegando hasta el 70% , con una sensibilidad de 30,8%, especificidad de 97.9% VPP 86,1% y VPN 77,1% (comunicación INS, Junio 2009); por lo que para fines prácticos estas pruebas no son adecuadas para el diagnostico de la Nueva influenza A H1N1 y pueden inducir a error en el diagnóstico, siendo esto un aspecto critico cuando estamos frente a un nuevo agente.
Luego de la presentación de los primeros casos en México y EEUU, el CDC diseñó una prueba de RT-PCR en tiempo real, que utiliza primer específicos del nuevo virus de la influenza A H1N1, la cual ha sido recomendada por la OMS361,362 y es utilizada en todos los países del mundo para el diagnóstico de esta nueva infección. Otro de los métodos para la confirmación del diagnóstico es el aislamiento viral.
El aislamiento viral, se realiza utilizando cultivo celular MDCK y permite identificar el tipo de virus respiratorio y proporciona las cepas virales para la producción de vacuna, que es útil en las estaciones de invierno del año siguiente en los hemisferios sur y norte. Este procedimiento de aislamiento viral tiene alta especificidad, sin embargo es complejo, de alto costo y requiere de laboratorios con bioseguridad 3, como es el caso del Instituto Nacional de Salud en el Perú.
Las técnicas moleculares permiten identificar el ARN viral en muestras directas y cultivos, tienen una alta sensibilidad y especificidad, son métodos rápidos dentro de las 24 horas. Requieren de recursos humanos altamente capacitados, equipos de alta tecnología e infraestructura adecuados. Entre estas tenemos el RT –PCR clásico que permite identificar la influenza A estacional.
El RT-PCR en tiempo real es el que permite diagnosticar con alta sensibilidad y especificidad al nuevo virus de Influenza A H1N1. Esta es una prueba cualitativa, que utiliza secuencias específicas del genoma del virus (Primers) para amplificar una región del material genético del virus Influenza A H1N1, siendo la detección del virus en tiempo real. Esta prueba está disponible en el INS, que lo viene desarrollando desde el ingreso de la nueva influenza A H1N1 en nuestro país.
Las técnicas moleculares también permiten investigar las mutaciones que pueda tener el virus o los niveles de resistencia que puedan aparecer durante la pandemia o en periodos interpandémicos.
ASPECTOS CLÍNICOS
La Infección por este nuevo Virus de la Nueva Influenza A H1N1 se caracteriza por presentar una morbilidad elevada y una baja mortalidad363. El periodo de incubación de la enfermedad en el ser humano varía entre 1 y 7 días, y es dependiente de la concentración del inóculo viral y de su capacidad replicativa. La duración entre el inicio de la enfermedad y la atención hospitalaria fluctúa en un rango de 0 a 7 días. Así mismo la duración de inicio y resolución de fiebre fue de 1 a 8 días aproximadamente, mientras que en niños se pude prolongar hasta los 10 a 12 días364. La edad de los pacientes con infección por virus de la nueva Influenza A H1 N1 fluctúa entre 3 meses a 81 años, pero un 40 % de la población afectada es de 10 a 18 años de edad denotando un comportamiento inusual en esta nueva infección.365
La información clínica y patológica preliminar reportada a la OMS sugería similitud entre casos severo de la infección por Virus de la nueva Influenza A H1N1 y casos humanos de Infección por Virus de Influenza Aviar H5N1. Estando el espectro clínico de la infección por Virus de la nueva Influenza A H1N1 aun en definición, pero ambas la forma leve auto limitada y la forma severa, incluyendo la falla respiratoria y muerte han sido observado entre los pacientes identificado con este nuevo virus366.
La infección por el virus de la Nueva Influenza A H1N1puede ocasionar una gran diversidad desde cuadros no febriles, infección respiratoria aguda del tracto respiratoria alto leve a severa baja cuya intensidad que puede progresar a neumonía ocasionando la muerte del paciente.
Dentro de la sintomatología y signología se destaca comúnmente malestar general , fiebre, seguidos de manifestaciones respiratorias altas como tos y rinorrea así como mialgias y cefalea; otras manifestaciones menos frecuentes son la conjuntivitis. Los síntomas gastrointestinales como nauseas, vómitos y diarreas se ha observado en un 25% de los pacientes de Estados Unidos, siendo esto diferente a los casos registrados en Japón donde se registro en el 14% de casos364,366. En el Perú los síntomas típicos han sido similares y un 20% se ha asociado a diarreas, nauseas y dolor abdominal. Existe similitud entre los casos de paciente infectados con el Virus de influenza A H1N1 y virus de de Influenza estacional, la severidad del la enfermedad y la muerte de asociada son en gran parte el resultado de complicaciones secundaria, incluyendo neumonía viral neumonía bacteriana secundaria Streptococcus pneumoniae, Streptococcus grupo A, Staphylococcus aureus367,368 y exacerbación de enfermedad crónica previa. Estas mismas complicaciones pueden ocurrir en pacientes con infección por Virus de la nueva Influenza A H1N1. La población de riesgo para presentar estas complicaciones son : niños menores de 5 años, adultos de 65 años, niños y adultos con morbilidades medicas crónicas y gestantes365.
A diferencia de las poblaciones humanas afectadas por el virus de influenza aviar H5N1 en la que se observa una tasa de letalidad extremadamente elevada (60,5%) y una asociación de contacto entre humanos y aves esencial, en la actual pandemia por el virus de la nueva Influenza A H1N1la tasa de letalidad mudial bordea el 0,3% según los reportes de la OMS, mientras que en le Perú es mucho menor todavía.
Entre los 45 casos fatales en México el 54 % ocurrió en pacientes previamente saludable, siendo la población de 20 a 59 años la más afectada. Las comorbilidades como asma, enfermedad cardiovascular, diabetes, obesidad mórbida, enfermedad autoinmune, neoplasia, tabaquismo, malformaciones congénitas, sindrome de Down, etc., se evidenciaron en un 46% de pacientes que fallecieron366. El tiempo medio de aparición de la enfermedad a la hospitalización fue de 6 días (rango, 1-20 días) en 45 casos fatales, en comparación con una mediana de 4 días en los casos hospitalizados en los EUA. En el grupo de 20 mujeres embarazadas en los EUA confirmadas que se infectaron con el nuevo virus 3 requieron hospitalización, 1 de los cuales falleció, es paciente inicio la terapia antiviral 13 días después del inicio de la enfermedad.
TRATAMIENTO
Hasta esta fecha no existe vacuna disponible, por ello el uso de antivirales es lo más recomendable tanto para la prevención como para el tratamiento.
Al tratarse de una infección viral similar a la Influenza estacional en la mayoría de los casos, esta se autolomita, por lo cual la finalidad del tratamiento antiviral es la de aminorar los síntomas así como disminuir la posibilidad de complicaciones asociadas a esta infección. Actualmente existe 2 grupo de antivirales que se usan en el caso de la Infección por Influenza y son: los inhibidores de neuroaminidasa como el oseltamivir y zanamivir y los adamantanos como la amantadina y la rimantadina, siendo el virus de la nueva Influenza A H1N1, resistente a estas últimas.
La nueva gripe A(H1N1) es susceptible a inhibidores de neuroaminidasa - oseltamivir (Tamiflu), zanamivir (Relenza) pero resistente a los inhibidores de canales de iones M2 (amantadina, rimantadina). Un detalle interesante es que durante el año 2008 a 2009 la influenza estacional del tipo H1N1 que circuló por EEUU era resistente a Oseltamivir369. Los medicamentos antivirales trabajan mejor si se inician dentro de los dos primeros días de presentarse las molestias
El tratamiento de soporte, para controlar la fiebre y otros síntomas incluye el uso del paractetamol y AINES, en cambios los salicilatos y la aspirina no deben utilizarse en niños y adultos jóvenes debido al riesgo del síndrome de REYE (daño hepático severo)370.
La OMS recomienda la administración de Oseltamivir o zanamivir, pero por ser el ultimo por vía inhalatoria se tiene dificultades para su uso masivo.
La efectividad del uso de antivirales esta en plena evaluación en la presente pandemia, así también se ha tenido que modificar las restricciones en cuanto al grupo etáreo dado que FDA indica su uso en niños menores de 1 año (anteriormente se indicaba en niños mayores de 1 año), y quimioprofilaxis en niños mayores de 3 meses(anteriormente se indicaba en niños mayores de 1 año) 371.
La OMS y el MINSA han elaborado guías específicas para el manejo de los casos, que varían según la gravedad tantopara el manejo ambulatorio y de hospitalización. De esta guía y de la Guia del Hospital Daniel Álcides Carrión del Callao hemos recogido los Fluxogramas 1 y 2372 y fluxogramas de manejo con los antivirales. Los contactos con enfermos afectados de la nueva Influenza humana deben recibir también quimioprofilaxis con antivirales, los que tienen demostrado una eficacia entre 70 a 90%. Para un adulto normal se recomienda el uso de oseltamivir una tableta via oral de 75 mg cada día por siete a 10 días. El oseltamivir requiere correción de dosis cuando la depuración de creatinina baja a menos de 30 ml/min.
CONCLUSIONES
-
La evidencias científicas indican que existe un patrón congruente entre segmentos genómicos de los VI circulantes desde la aparición del VIAH H1N1 en 1918 hasta la fecha.
-
La infección cruzada y el salto esporádico interespecies es un hecho tangible basado en las evidencias científicas. De hecho en la actualidad ya se han reportado casos de infección por el nuevo subtipo A H1N1 desde humanos a cerdos en Canada y Argentina373.
-
Los diferentes virus de influenza tanto de los linajes humanos, aviares y porcinos constituyen un amenaza permanente para la salud y estabilidad social del mundo.
-
La enfermedad por el nuevo VI A/H1N1actualmente se encuentra en fase de pandemia, diseminándose por todo el mundo con una tasa de letalidad baja hasta el momento.
-
Una segunda oleada por este nuevo virus con cambios antigénicos producto de las derivas antigénicas por presión selectiva puede ocurrir en los próximos meses con resultados impredecibles en el aumento de la tasa de letalidad.
-
La clínica del nuevo VI A/H1N1 es muy similar a la observada en las influenzas interpandémicas o estacionales. La mayor mortalidad se ha reportado en pacientes con comorbilidades tales como obesidad mórbida, enfermedad cardiovascular y respiratoria como asma, EPOC, etc.
-
A la fecha se cuenta con dos inhibidores de la neuroaminidasa para la terapia del nuevo VI A/H1N1 conocidos como oseltamivir y zanamivir. Estos deben ser manejados prudentemente para evitar la aparición de resistencia.
REFERENCIAS BIBLIOGRÁFICAS
1. Osores Plenge Fernando, Cabezas Sáchez César, Gómez Benavides Jorge , Maguiña Vargas Ciro. Influenzas humana y aviar: amenaza de una pandemia humana. Acta méd. peruana, ene./abr. 2006, vol.23, no.1, p.35-47.
2. Debra Elton, Paul Digard, Laurence Tiley and Juan Ortin. In: Influenza Virology: Current Topics. Editor: Yoshihiro Kawaoka School of Veterinary Medicine, University of Wisconsin-Madison, Madison, WI 53706, USA . Publisher: Caister Academic Press 2006. ISBN 978-1-904455-06-6.
3. Centers for Disease Control and Prevention CDC. USA. 2009. http://www.cdc.gov/h1n1flu/pdf/keyfacts.pdf
4. CDC. Swine Influenza A (H1N1) Infection in Two Children Southern California, March-April 2009. MMWR Morb Mortal Wkly Rep April 24, 2009; 58(15):400-402. http://www.cdc.gov/mmwr/preview/mmwrhtml/mm5815a5.htm
5. CDC. Outbreak of swine-origin influenza A (H1N1) virus infectionMexico, MarchApril 2009. MMWR Morb Mortal Wkly Rep May 8, 2009 / 58(17);467-470. http://www.cdc.gov/mmwr/preview/mmwrhtml/mm5817a5.htm
6. Thompson WW, Shay DK, Weintraub E, Brammer L. Influenza-associated hospitalizations in the United States. JAMA 2004;292:1333-1340.
7. Uyeki, TM, Zane SB, Bodnar UR, et al. Large summertime influenza A outbreak among tourists in Alaska and the Yukon Territory. Clin Infect Dis 2003;36:1095- 1102.
8. Miller JM, Tam TW, Maloney S, et al. Cruise ships: high risk passengers and the global spread of new influenza viruses. Clin Infect Dis 2000;31:433-438
9. Kohn MA, Farley TA, Sundin D, Tapia R, McFarland LM, Arden NH. Three summertime outbreaks of influenza type A. J Infect Dis 1995;172:246-249.
10. Gaillat J, Dennetière G, Raffin-Bru E, Valette M, Blanc MC. Summer influenza outbreak in a home for the elderly: application of preventive measures. J Hosp Infect 2008;70:272-277.
11. Young LC, Dwyer DE, Harris M, Guse Z, Noel V, Levy MH; Prison Influenza Outbreak Investigation Team. Summer outbreak of respiratory disease in an Australian prison due to an influenza A/Fujian/411/2002(H3N2)-like virus. Epidemiol Infect 2005;133:107-112.
12. Myers KP, Olsen CW, Gray GC. Cases of swine influenza in humans: a review of the literature. Clin Infect Dis 2007;44:10848.
13. Nicholson K, Wood J, Zambon M. Influenza. Lancet 2003; 362:1733-1745.
14. Bottenon CA, Aquilino J. Avian Influenza (The Bird Flu). A Worldwide Cause for Concern. International Foundation for the Conservation of Natural Resources. April 2004.
15. Michael J. Bradley, Susan J.Kutz, Emily Jenkins, Todd M. OHara. International Journal of Circumpolar Health 64:5 2005
16. Widjaja LS, Krauss L, Webby RJ, Xie T, Webster RG.Matrix gene of influenza viruses isolated from wild aquatic birds: ecology and emergence of influenza a viruses. J Virol 2004; 78: 8771-9.
17. Webster RG. The importance of animal influenza for human disease. Vaccine 2002; Suppl 2: S16-20.
18. Ce´ cile Viboud1, Khashayar Pakdaman, Pierre-Yves Boe¨ lle et al. Association of influenza epidemics with global climate variability. European Journal of Epidemiology 19: 1055–1059, 2004.
19. Andrew Comrie. Geography Compass 1/3 (2007): 325–339
20. Pablo M. Beldomenico, Damien O. Joly,Marcela M. Uhart, and William B. Karesh. in Wildlife Health as an Indicator of Climate Change in Global Climate Change and Extreme Weather Events, Understanding the Contributions to Infectious Disease Emergence: Workshop Summary. published by the National Academy of Sciences/Institute of Medicine of the National Academies 2008 ISBN: 0-309- 12403-4. P 192.
21. Monica Flores Luna. Actualización sobre la prevención de la influenza. Rev Soc Per Neumol, 2005; 49(1): 57-61
22. Leonor Jofré M., Cecilia Perret P , Jeannette Dabanch P Et al. Influenza: reemergencia de una antigua enfermedad y el riesgo de pandemia. Rev Chil Infect 2005; 22 (1): 75-88
23. Murphy BR, Webster RG. Orthomyxoviruses. In: Fields BN, Knipe DM, Howley PM, Chanock RM, Melnick JL, Monath TP, et al., editors. Fields virology. 3rd ed. Philadelphia: Lippincott-Raven Publishers; 1996. p. 1397–445.
24. Simonsen L, Clarke MJ, Williamson GD, Stroup DF, Arden NH, Schonberger LB. The impact of influenza epidemics on mortality: introducing a severity index. Am J Public Health 1997;87:1944–50.
25. Bridges CB, Fukuda K, Cox NJ, Singleton JA. Advisory committee on immunization practices: Prevention and control of influenza. Recommendations of the Advisory Committee on Immunization Practices (ACIP). MMWR Morb Mort Weekly Report 2001;50:1–44.
26. Heinen P. Swine influenza: a zoonosis. Vet Sci Tomorrow. 2002. p. 1-11.
27. Bottenon CA, Aquilino J. Avian Influenza (The Bird Flu). A Worldwide Cause for Concern. International Foundation for the Conservation of Natural Resources. 2004.
28. Karoline Bragstad1, Lars P Nielsen2 and Anders Fomsgaard. The evolution of human influenza A viruses from 1999 to 2006: A complete genome study. Virology Journal 2008, 5:40
29. Gripe en informe de la secretaría. Consejo Consultivo 111a reunión. WHO EB111/10 Influenza. 2002. p: 1-2
30. Stöhr K. InfluenzaWHO cares. Lancet Infect Dis. 2002 Sep;2(9):517
31. Prevention CfDCa (2008) Update: Influenza Activity- United States, September 30, 2007–February 9, 2008. MMWR 57: 179–183.
32. T. Pumarola, M.A. Marcos y M.T. Jiménez de Anta. Variaciones antigénicas del virus de la influenza como determinante epidemiológico clave. Vacunas 2002;3(Supl 1):1-4
33. Emergence of a Novel Swine-Origin Influenza A (H1N1) Virus in Humans. Emergence of a Novel Swine- Origin Influenza A (H1N1) Virus in Humans. N Engl J Med 2009;361.
34. Kendall P. Myers, Sharon F. Setterquist, Ana W. Capuano, and Gregory C. Gray. Infection Due to 3 Avian Influenza Subtypes in United States Veterinarians Clin Infect Dis. 2007 July 1; 45(1): 4–9.
35. Thijs Kuiken, Edward C. Holmes, John McCauley,Guus F. Rimmelzwaan, Catherine S. Williams,2 Bryan T. Grenfell. Host Species Barriers to Influenza Virus Infections. Science 2006, 312(5772): 394 – 397.
36. Christopher W. Olsen, Lynnette Brammer, Bernard C. Easterday, Nancy Arden, Ermias Belay,Inger Baker,and Nancy J. Cox. Serologic Evidence of H1 Swine Influenza Virus Infection in Swine Farm Residents and Employees. Emerging Infectious Diseases 2002; 8(8): 814-819.
37. P. C. Crawford, Edward J. Dubovi, William L. Castleman, Iain Stephenson, Et al. Transmission of Equine Influenza Virus to Dogs. Science 2005; 310(5747): 482 – 485.
38. J. S. Malik Peiris, Menno D. de Jong, and Yi Guan. Avian Influenza Virus (H5N1): a Threat to Human Health. Cli Microbiol Rev, Apr. 2007, 20(2): 243–267.
39. Margaret A. Scull, Laura Gillim-Ross, Celia Santos. Avian Influenza Virus Glycoproteins Restrict Virus Replication and Spread through Human Airway Epithelium at Temperatures of the Proximal Airways. PLoS Pathogens, 2009; 5(5): e1000424
40. V. S. Hinshaw, W. J. Bean, R. G. Webster and G. Sriram. Genetic reassortment of influenza A viruses in the intestinal tract of ducks . Virology 1980 102(2): 412-419
41. Martha I. Nelson and Edward C. Holmes .The evolution of epidemic influenza. Nature reviews genetics 2007; 8(3): 196-205
42 Toshihiro Ito, J. Nelson S. S. Couceiro, Sørge kelm Et al. Molecular Basis for the Generation in Pigs of Influenza A Viruses with Pandemic Potential J Virolgy 1998, 72(9) 7367–7373
43. Reid AT, Fanning T, Janczewski T, Taubenberger J. Characterization of the 1918 Spanish influenza virus neuraminidase gene. Proc Natl Acad Sci U S A 2000;97:6785– 90.
44. Capps J, Moody A. The recent epidemic of grip. JAMA. 1916;67:1349–50.
45. Reid AH, Fanning TG, Hultin JV, Taubenberger JK. Origin and evolution of the 1918 Spanish influenza virus hemagglutinin gene. Proc Natl Acad Sci U S A 1999;96:1651–6.
46. Fanning TG, Slemons RD, Reid AH, Janczewski TA, Dean J, Taubenberger JK. 1917 avian influenza virus sequences suggest that the 1918 pandemic virus did not acquire its hemagglutinin directly from birds. J Virol. 2002;76:7860–2.
47. Johnson NP, Mueller J. Updating the account lobal mortality of the 1918-1920 Spanish influenza pandemic. Bull Hist Med 2002;76:105-15.
48. Patterson KD, Pyle GF. The geography and mortality of the 1918 influenza pandemic. Bull. Hist. Med. (1991) 65:4–21.
49. Johnson NP, Mueller J. Updating the accounts: global mortality of the 1918–1920 Spanish influenza pandemic. Bull. Hist. Med. (2002) 76:105–115.
50. Burnet FM. Portraits of viruses: influenza virus A. Intervirology (1979) 11:201–214
51 Schafer J, Kawaoka Y, Bean W, Suss J, Senne D, Webster R. Origin of the pandemic 1957 H2 influenza A virus and the persistence of its possible progenitors in the avian reservoir. Virology 1993;194:781–788.
52. Scholtissek C, Rohde W, Von Hoyningen V, Rott R. On the origin of the human influenza virus subtypes H2N2 and H3N2. Virology 1978;87:13–20.
53. Kawaoka Y, Krauss S, Webster R. Avian-to-human transmission of the PB1 gene of influenza A viruses in the 1957 and 1968 pandemics. J Virol 1989;63:4603–4608.
54. José Neyra. En: Imágenes históricas de la Medicina Peruana. Capítulo VI: El primer Tisiólogo del Perú Dr. Aníbal Corvetto Bisagno. Lima : UNMSM, Fondo Editorial, 1997. ISBN: 9972-46-226-9
55. Rómulo Eyzaguirre. La gripe infantil en la epidemia limeña de 1919-1919. Anales de la facultad de Medicina 1920, Año III Nº 5;p. 251-263
56. Julián Arce. Algunas consideraciones sobre la insuficiencia renal de la gripe. 1918, Año I Nº 2 p. 18-24
57. Edmundo Escomel. La grippr RN Rio de Janeiro, Montevideo y Buenos Aires. Medidas profilacticas y terapêuticas: Informe presentados a La Dirección de Salubridad. La Crônica Médica 1919: 373-375
58. Roberto J. Cornejo Ubilluz – Jorge Villena Piérola. Departamento de Epidemiología MSPS - UNMSM 1957
59. Ministerio de Salud. Oficina General de Epidemiología. Boletín Epidemiológico Semanal No 11-2004
60. Falcon Caceres Luiz, Torres Ivonne, León Carlos, Guerra Diaz Ana. Agentes virales en niños menores de un año, hospitalizados por IRA baja en el (HEP) Hospital de Emergencias pediatricas 2001-2002. Rev Soc Per Neumo 2004, 48(2): 145-148. http://sisbib.unmsm.edu.pe/BVrevistas/enfermedades_torax/v48_n2/Pdf/a07.pdf
61. Juan Arrasco . Vigilancia centinela de influenza y otros virus respiratorios en la Región Tumbes – I trimestre 2006. Bol. Epidemiol. (Lima) 15 (19), 2006 Pag 12
62. Yvonne Torres de Yon, Julio Mayca P, Fernando Llanos- Zavalaga, José Velásquez H, Silvia Capristano V. Virus influenza y el diagnóstico diferencial de sintomáticos febriles en la costa norte delPerú (mayo, 2001). Rev peru med exp salud publica 21(1), 2004. Pag 392
63. Jorge Gómez Benavidez, César Munayco Escate, Técnica Marienela Ulloa Rea. Boletin del Instituto Nacional de Salud. Año 2 N 41 Octubre 2005. Pag 4.
64. Vigilancia Centinela de Influenza y otros virus respiratorios. Bol. Epidemiol. (Lima) 18 (20), 2009: 392. http://www.dge.gob.pe/boletines/2009/20.pdf Accesado 09/06/09
65. Vigilancia Centinela de Influenza y otros virus respiratorios. Bol. Epidemiol. (Lima) 18 (21), 2009: 410- 411. http://www.dge.gob.pe/boletines/2009/20.pdf Accesado 09/06/09
66. Laguna- Torres VA, Gómez J, Ocaña V, Aguilar P, Saldarriaga T, et al. 2009 Influenza-Like Illness Sentinel Surveillance in Peru. PLoS ONE 4(7): e6118. doi:10.1371/ journal.pone.0006118
67. Tatiana Saldarriaga, V. Alberto Laguna-Torres, Juan Arrasco Et al.Carácterísticas clínicas y moleculares de un brote de influenza en dos bases militares, Tumbes- Perú, 2007 . Rev Peru Med Exp Salud Publica. 2008; 25(1):35-43.
68. Jorge Alarcón Villaverde . Perú: El perfil epidemiológico en un contexto de demanda reprimida de servicios de salud (Documento 2). Partners for Health Reform. Abt Associates Inc. 4800 Montgomery Lane, Suite 600 Bethesda, Maryland 20814 Tel: 301/913-0500 Fax: 301/652-3916 In collaboration with: Development Associates, Inc. Emory University Rollins School of Public Health Philoxenia International Travel, Inc. Program for Appropriate Training in Health SAG Corporation Social Sectors Development Strategies, Inc. Training Resource Group Tulane University School of Public Health and Tropical Medicine University Research Co., LLC.
69. Michael T. Osterholm. Preparing for the Next Pandemic. N Engl J Med 2005; 352;18: 1839-1842
70. Pilar Pérez Breña. En Monografía XXI. Influencia aviar y Gripe humana de origen aviario. La prevención y lucha contra una posiblepandemia de gripe aviar. Monografías de la Real Academia Nacional de Farmacia.Instituto 2009. www.analesranf.com/index.php/mono/issue/view/116. Accesado 6l 11/06/09
71. Oldstone MBA (1998). Influenza viruses: The pest can returm. In: Texbook of viruses, plagues and history. First Edition. New York, Oxford University Press, Inc. 1998:222- 239.
72. Ayora-Talavera G. Influenza: Historia de una enfermedad. Rev Biomed 1999; 10(1): 57-59.
73. Potter CW. A history of influenza. J Appl Microbiol. 2001; 91(4):572-579
74. Lazzari S, Stohr K. Avian influenza and influenza pandemics. Bull WHO. 2004; 82(4): 242-243.
75. Agustín Muñoz-Sanz. La gripe de Cristóbal Colón. Hipótesis sobre una catástrofe ecológica. Enferm Infecc Microbiol Clin 2006;24(5):326-34
76. Guerra F. La epidemia americana de influenza en 1493. Revista de Indias.1985;176:325-47.
77. Guillermo Olague de Ros. La epidemia europea de gripe de 1708-1709. Difusión teporoespacial e interpretaciones contemporáneas. Acta Hispanica at medicinae Scienteariumque Hystoriam Illustrandam; 1981; 1: 51-86
78. Beveridge WI. The chronicle of influenza epidemics. Hist Philos Life Sci. 1991;13(2):223-34.
79. Richard E. Shope. Influenza: History, Epidemiology, and Speculation. Public Healht Report. The R.E. Dyer lecture 1958; 73(2): 165-179
80. Weir E, Wong T, Gemmill I. Avian influenza outbreak: update Canadian Medical Association Journal. Canadian Medical Association Public Health 2004; 170.
81. Haemophilus influenzae. En Harrison Principios de Medicina Interna 16a edición
82. Shope R.E. III. Filtratión experiments and etiology. J Exp Med 1931;54: 373-380
83. Smith W., Andrewes C.H. and Landlaw P.O. A virus obtained fron influenza patients. Lancet 1933; 2:66
84. The Threat of Pandemic Influenza: Are We Ready?. Stacey L. Knobler, Alison Mack, Adel Mahmoud, Stanley M. Lemon, Editors. The National Academies. 2005. ISBN: 0-309-54685-0
85. Hay AJ, Gregory V, Douglas AR, Lin YP. The evolution of human influenza viruses. Philos Trans R Soc Lond B Biol Sci 2001; 356:1861-70.
86. John Treanor. Influenza Vaccine Outmaneuvering Antigenic Shift and Drift. N Engl J Med. 2004;350;3
87. Taubenberger JK, Morens DM: 1918 Influenza: the Mother of All Pandemics. Emerg Infect Dis 2006, 12:15- 22.
88. John M. Barry. The great influenza. The epic history of the deadtliest plague in history. Edit for Penguin, 2005 ISBN 0143036491, 9780143036494. p546
89. Terrence M. Tumpey, Christopher F. Basler, Patricia V. Aguilar Et al. Characterization of the Reconstructed 1918 Spanish Influenza Pandemic Virus. Science 2005; 310: 77-80
90. Taubenberger JK , Ann H. Reid, Thomas A Janczewski and Thomas G. Fannig, Integrating historical, clinical and molecular genetic data in order to explain the origin and virulence of the 1918 spanish influenza virus. Phil Trans. R. Soc Lond. B 2001; 356: 1829-1839
91. Jeffery K. Taubenberger J.K, Ann H. Reid, Amy E. Krafft, Karen E. Bijwaard, Thomas G. Fanning Initial Genetic Characterization of the 1918 Spanish Influenza Virus Science 1997; 275(5307), 1793-1796.
92. Jeffery K Taubenberger*, Johan V Hultin, and David M Morens. Discovery and characterization of the 1918 pandemic influenza virus in historical context. Antivir Ther. 2007 ; 12(4 Pt B): 581–591.
93. Taubenberger JK, Reid AH, Lourens RM, Wang R, Jin G, Fanning TG. Characterization of the 1918 influenza virus polymerase genes. Nature. 2005;437:889–93.
94. Basler, C. F. et al. Sequence of the 1918 pandemic influenza virus nonstructural gene (NS) segment and characterization of recombinant viruses bearing the 1918 NS genes. Proc. Natl Acad. Sci. USA 98, 2746–-2751 (2001).
95. Koen JS. A practical method for field diagnosis of swine diseases. Am J Vet Med 1919;14:468-70.
96. Laidlaw PP. Epidemic influenza: a virus disease. Lancet 1935;1:1118-24.
97. Taubenberger JK, reid AH, Fanning TG. The 1918 influenza virus: a killer comes into view. Virollogy 2000, 274:241-245
98. Hana M. Weingartl, Randy A. Albrecht, Kelly M. Lager, Et al Experimental Infection of Pigs with the Human 1918 Pandemic Influenza Virus. Journal of Virology, 2009, 83(9): 4287-4296
99. Kendal AP, Noble GR, Skehel JJ, Dowdle WR. Antigenic similarity of influenza A (H1N1) viruses from epidemics in 1977–1978 to Scandinavian strains isolated in epidemics of 1950–1951. Virology. 1978;89:632–6.
100. Lei Suna, Guihong Zhangb, Yuelong Shuc Et al.Genetic correlation between H3N2 human and swine influenza viruses. Journal of Clinical Virology 44 (2009) 141–144
101. Richt JA, Lager KM, Janke BH, Woods RD, Webster RG, Webby RJ. Pathogenic and antigenic properties of phylogenetically distinct reassortant H3N2 swine influenza viruses cocirculating in the United States. J Clin Microbiol 2003;41(7):3198–205.
102. Zhou NN, SenneDA, Landgraf JS,Swenson SL, Erickson G,RossowK, et al. Genetic reassortment of avian, swine, and human influenza A viruses in American pigs. J Virol 1999;73(10):8851–6.
103. Zhou NN, Senne DA, Landgraf JS, Swenson SL, Erickson G, RossowK, et al. Emergence of H3N2 reassortant influenza A viruses in North American pigs. Vet Microbiol 2000;74(1–2):47–58.
104. Lin YP, Gregory V, Bennett M, Hay A: Recent changes among human influenza viruses. Virus Research 2004, 103:47-52.
105. Derek J. Smith, Alan S. Lapedes, Jan C. de Jong, Theo M. Bestebroer, Guus F. Rimmelzwaan, Albert D. M. E. Osterhaus, and Ron A. M. Fouchier Mapping the Antigenic and Genetic Evolution of Influenza Virus .Science 2004 305: 371-376
106. Paget WJ, Meerhoff TJ, Goddard NL: Mild to moderate influenzaactivity in Europe and the detection of novel A(H1N2) and Bviruses during the winter of 2001-02. Euro Surveill 2002,7:147-157
107. Ellis JS, Alvarez-Aguero A, Gregory V, Lin YP, Hay A, Zambon MC:Influenza AH1N2 viruses, United Kingdom, 2001- 02 influ-enza season. Emerg Infect Dis 2003, 9:304-310.
108. Chi SX, Bolar TV, Zhao P, Tam JS, Rappaport R, Cheng S: MolecularEvolution of Human Influenza A/H3N2 Virus in Asia adnEurope from 2001 to 2003. Journal of Clinical Microbiology 2005,43:6130-6132.
109. Barr IG, Komadina N, Hurt A, Iannello C, Tomasov R, Shaw R, Dur-rant C, Sjogren H, Hampson AW: An Influenza A (H3) Reassor-tant Was Epidemic in Australia and New Zealand in 2003. JMed Virol 2005, 76:391-397.
110. Update: Influenza Activity United States and Worldwide, 2004-2005 Season. MMWR 2005;54(25);631- 634
111. Fluview. 2008-2009 Influenza Season/ Week 18 ending May 9, 2009 Center of Control Diseases. http://www.cdc.gov/flu/weekly/pdf/External_F0918.pdf. Accesado 19/05/09
112. Andrew W Park, Kathryn Glass Dynamic patterns of avian and human infl uenza in east and southeast Asia. http://infection.thelancet.com Vol 7 August 2007: 543-548
113. Bean WJ, Kawaoka Y, Wood JM, Pearson JE, Webster RG. Characterization of virulent and avirulent A/Chicken/ Pennsylvania/83 influenza A viruses: potential role of defective interfering RNAs in nature. J Virol 1985;54:151- 60.
114. Horimoto T, Rivera E, Pearson J, Senne D, Krauss S, Kawaoka Y, et al. Origin and molecular changes associated with emergence of a highly pathogenic H5N2 influenza virus in Mexico. Virology 1995;213:223-30.
115. Robert G. Webster. Influenza: An Emerging Disease. Emerging Infectious Diseases 1998; 4(3): 436-441
116. Senne DA, Pearson JE, Panigrahy B. Live poultry markets: a missing link in the epidemiology of avian influenza. In: Proceedings of the 3rd International Symposium on Avian Influenza; 1997 27-29 May; The Wisconsin Center, The University of Wisconsin-Madison. p. 50-8.
117. Kurtz, J., R. Manvell, and J. Banks. 1996. Avian influenza virus isolated from a woman with conjunctivitis. Lancet 348:901–902.
118. Taylor, H. R., and A. J. Turner. 1977. A case report of fowl plaque keratoconjunctivitis. Br. J. Ophthalmol. 61:86–88.
119. DeLay, P. D., H. L. Casey, and H. S. Tubiash. 1967. Comparative study of fowl plaque virus and a virus isolated from man. Pub. Health Rep. 82:615– 620.
120. Beigel H, Phil JP, Han AM et al. Avian influenza A (H5N1) infections in humans. New Engl J Med 2005;353(13):1374- 1385.
121. Avian influenza H5N1 Timeline. OIE - World Organisation for Animal Health 2009 http://www.oie.int/eng/info_ev/en_AI_factoids_H5N1_Timeline.htm
122. World Healht Organization.Cumulative Number of Confirmed Human Cases of Avian Influenza A/(H5N1) Reported to WHO 2 june 2009. http://www.who.int/csr/disease/avian_influenza/country/cases_table_2009_06_02/en/index.html
123. Taronna R. Maines, Xui Hua Lu, Steven M. Erb Et al.Avian Influenza (H5N1) Viruses Isolated from Humans in Asia in 2004 Exhibit Increased Virulence in Mammals. J Virol 2005 79(18):11788-11780
124. Guus F. Rimmelzwaan, Debby van Riel, Marianne Baars Et al.Influenza A Virus (H5N1) Infection in Cats Causes Systemic Disease with Potential Novel Routes of Virus Spread within and between Hosts. American Journal of Pathology 2006; 168 (1)1: 176-183
125. Thijs Kuiken, Guus Rimmelzwaan, Debby van Riel, Geert van Amerongen, Marianne Baars, Ron Fouchier, Albert Osterhaus. Avian H5N1 Influenza in Cats. Science 2004: 306(5694): 241
126. Daesub Song, Bokyu Kang, Chulseung Lee.Transmission of Avian Infl uenza Virus (H3N2) to Dogs. Emerging Infectious Diseases 2008; 14(5): 741-746
127. Lin, Y. P., M. Shaw, V. Gregory, K. Cameron, W. Lim, A. Klimov, K. Subbarao, Y. Guan, S. Krauss, K. Shortridge, R. Webster, N. Cox, and A. Hay. 2000. Avianto- human transmission of H9N2 subtype influenza A viruses: relationship between H9N2 and H5N1 human isolates. Proc. Natl. Acad. Sci. USA 97:9654-9658
128. Peiris, M., K. Y. Yuen, C. W. Leung, K. H. Chan, P. L. S. Ip, R. W. M. Lai, W. K. Orr, and K. F. Shortridge. 1999. Human infection with influenza H9N2. Lancet 354:916-917
129. Eric H.Y. Lau, Y.H. Connie Leung, Li Juan Zhang, Benjamin J. Cowling,Sin Ping Mak, Yi Guan, Gabriel M. Leung, and J. S. Malik Peiris. Effect of Interventions on Infl uenza A (H9N2) Isolation in Hong KongsLive Poultry Markets, 1999–2005
130. Van Beest Holle MDR, Meijer A, Koopmans M et al. Human-to-human transmission of avian influenza A/H7N7, the Netherlands, 2003. Euro Surveill. 2005;10(12):264-8
131. Fouchier RAM, Schneeberger PM, Rozendaal FW et al. Avian influenza A virus (H7N7) associated with human conjuntivitis and fatal case of acute respiratory distress síndrome. Proc Natl Acad Sci USA 2004; 101:1356-1361.
132. Human Cases of Avian Influenza A (H7N7) Infection - The Netherlands, 2003. Centers for Disease Control and Prevention. Accesado el 19/05/09 desde http://www.cdc.gov/flu/avian/h7n7-netherlands.htm
133. A. R W. Elbers, T. H F. Fabri, T. S. de Vries, J. J. de Wit, A. Pijpers, and G. Koch.The Highly Pathogenic Avian Influenza A (H7N7) Virus Epidemic in the Netherlands in 2003Lessons Learned from the First Five Outbreaks. Avian Diseases 48(3):691-705. 2004
134. Marion Koopmans, Berry Wilbrink, Marina Conyn, et alTransmission of H7N7 avian influenza A virus to human beings during a large outbreak in commercial poultry farms in the NetherlandsThe Lancet, 363(9409): 587-593
135. Tweed SA, Skowronski DM, David ST, et al. Human illness from avian influenza H7N3, British Columbia. Emerg Infect Dis 2004;10:2196–2199.
136. Marlo Libel. Emerging and Reemerging Infectious Diseases, Region of the Americas. Vol. 2, No. 187 May 2004. http://www.paho.org/English/AD/DPC/CD/eid-eer-07-may-2004.htm
137. Myers KP, Olsen CW, Gray GC. Cases of swine influenza in humans: a review of the literature. Clin Infect Dis. 2007;44(8):1084-8.
138. Kluska V, Macku M, Mensik J. Demon. stration of antibodies against swine influenza viruses in man. Cesk Pediatr 1961;16:408-14.
139. Joel C. Gaydos, Franklin H. Top, Jr, Richard A. Hodder, and Philip K. Russell. Swine Influenza A Outbreak, Fort Dix, New Jersey, 1976. Emerging Infectious Diseases 2006;12(1): 23-28
140. Thompson, R. L., M. A. Sande, R. P. Wenzel et al. Swine influenza infection in civilians: Report of two cases 1976. Submitted for publication. N EngI J Med 295(714-715). 1976.
141. Smith T.F. Burgert E.O, Dowdle W.R, Noble G.R and Van Scoy R.E. Isolation of swine influenza virus from autopsy lung tissue of man. N Eng J Med 1976; 294:708
142. U.S. Public Health Service, Center for Disease Control: Influenza Morbidity and Mortality weekly report: En: Morbidity ond Mortality Weekly Rep 2X6-15). 1976.
143. Michael A. W. Hattwick, Richard J. OBrien,Charles H. Hoke and Walter R. Dowdle. La influenza pandemica y el virus de la influenza porcina. Bol Ofic San Panam 1977; 120-131
144. Writing Committee of the Second World Health Organization Consultation on Clinical Aspects of Human Infection with Avian Influenza A (H5N1) Virus, Abdel-Ghafar AN, Chotpitayasunondh T, Gao Z, Hayden FG, Nguyen DH, et al. Update on avian influenza A (H5N1) virus infection in humans. N Engl J Med. 2008 17;358(3):261-73.
145. Influenza team (ECDC). Low Pathogenicity Avian Influenzas and human health. Euro Surveill. 2007;12(22):pii=3209. Available from: http://www. eurosurveillance.org/ViewArticle.aspx?ArticleId=3209
146. Hai Yu, Rong-Hong Hua, Qiang Zhang, Tian-Qiang Liu, Hui-Li Liu,Guo-Xin Li, and Guang-Zhi Tong.Genetic Evolution of Swine Influenza A (H3N2) Viruses in China from 1970 to 2006. 2008, J Clin Microbiol 46(3) p. 1067–1075
147. Webby, R. J., S. L. Swenson, S. L. Krauss, P. J. Gerrish, S. M. Goyal, and 1074 YU ET AL. J. CLIN. MICROBIOL. R. G. Webster. 2000. Evolution of swine H3N2 influenza viruses in the United States. J. Virol. 74:8243–8251.
148. Zhou, N. N., D. A. Senne, J. S. Landgraf, S. L. Swenson, G. Erickson, K. Rossow, L. Liu, K. J. Yoon, S. Krauss, and R. G. Webster. 1999. Genetic reassortment of avian, swine, and human influenza A viruses in American pigs. J. Virol. 73:8851–8856.
149. A I Karasin, C W Olsen, I H Brown, S Carman, M Stalker, and G Josephson. H4N6 influenza virus isolated from pigs in Ontario. Can Vet J. 2000 December; 41(12): 938–939
150. Zhou NN, Senne DA, Landgraf JS, Swenson SL, Erickson G, Rossow K, et al. Genetic reassortment of avian, swine, and human influenza A viruses in American pigs. J Virol. 1999;73:8851–6.
151. Karasin AI, Schutten MM, Cooper LA, Smith CB, Subbarao K, Anderson GA, et al. Genetic characterization of H3N2 influenza viruses isolated from pigs in North America, 1977-1999: evidence for wholly human and reassortant virus genotypes. Virus Res. 2000;68:71–85.
152. Webby RJ, Swenson SL, Krauss SL, Gerrish PJ, Goyal SM, Webster RG. Evolution of swine H3N2 influenza viruses in the United States. J Virol. 2000;74:8243–51.
153. Christopher W. Olsen, Alexander I. Karasin,Suzanne Carman Et al. Triple Reassortant H3N2 Influenza A Viruses, Canada, 2005. Emerging Infectious Diseases 2006; 12(7): 1132-1135
154. Garten RJ, Davis CT, Russell CA .Antigenic and Genetic Characteristics of Swine-Origin 2009 A(H1N1) Influenza Viruses Circulating in Humans. Science. 2009 May 22. [Epub ahead of print] PMID: 19465683 [PubMed - as supplied by publisher]
155. Olsen CW. Emergence of novel strains of swine influenza virus in North America. In: Morilla A, Yoon K-J, Zimmerman JJ, editors. Trends in emerging viral infections of swine. Ames (IA): Iowa State University Press; 2002. p. 37–43.
156. Karasin AI, Schutten MM, Cooper LA, Smith CB, Subbarao K, Anderson GA, Carman S, Olsen CW.Genetic characterization of H3N2 influenza viruses isolated from pigs in North America, 1977-1999: evidence for wholly human and reassortant virus genotypes. Virus Res. 2000 Jun;68(1):71- 85
157. Vincet AL, Lager KM, MA W, Lekcharoensuk P, Gramer MR, Loiacono C et al. Evaluation of hemagglutinin subtype 1 swine influenzaviruses from the Uited States. Veterinary Microbiology. 2006/12/20;118(3-4):212-22.
158. Brown IH, Harris PA, McCauley JW, Alexander DJ. Multiple genetic reassortment of avian and human influenza A viruses in European pigs, resulting in the emergence of an H1N2 virus of novel genotype. J Gen Virol. 1998;79 ( Pt 12):2947-55
159. Shinde V, Bridges CB, Uyeki TM, et al. Triple-reassortant swine influenza A (H1) in humans in the United States, 2005– 2009. N Engl J Med 2009;360:2616-25.
160. Shinde V, Bridges CB, Uyeki TM, et al. Triplereassortant swine influenza A (H1) in humans in the United States, 2005– 2009. N Engl J Med 2009;360:2616-25.
161. Vivek Shinde, Carolyn B. Bridges, Timothy M. Uyeki et al. Triple-Reassortant Swine Influenza A (H1) in Humans in the United States, 2005–2009. N Engl J Med 2009; 360: 25
162. Newman AP, Reisdorf E, Beinemann J, et al. Human case of swine influenza A (H2N1) triple reassortant virus infection, Wisconsin. Emerg Infect Dis 2008;14: 1470-1472.
163. Brown, I. H. 2000. The epidemiology and evolution of influenza viruses in pigs. Vet. Microbiol. 74:29–46.
164. Pensaert, M., K. Ottis, J. Vandeputte, M. M. Kaplan, and P. A. Bachmann. 1981. Evidence for the natural transmission of influenza A virus from wild ducks to swine and its potential importance for man. Bull. W. H. O. 59:75–78.
165. Scholtissek, C., H. Burger, P. A. Bachmann, and C. Hannoun. 1983. Genetic relatedness of hemagglutinins of the H1 subtype of influenza A viruses isolated from swine and birds. Virology 129:521–523.
166. Olsen, C. W., I. H. Brown, B. C. Easterday, and K. Van Reeth. 2006. Swine influenza, p. 469–482. In B. E. Straw, J. J. Zimmerman, S. DAllaire, and D. J. Taylor (ed.), Diseases of swine, 9th ed. Blackwell Publishing Professional, Ames, IA.
167. Hai Yu, Yan-Jun Zhou, Guo-Xin Li, Gui-Hong Zhang, Hui-Li Liu, Li-Ping Yan, Ming Liao, Guang-Zhi Tong. Further evidence for infection of pigs with human-like H1N1 influenza viruses in China. Virus Research 140 (2009) 85–90
168. Lee CS, Kang BK, Kim HK, Park SJ, Park BK, Jung K, Song DS. Phylogenetic analysis of swine influenza viruses recently isolated in Korea.Virus Genes. 2008;37(2):168-76.
169. Jung K, Song DS.Evidence of the co-circulation of influenza H1N1, H1N2 and H3N2 viruses in the pig population of Korea.Vet Rec. 2007 Jul 21;161(3):104-5.
170. Maldonado J, Van Reeth K, Riera P, Sitjà M, Saubi N, Espuña E, Artigas .Evidence of the concurrent circulation of H1N2, H1N1 and H3N2 influenza A viruses in densely populated pig areas in Spain. C.Vet J. 2006 Sep;172(2):377- 81.
171. Guan, Y., K. F. Shortridge, S. Krauss, P. H. Li, Y. Kawaoka, and R. G. Webster. 1996. Emergence of avian H1N1 influenza viruses in pigs in China. J. Virol. 70:8041– 8046.
172. Yu H, Zhang PC, Zhou YJ, Li GX, Pan J, Yan LP, Shi XX, Liu HL, Tong GZ. Isolation and genetic characterization of avian-like H1N1 and novel ressortant H1N2 influenza viruses from pigs in China. Biochem Biophys Res Commun. 2009.
173. Chen Y, Meng X, Liu Q, Huang X, Huang S, Liu C, Shi K, Guo J, Chen F, Hu L. Phylogenetic analysis of human/ swine/avian gene reassortant H1N2 influenza A virus isolated from a pig in China. Wei Sheng Wu Xue Bao. 2008 Apr 4;48(4):466-72
174. Karasin, A. I., M. M. Schutten, L. A. Cooper, C. B. Smith, K. Subbarao, G. A. Anderson, S. Carman, and C. W. Olsen. Genetic characterization of H3N2 influenza viruses isolated from pigs in North America, 1977–1999: evidence for wholly human and reassortant virus genotypes. Virus Res 2000. 68:71–85.
175. Ma, W., A. L. Vincent, M. R. Gramer, C. B. Brockwell, K. M. Lager, B. H. Janke, P. C. Gauger, D. P. Patnayak, R. J. Webby, and J. A. Richt. 2007. Identification of H2N3 influenza A viruses from swine in the United States. Proc. Natl. Acad. Sci. USA 104:20949–20954.
176. Karasin AI, Olsen CW, Brown IH, Carman S, Stalker M, Josephson G. H4N6 influenza virus isolated from pigs in Ontario. Can Vet J. 2000 Dec;41(12):938-9.
177. Song DS, Lee CS, Jung K, Kang BK, Oh JS, Yoon YD, Lee JH, Park BK. Isolation and phylogenetic analysis of H1N1 swine influenza virus isolated in Korea. Virus Res. 2007 Apr;125(1):98-103.
178. Eleca J. Dunham, Vivien G. Dugan, Emilee K. Kaser, Sarah E. Perkins,Ian H. Brown, Edward C. Holmes,and Jeffery K. Taubenberger. Different Evolutionary Trajectories of European Avian-Like and Classical Swine H1N1 Influenza A Viruses. J Virol 2009; 83(11): 5485-5494
179. Nelson MI, Viboud C, Simonsen L, et al. Multiple reassortment events in the evolutionary history of H1N1 influenza virus since 1918. PLoS Pathog 2008;4(2): e1000012.
180. H1N1 Flu (Swine Flu).Center of Control Diseases. http://www.cdc.gov/h1n1flu/. Accesado 19/05/09
181. Christophe Fraser, Christl A. Donnelly, Simon Cauchemez, Et al. Pandemic Potential of a Strain of Influenza A (H1N1): Early Findings. Science. 2009 May 14. [Epub ahead of print] PMID: 19433588 [PubMed - as supplied by publisher]
182. Realizan barrido sanitario por foco de infección en Perote, Veracruz», Notimex, SDP Noticias, 5 de abril de 2009. http://sdpnoticias.com/sdp/contenido/2009/04/05/369612 Consultado el 06-66-2009. (en español)
183. Smithfield Foods - Smithfield Foods Says It Found No Evidence of Swine Influenza at Its Mexican Joint Ventures, http://investors.smithfieldfoods.com/releasedetail.cfm?ReleaseID=379761
184. CDC. Update: Swine Influenza A (H1N1) Infections - California and Texas, April 2009. MMWR Morb Mortal Wkly Rep. 2009 May 1;58(16):435-7.
185. David Brown. «New Strain of Swine Flu Investigated: Two Children in San Diego Area Had No Contact with Pigs», Washington Post, 22 de abril de 2009. http://www.washingtonpost.com/wp-dyn/content/article/2009/04/21/AR2009042103694.html
186. World Health Organization. Swine flu illness in the United States and Mexico - update 2. Geneva: World Health Organization;2009 [updated 26 Apr 2009]; Available from: http://www.who.int/csr/don/2009_04_26/es/index.html.
187. OMS. Reglamento sanitario internacional (2005). 2° ed. Ginebra: Organización Mundial de la Salud; 2008.
188. Cohen J. Swine flu outbreak. Out of Mexico? Scientists ponder swine flus origins. Science. 2009 May 8;324(5928):700-2.
189. WHO Global Influenza Programme, World Health Organization. Pandemic influenza preparedness and response. Geneva: World Health Organization; 2009.
190. Organización Mundial de la salud. Declaración de la Directora General de la OMS a la prensa 11 de junio de 2009 . http://www.who.int/mediacentre/news/statements/2009/h1n1_pandemic_phase6_20090611/es/print.html
191. World Health Organization. Influenza A(H1N1) - update 41. Geneva: World Health Organization; 2009 [updated 17 May 2009]; Available from: http://www.who.int/csr/don/2009_05_29/en/index.html
192. Ministerio de Salud. Comunicado Oficial Nº 13: sobre situación de nueva influenza a H1N1. Oficina Ejecutiva de Prensa. Lima, 15 de Mayo del 2009.
193. MINSA. Comunicado Oficial Nº 85: sobre la situación nueva influenza AH1N1. 31 de julio del 2009 . http://www.minsa.gob.pe/portada/prensa/notas_auxiliar.asp?nota=7676
194. Research on Complex Systems Engineering Sciences and Applied Mathematics, Northwestern University. Computational quantitative projections for H1N1 flu dynamics in the United States june 2009. http://rocs.northwestern.edu/projects/swine_flu/#WCS
195. The research group of Alessandro Vespignani at Indiana University. Mexican Flu projections: worldwide report updated to May 16 2009. GLEaMviz.org. http://www.gleamviz.org/2009/05/mexican-flu-projections-worldwidereport-updated-to-may-16/
196. Andrew Rambaut, Oliver G. Pybus, Martha I. Nelson, Cecile Viboud, Jeffery K. Taubenberger & Edward C. Holmes. The genomic and epidemiological dynamics of human influenza A virus.Nature 2008;453: 615-618.
197. Colin A. Russell, Terry C. Jones, Ian G. Barr et al. The Global Circulation of Seasonal Influenza A (H3N2) Viruses. Science 2008; 230: 340-346
198. L. Simonsen et al., Mol. Biol. Evol. 24, 1811 (2007).
199. N. J. Cox, K. Subbarao, Annu. Rev. Med. 51, 407(2000).
200. Wladimir J. Alonso, Ce´ cile Viboud, Lone Simonsen, Eduardo W. Hirano, Luciane Z. Daufenbach, and Mark A. Miller. Seasonality of Influenza in Brazil: A Traveling Wave from the Amazon to the Subtropics. American Journal of Epidemiology 2007; 165 (12): 1434-1442
201. Li-Qun Fang1,2., Sake J. de Vlas2., Song Liang3.,Et al. Environmental Factors Contributing to the Spread of H5N1 Avian Influenza in Mainland China. PLoS ONE 2088; 3(5): e2268
202. Shek LP, Lee BW. Epidemiology and seasonality of respiratory tract virus infections in the tropics. Paediatr Respir Rev 2003; 4: 105–11.
203. Nicolas Gaidet, Tim Dodman, Alexandre Caron,Avian Influenza. Viruses in Water Birds, Africa. Emerging Infectious Diseases 2007; 13(4): 626-629
204. Krauss S, Walker D, Pryor SP, Niles L, Chenghong L, Hinshaw VS, et al. Influenza A viruses of migrating wild aquatic birds in North America. Vector Borne Zoonotic Dis. 2004;4:177–89.
205. The role of environmental transmission in recurrent avian influenza epidemics.Breban R, Drake JM, Stallknecht DE, Rohani P. PLoS Comput Biol. 2009 Apr;5(4):e1000346
206. Harder B (2005) When flu flies the coop: A pandemic threatens. Science News 168: 171.
207. Andrew S. Lang, Anke Kelly and Jonathan A. Runstadler. Prevalence and dive/ Week 18 ending May 9, 2009 Center of Control Diseases. http://www.cdc.gov/flu/weekly/pdf/External_F0918.pdf. Accesado 19/05/09
208. Runstadler, J. A., Happ, G. M., Slemons, R. D., Sheng, Z. M., Gundlach, N., Petrula, M., Senne, D., Nolting, J., Evers, D. L. & other authors (2007). Using RRT-PCR analysis and virus isolation to determine the prevalence of avian influenza virus infections in ducks at Minto Flats State Game Refuge, Alaska, during August 2005. Arch Virol 152, 1901–1910.
209. Hoyo JD, Elliot A, Sartagal J (1996) Hoatzin to Auks. Handbook of the birds of the World. Barcelona: Lynx Edicions. Vols. 1 and 3.
210. Olsen B, Munster VJ, Wallensten A, Waldenstrom J, Osterhaus ADME, et al. (2006) Global patterns of influenza A virus in wild birds. Science 312: 384–388.
211. Fouchier R, Osterhaus A, Brown IH.Animal influenza virus surveillance. Vaccine 2003; 21:1754-1757
212. Horimoto T, Kawaoka Y. Pandemic threat posed by avian influenza A virases. Clinical Microbiology Reviews 2001;14(1):129-149.
213. Zeitlin GA, Maslow MJ. Avian influenza. Curr Infect Dis Rep 2005;7:193-199.
214. Fauci AS. Emerging and re-emerging infectious diseases : Influenza as a prototype of the host-pathogen balancing act. Cell 2006;124(4):665-670
215. Vahlenkamp T.W and Harder T.C. Influenza virus infections in mammals. Berl Mnch Tierärztl Wochenschr 2006; 119(3-4):123-131
216. B r o w C . Av i a n i n f l u e n z a . A m J P a t h o l . 2006;168(1):6-8.
217. Suzuki Y. Sialobiology of influenza: molecular mechanism of host range variation of influenza viruses. Biol Pharm Bull. 2005;28(3):399-408.
218. Stallknecht DE, Kearney MT, Shane SM, Zwank PJ. Effects of pH, temperature, and salinity on persistence of avian influenza viruses in water. Avian Dis. 1990 Apr- Jun;34(2):412-8
219. Brown JD, Swayne DE, Cooper RJ, Burns RE, Stallknecht DE.Persistence of H5 and H7 avian influenza viruses in water. Avian Dis. 2007 Mar;51(1 Suppl):285-9.
220. Brown JD, Goekjian G, Poulson R, Valeika S, Stallknecht DE.Avian influenza virus in water: infectivity is dependent on pH, salinity and temperature.Vet Microbiol. 2009 Apr 14;136(1-2):20-6.
221. Stallknecht DE, Shane SM, Kearney MT, Zwank PJ. Persistence of avian influenza virusesin water. Avian Dis. 1990 Apr-Jun;34(2):406-11
222. Brown JD, Goekjian G, Poulson R, Valeika S, Stallknecht DE.Avian influenza virus in water: infectivity is dependent on pH, salinity and temperature. Vet Microbiol. 2009 Apr 14;136(1-2):20-6.
223.
224. Webster, R. G., Bean, W., Gorman, O., Chambers, T. & Kawaoka, Y. Evolution and ecology of influenza A viruses. Microbiol Rev 1992; 56, 152–179.
225. Zhang, G., Shoham, D., Gilichinsky, D., Davydov, S., Castello, J. D. & Rogers, S. O. (2006). Evidence of influenza A virus RNA in Siberian lake ice. J Virol 80, 12229–12235.
226. Ito, T., Okazaki, K., Kawaoka, Y., Takada, A., Webster, R. G. & Kida, H. (1995). Perpetuation of influenza A viruses in Alaskan waterfowl reservoirs. Arch Virol 140, 1163–1172.
227. Rohani P, Breban R, Stallknecht DE, Drake JM. Environmental transmission of low pathogenicity avian influenza viruses and its implications for pathogen invasion. Proc Natl Acad Sci U S A. 2009; Early Edition 1 of 5. www.pnas.org/cgi/doi/10.1073 / pnas.0809026106.
228. Vivien G. Dugan, Rubing Chen, David J. Spiro. The Evolutionary Genetics and Emergence of Avian Influenza Viruses in Wild Birds. PLoS Pathogens 208; 4(5): e1000076
229. Yves Thomas, Guido Vogel, Werner Wunderli, Patricia Suter, Mark Witschi, Daniel Koch, Caroline Tapparel, and Laurent Kaiser.Survival of Influenza Virus on Banknotes. Applied and Environmental Microbiology, 2008, 74(10): 3002–3007
230. Tellier, R. 2006. Review of aerosol transmission of influenza A virus. Emerg. Infect. Dis. 12:1657–1662.
231. Weber TP, Stilianakis NI. Inactivation of influenza A viruses in the environment and modes of transmission: a critical review.J Infect. 2008 Nov;57(5):361-73
232. Bruno M. Ghersi, David L. Blazes, Eliana Icochea, Et al. Avian Influenza in Wild Birds, Central Coast of Peru. Emerging Infectious Diseases . 2006; 15(9):935-938
233. Vahlenkamp T.W and Harder T.C. Influenza virus infections in mammals. Berl Mnch Tierärztl Wochenschr 2006; 119(3-4):123-131
234. Rimmelzwaan GF, de Jong JC, Bestebroer TM, et al. Antigenic and genetic characterization and swin influenza A (H1N1) virases isolated from pneumonia patients in The Netherlands. Virology. 2001;282:301-306.
235. Claas, E.C. et al. (1994) Infection of children with avian-human reassortant influenza virus from pigs in Europe. Virology 204, 453–457
236. Berrios E., Patricio. Influenza equina en Chile (1963 - 1992): Un posible caso en un ser humano. Rev. chil. infectol. [online]. 2005, 22(1):. 47-50.
237. Jukius A. Kasel, Robert H. Alford, Vernon Knight, Et al. Experimental Infection of Human Volunteers with Equine Influenza Virus Nature 1965; 206(4979), 41-43
238. Renegar KB. Influenza virus infections and immunity: a review of human and animals models. Lab Anim Sci. 1992;42:222-232a.
239. Smith H, C Sweet. Lessons for human influenza from pathogenicity studies with ferrets. Rev Infect Dis. 1988;10: 56-75.
240. Govorkova EA, Rehg JE, Krauss S, Yen HL, Et al. Lethality to ferrets of H5N1 influenza viruses isolated from humans and poultry in 2004. J Virol. 2005 Feb;79(4):2191-8
241. P. Marois, A. Boudreault, E. DiFranco, and V. Pavilanis. Response of Ferrets and Monkeys to Intranasal Infection with Human, Equine and Avian Influenza Viruses. Can J Comp Med. 1971 January; 35(1): 71–76.
242. Horimoto T, Kawaoka Y. Pandemic threat posed by avian influenza A virases. Clinical Microbiology Reviews 2001;14(1):129-149.
243. Chan PK. Outbreak of avian influenza A (H5N1) virus infection in Hong Kong in 1997. Clin Infect Dis. 2002;34(Suppl 2):S58-64.
244. Peiris JSM, Yu WC, Leung CW, et al. Re-emergence of fatal human influenza A subtype H5N1 disease. Lancet. 2004;363:617-619.
245. WHO. Advice on the use of masks1 in the community setting in Influenza A (H1N1) outbreaks Interim guidance. 2009.
246. N.F. Phin, A.J. Rylands, J. Allan, C. Edwards, J.E. Enstone, J.S. Nguyen-Van-Tam.Personal protective equipment in an influenza pandemic: a UK simulation exercise. Journal of Hospital Infection 2009 71, 15-21
247. Centers for Disease Control and Prevention, USA. Guidelines and recommendations.Interim Antiviral Guidance for 2008-09. http://www.cdc.gov/flu/professionals/antivirals/index.htm
248. Centers for Disease Control and Prevention, USA. Guidelines and recommendations. Infection Control Guidance for the Prevention and Control of Influenza in Acute-Care Facilities. http://www.cdc.gov/flu/professionals/infectioncontrol/healthcarefacilities.htm
249. Srinivasan A, Viswanathan K, Raman R, Chandrasekaran A, Raguram S, Tumpey TM, et al. Quantitative biochemical rationale for differences in transmissibility of 1918 pandemic influenza A viruses. Proc Natl Acad Sci USA 2008;105: 2800–5.
250. Hayden F, Croisier A. Transmission of avian influenza viruses to and between humans. J Infect Dis 2005;192:1311–4.
251. US Department opf Healht & Human Services. HHS Pandemic Influenza Plan Supplement 4 Infection Control. http://www.hhs.gov/pandemicflu/plan/sup4.html
252. Brankston, G., L. Gitterman, Z. Hirji, C. Lemieux, and M. Gardam..Transmission of influenza A in human beings. Lancet Infect. Dis. 2007; 7:257–265.
253. Mubareka S, Lowen AC, Steel J, Coates AL, García- Sastre A, Palese P.Transmission of influenza virus via aerosols and fomites in the guinea pig model.J Infect Dis. 2009 Mar 15;199(6):858-65
254. Ann H. Reid, Thomas A. Janczewski, Raina M. Lourens Et al. 1918 Influenza Pandemic Caused by Highly Conserved Viruses with Two Receptor-Binding Variants. Emerging Infectious Diseases 2003; 9(10): 1251 -1253
255. Bean B, Moore BM, Sterner B, Peterson LR, Gerding DN, Balfour HH Jr.Survival of influenza viruses on environmental surfaces.J Infect Dis. 1982 Jul;146(1):47-51
256. Cécile Viboud, Mark A. Miller, Bryan T. Grenfell, Ottar N. Bjørnstad, Lone Simonsen Air Travel and the Spread of Infl uenza: Important Caveats. PLoS Medicine 2006 (3) 11: 2159-2161
257. Brownstein JS, Wolfe CJ, Mandl KD. Empirical evidence for the effect of airline travel on inter-regional inûuenza spread in the United States. PLoS Med 2006 3(10): 1826- 1835.
258. V. Colizza, A. Barrat, M. Barthelemy, A. Vespignani. The role of the airline transportation network in the prediction and predictability of global epidemics.Proc. Natl. Acad. Sci. 2006; 103: 2015-2020
259. Cooper BS, Pitman RJ, Edmunds WJ, Gay NJ (2006) Delaying the international spread of pandemic influenza. PLoS Med 2006; 3(6): 845-855.
260. Ferguson NM, Cummings DA, Fraser C, Cajka JC, Cooley PC, et al. Strategies for mitigating an infl uenza pandemic. Nature 2006; 442: 448–452.
261. Germann TC, Kadau K, Longini IM Jr, Macken CA. Mitigation strategies for pandemic influenza in the United States. Proc Natl Acad Sci USA 2006; 103: 5935–5940.
262. Helen J. Wearing, Pejman Rohani, Matt J. Keeling. Appropriate Models for the Management of Infectious Diseases. PLoS Medicine 2005; 2(7): e174
263. Jefferson T, Foxlee R, Del Mar C et al. Physical interventions to interrupt or reduce the spread of respiratory viruses: systematic review. BMJ 2008; 336;77-80.
264. Marianne van der Sande, Peter Teunis, Rob Sabel. Professional and Home-Made Face Masks Reduce Exposure to Respiratory Infections among the General Population. PLoS ONE 2008 3(7): e2618
265. Ministerio de Salud Guia Tecnica: Guia de Prática Clinica para el diagnóstico y tratamiento de influenza por virus A H1N1, 2009. aprobado por R.M. Nº 326-2009/MINSA (R.M. Nº 352-2009/MINSA) El Peruano, 30/05/2009; Pág. 396716
266. WHO . Recomendación provisional para el tratamiento con antivirales de los pacientes confirmados o sospechosos de estar infectados por el nuevo subtipo del virus influenza A(H1N1). 30 de abril 2009. http://new.paho.org/hq/index.php?option=com_docman&task=doc_download&gid=1392&Itemid=1392
267. Sistema Nacional de Información en Salud de Mexico. www.sinais.salud.gob.mx http://sinais.salud.gob.mx/descargas/pdf/influenza/influenza_porcina_drtena.pdf
268. Directiva Nº 026-MINSA/DGE-V.01. Directiva sanitária para La vigilancia epidemiológica intensificada de influenza e infecciones respiratórias agudas (IRA) em el Perú . Resolucion Ministerial Nº 455-2009, MINSA 07 de julio del 2009.
269. Ministerio de Salud –Estrategia Sanitaria Nacional de Control de Inmunizaciones. http://www.minsa.gob.pe/portada/prevencion.htm
270. Ghersi BM, Blazes DL, Icochea E, Gonzalez RI, Kochel T, Tinoco Y, et al. Avian influenza in wild birds, central coast of Peru. Emerg Infect Dis [serial on the Internet]. 2009 Jun [date cited]. Available from http://www.cdc.gov/EID/content/15/6/935.htm
271. MINSA: Dirección General de Epidemiología Análisis de Situación de Salud del Perú 2005(ASIS Perú). Disponible en. http://www.dge.gob.pe/publicaciones/pub_asis/asis15.pdf
272. Pueblos en Situación de Extrema vulnerabilidad, el Caso de los Nanti de la reserva territorial Kugapakori – Nahua del Río Camisea Cusco (ASIS NANTI) http://www.dge.gob.pe/publicaciones/pub_asis/asis12.pdf
273. MINSA: Plan nacional de preparación y respuesta frente a una potencial pandemia de influenza. Disponible en: http://www.dge.gob.pe/aviar/PDF/Plan_Influenza_Peru.pdf
274. OMS. Plan de preparación para la pandemia de influenza El Rol de la Organización Mundial de la Salud y Guías para la Planificación Nacional y Regional Disponible en: http://www.who.int/csr/resources/publications/influenza/whoplanspanish.pdf
275. Estrategia comunitaria para mitigar la influenza pandémica, disponible en http://espanol.pandemicflu.gov/pandemicflu/enes/24/_pandemicflu_gov/plan/community/commitigation.html
276. Longini IM, Jr., Nizam A, Xu S, Ungchusak K, Hanshaoworakul W, Cummings DA, et al. Contención de la influenza pandémica en la fuente. Science. 2005 Agos 12;309(5737):1083
277. OMS. Declaración de la Fase 6 de la Pandemia por la OMS. Disponible en : http://www.who.int/mediacentre/news/statements/2009/h1n1_pandemic_phase6_20090611/es/index.html
278. OMS: Reglamento Sanitario Internacional. http://www.who.int/csr/ihr/es/
279. Bell DM. Public Health Interventions and SARS Spread, 2003. Emerging Infectious Diseases Vol. 10, No. 11, November 2004. http://www.cdc.gov/ncidod/EID/vol10no11/04-0729.htm
280. Bell DM. Non-pharmaceutical interventions for pandemic influenza, national and community measures. Emerg Infect Dis. 2006 Jan;12(1):88-94. http://www.cdc.gov/ncidod/EID/vol12no01/pdfs/05-1371.pdf
281. Ball F, Britton T, Lyne O. Stochastic multitype epidemics in a community of households: estimation of threshold parameter R-* and secure vaccination coverage. Biometrika. 2004;91:345-62.
282. Heesterbeek JA. A brief history of R0 and a recipe for its calculation. Acta Biotheor. 2002;50(3):189-204.
283. Sanchez MA, Blower SM. Uncertainty and sensitivity analysis of the basic reproductive rate. Tuberculosis as an example. Am J Epidemiol. 1997 Jun 15;145(12):1127
284. Mujica Oscar Conferencia de frente a las Autoridades de Salud Mexicana como Analista Senior de la OPS. http://portal.salud.gob.mx/contenidos/noticias/influenza/conferencias/conferencia_prensa_080509_mat_02.html
285. Glass and Glass. Social contact networks for the spread of pandemic influenza in children and teenagers BMC Public Health 2008 8:61 doi:10.1186/1471-2458-8-61
286. Glass RJ. Targeted social distancing design for pandemic influenza. Emerg Infect Dis. 2006;12:1671-81.
287. Bootsma MC, Ferguson NM: The effect of public health measures on the 1918 influenza pandemic in U.S. cities. PNAS 2007, 18:7588-93.
288. Hatchett RJ, Mecher CE, Lipsitch M: Public health interventions and epidemic intensity during the 1918 influenza pandemic. PNAS 2007, 18:7582-7.
289. Vynnycky E, Edmunds WJ: Analyses of the 1957 (Asian) influenza pandemic in the United Kingdom and the impact of school closures. Epidemiol Infect 2007:1-14.
290. Heymann A, Chodick G, Reichman B, Kokia E, Laufer J: Influence of school closure on the incidence of viral respiratory diseases among children and on health care utilization
291. Institute of Medicine: Modeling Community Containment for Pandemic Influenza-A Letter Report. The National Academy Press; 2006.
292 Milne GJ, Kelso JK, Kelly HA, Huband ST, McVernon J. A small community model for the transmission of infectious diseases: comparison of school closure as an intervention in individual-based models of an influenza pandemic. PLoS One. 2008;3(12):e4005.
293. Aledort J, Lurie N, Wasserman J, Bozzette SA: Nonpharmaceutical public health interventions for pandemic influenza: an evaluation of the evidence base. BMC Public Health 2007, 7:208.
294. Novel Influenza A (H1N1) Virus Infections in Three Pregnant Women United States, AprilMay 2009 MMWR May 15, 2009 / 58(18);497-500 http://www.cdc.gov/mmwr/preview/mmwrhtml/mm5818a3.htm
295. MINSA. Dirección General de Epidemiología – Vigilancia Epidemiológica de la Influenza. http://www.dge.gob.pe/influenza/AH1N1/
296. Cox NJ, Subbarao K. Influenza. Lancet 1999;354:1277– 82.
297. John C. Obenauer, Jackie Denson, Perdeep K. Mehta Et al. Large-Scale Sequence Analysis of Avian Influenza Isolates. Science 2006; 311(): 1576-1580
298. R. J. Cox, K. A. Brokstady & P. Ograz. Influenza Virus: Immunity and Vaccination Strategies. Comparison of the Immune Response to Inactivated and Live, Attenuated Influenza Vaccines Scandinavian Journal of Immunology, Volume 59, Issue 1 (p 1-15)
299. Zeitlin GA, Maslow MJ. Avian influenza. Curr Infect Dis Rep 2005;7:193-199.
300. Lisa FP Ng, Ian Barr, Tung Nguyen et al. Specific detection of H5N1 avian influenza A virus in field specimens by a one-step RT-PCR assay. BMC Infect Dis. 2006;6:40.
301. Dobrotvortseva VG, Milovidova NI. Type C influenza virus. Acta Virol. 1984;28(4): 334-42.
302. John M. Nicholls, Renee W.Y. Chan, Rupert J. Russell, Gillian M. Air and J.S. Malik Peiris.Evolving complexities of influenzavirus and its receptors. Trends in Microbiology Vol.16 No.4
303. N.M. Ferguson, A.P. Galvani and R.M. Bush. Ecological and immunological determinants of influenza evolution, Nature 2003 422(6930) : 428–433
304. Fodor, E. & Brownlee, G. G. in Influenza (ed. Potter, C. W.) 1–-29 (Elsevier, Amsterdam, 2002).
305. Naffakh, N., Massin, P., Escriou, N., Crescenzo- Chaigne, B. & van der Werf, S. Genetic analysis of the compatibility between polymerase proteins from human and avian strains of influenza A viruses. J. Gen. Virol. 2000; 81, 1283–-1291.
306. Chen, W. et al. A novel influenza A virus mitochondrial protein that induces cell death. Nature Med. 7, 1306–-1312 (2001).
307. Concern as revived 1918 flu virus kills monkeys. NATURE 2007; 445(7125): 237
308. Darwyn Kobasa, Steven M. Jones, Kyoko Shinya Et al. Aberrant innate immune response in lethal infection of macaques with the 1918 influenza virus. Nature 2007; 445(7124): 319–323
309. Fauci AS. Emerging and re-emerging infectious diseases: Influenza as a prototype of the host-pathogen balancing act. Cell 2006;124(4):665-670.
310. Shinya K, Ebina M, Yamada S, et al.Influenza receptors in the human airway.Nature. 2006;440(23):435-436.
311. van Riel D, Mumster VJ, de Wint E, et al.H5N1 virus attachment to coger respiratory trac. Sciencexpress 2006.;312(5772):399. www.sciencexpress.org
312. Connor RJ, Kawaoka Y, Webster RG, Paulson JC (1994) Receptor specificity in human, avian, and equine H2 and H3 influenza virus isolates. Virology 205: 17–23.
313. Gambaryan AS, Tuzikov AB, Piskarev VE, Yamnikova SS, Lvov DK, et al. (1997) Specification of receptor-binding phenotypes of influenza virus isolates from different hosts using synthetic sialylglycopolymers: non-egg-adapted human H1 and H3 influenza A and influenza B viruses share a common high binding affinity for 69-sialyl(Nacetyllactosamine). Virology 232: 345–350.
314. Matrosovich MN, Gambaryan AS, Teneberg S, Piskarev VE, Yamnikova SS, et al. (1997) Avian influenza A viruses differ from human viruses by recognition of sialyloligosaccharides and gangliosides and by a higher conservation of the HA receptor-binding site. Virology 233: 224–234.
315. Rogers GN, Paulson JC (1983) Receptor determinants of human and animal influenza virus isolates: differences in receptor specificity of the H3 hemagglutinin based on species of origin. Virology 127: 361–373.
316. Suzuki Y.Receptor sialylsugar chains as determinants of host range of influenza viruses. Nippon Rinsho. 2000 Nov;58(11):2206-10.
317. Lipatov AS, Kwon YK, Sarmento LV, Lager KM, Spackman E, Suarez DL, Swayne DE. Domestic pigs have low susceptibility to H5N1 highly pathogenic avian influenza viruses. PLoS Pathog. 2008 Jul 11;4(7):e1000102.
318. van Riel D, Mumster VJ, de Wint E, et al. H5N1 Virus Attachment to Lower Respiratory Tract. H5N1 virus attachment to coger respiratory trac. Science. 2006 Apr 21;312(5772):399
319. Shinya K, Kawaoka Y. Influenza virus receptors in the human airway. Uirusu. 2006 Jun;56(1):85-9
320. Debby van Riel, Vincent J. Munster, Emmie de Wit et al. Human and Avian Influenza Viruses Target Different Cells in the Lower Respiratory Tract of Humans and Other Mammals. The American Journal of Pathology,2007; 171( 4): 1215-1223
321. NichollsJM,BourneAJ,ChenH,GuanY,PeirisJS(200 7)S ialicacidreceptordetectioninthehumanrespiratorytra ct:evidenc eforwidespreaddistributionofpotentialbindin gsitesforhumanandavianinfluenzaviruses.RespirRes8:73.
322. NichollsJM,ChanMC,ChanWY,WongHK,Cheun gCY et al.(2007)TropismofavianinfluenzaA(H5N1)i ntheupperandlowerrespiratorytract.NatMed13:147–149.
323. T h o m p s o n C I , B a r c l a y W S , Z a m b o n M C , P i c k l esRJ(2006)Infection of humanairway epithelium by humanandavianstrainsofinfluen zaavirus.JVirol80:8060– 8068.
324. Matrosovich, M.N. et al. (2004) Human and avian influenza viruses target different cell types in cultures of human airway epithelium.Proc. Natl. Acad. Sci. U.S.A. 101, 4620–4624
325. MatrosovichMN, MatrosovichTY, GrayT, RobertsN A,KlenkHD(2004)Humanandavianinfluenzavirusestarget differentcelltypes inculturesofhumanairwayepithelium. ProcNatlAcadSciUSA101:4620–4624.
326. Tumpey TM, Maines TR, Van Hoeven N, Et al: A twoamino acid change in the hemagglutinin of the 1918 influenza virus abolishes transmission. Science 2007, 315:655–659
327. NatalieJ. McDonald, CatherineB.Smith and NancyJ. Cox.Antigenic drift in the evolution of H1N1 influenza A viruses resulting from deletion of a single amino acidin the haemagglutinin gene. Journal of General Virology (2007);88:3209–3213
328. Jeffery K Taubenberger.Influenza hemagglutinin attachment to target cells: birds do it, we do it Future Virol. 2006 ; 1(4): 415–418.
329. Guo, C.T. et al. (2007) The quail and chicken intestine have sialylgalactose sugar chains responsible for the binding of influenza A viruses to human type receptors. Glycobiology 17, 713–724
330. Gagneux P, Cheriyan M, Hurtado-Ziola N, et al. Humanspecific regulation of á2–6-linked sialic acids.J Biol Chem 2003;278(48):48245–48250.
331. Ito T, Couceiro JN, Kelm S. Molecular basis for the generation in pigs of influenza A viruses with pandemic potential. J Virol 1998;72(9):7367–7373. [PubMed: 9696833]
332. Wan H, Perez DR. Quail carry sialic acid receptors compatible with binding of avian and human influenza viruses. Virology 2006;346(2):278–286.
333. Thijs Kuikena, Jeffery K. Taubenberger.Pathology of human influenza revisited. Vaccine 26S (2008) D59–D66
334. van Riel D, Munster VJ, de Wit E, Rimmelzwaan GF, Fouchier RA, Osterhaus AD, et al. Human and avian influenza viruses target different cells in the lower respiratory tract of humans and other mammals. Am J Pathol 2007;171: 1215–23.
335. Kaiser L, Fritz RS, Straus SE, Guberava L, Hayden FG. Symptom patogénesis during acute influenza: interleukin-6 and other cytokine responses. J Med Virol. 2001;62(3):262- 268.
336. Hayden FG, Fritz R, Lobo MC, Alvord W, Strober W, Straus SE. Local and systemic cytokine responses during experimental human influenza A virus infection. Relation to symptom formation and host defense. J Clin Invest. 1998;101: 643-649.
337. Lee N, Wong CK, Chan PK Et al .Hypercytokinemia and hyperactivation of phospho-p38 mitogen-activated protein kinase in severe human influenza A virus infection. Clin Infect Dis. 2007 15;45(6):723-31.
338. Houde M, Arora DJ. Stimulation of tumor necrosis factor secretion by purified influenza virus neuraminidase. Cell Immunol. 1990;129:104-111.
339. Hofmann P, Sprenger H, Kaufmann A et al. Susseptibility of mononuclear phagocytes to influenza A virus infection and posible role in the antiviral response. J of Leuk Biology 1997; 66: 408-414.
340. Gong J-H, Sprenger H, Hinder F, et al. Influenza A virus infection of macrophages. Enhanced tumor necrosis factor- (TNF-) gene expression and lipopolysaccharide- triggered TNF- release. J Immunol. 1991;147:3507-3513.
341. Hartshorm KL, Reid KB, Jens MR, et al. Neutrophil deactivaction by influenza A virases: Mechanisms of protection alter viral opsonization with collectins and hemagglutinationinhibiting antibodies. Blood. 1996;87(8):3450-3461.
342. Gentile D, Doyle W, Whiteside T, Fireman P, Hayden FG, Skoner D. Increased interleukin-6 levels in nasal lavage samples following experimental influenza A virus infection. Clin Diagn Lab Immunol 1998; 5:604–8.
343. de Jong MD, Simmons CP, Thanh TT, et al. Fatal outcome of human influenza A (H5N1) is associated with high viral load and hypercytokinemia.Nat Med 2006; 12:1203–7.
344. de Jong M, Simmons CP, Thanh TT, et al. Fatal outcome of human influenza A (H5N1) is associated with high viral load and hypercytokinemia. Nat Med 2006;12: 1203-7.
345. Chan MC, Cheung CY, Chui WH, et al. Proinflammatory cytokine responses induced by influenza A (H5N1) viruses in primary human alveolar and bronchial epithelial cells. Respir Res 2005;6:135.
346. Aleksandr S. Lipatov, Samita Andreansky, Richard J. Webby Et al. Pathogenesis of Hong Kong H5N1 influenza virus NS gene reassortants in mice: the role of cytokines and B- and T-cell responses. J Gen Virol 2005, 86: 1121–1130
347. Seo SH, Hoffmann E, Webster RG. The NS1 gene of H5N1 influenza viruses circumvents the host anti-viral cytokine responses.Virus Res. 2004;103(1-2):107-13
348. van Riel D, Mumster VJ, de Wint E, et al.H5N1 virus attachment to coger respiratory trac. Sciencexpress 2006. www.sciencexpress.org
349. Guus F. Rimmelzwaan, Debby van Riel, Marianne Baars Et al.Influenza A Virus (H5N1) Infection in Cats Causes Systemic Disease with Potential Novel Routes of Virus Spread within and between Hosts. American Journal of Pathology 2006; 168 (1)1: 176-183
350. Uiprasertkul MP, Puthavathana K, Sangsiriwut P, Pooruk P, Srisook K, Peiris M, Nicholls JM, Chokephaibulkit K, Vanprapar N, Auewarakul P: Influenza A H5N1 replication sites in humans. Emerg Infect Dis 2005, 11:1036–1041
351. Gu J, Xie Z, Gao Z, et al. H5N1 infection of the respiratory tract and beyond: a molecular pathology study. Lancet 2007;370:1137-45.
352. Ng WF, To KF, Lam WW, Ng TK, Lee KC. The comparative pathology of severe acute respiratory syndrome and avian influenza A subtype H5N1-a review. Hum Pathol. 2006;37(4):381-90.
353. Lee DC, Cheung CY, Law AH, Mok CK, Peiris M, Lau AS: p38mitogen-activated protein kinase-dependent hyperinduction of tumornecrosis factor _ expression in response to avian influenza virusH5N1. J Virol 2005, 79:10147–10154
354. To KF, Chan PK, Chan KF, Lee WK, Lam WY, Wong KF, Tang NL, Tsang DN, Sung RY, Buckley TA, Tam JS, Cheng AF: Pathology of fatal human infection associated with avian influenza A H5N1 virus. J Med Virol 2001, 63:242–246
355. Margaret Chan. World now at the start of 2009 influenza pandemic. http://www.who.int/mediacentre/news/statements/2009/h1n1_pandemic_phase6_20090611/en/index.html
356. http://www.who.int/csr/disease/avian_influenza/guidelines/humanspecimens/en/index.html
357. http://www.who.int/csr/disease/avian_influenza/guidelines/transport/en/index.html
358. http://www.who.int/csr/disease/avian_influenza/guidelines/handlingspecimens/en/index.html
359. http://www.who.int/csr/resources/publications/swineflu/realtimeptpcr/en/index.html
360. OPS/OMS: Orientaciones para Laboratorios de Influenza para el diagnóstico de influenza porcina A/H1N1 25 de abril de 2009 http://www.mex.ops-oms.org/influenza/docs/doctecOrientaciones_para_Laboratorios_de_Influenza.pdf
361. WHO: CDC protocol of realtime RT-PCR for swine influenza A(H1N1). 28 April 2009, revision 1 30 april 2009http://new.paho.org/hq/index.phpoption=com_docman&task=doc_details&gid=1431&Itemid=99999999
362. Lau SK, Chan KH, Yip CC, Ng TK, et al. Confirmation of the First Hong Kong Case of Human Infection by Novel Swine Origin Influenza A (H1N1) Virus Diagnosed Using Ultrarapid, Real-Time Reverse Transcriptase PCR. J Clin Microbiol. 2009 Jul;47(7):2344-2346.
363. OMS, Evaluación de la gravedad de una pandemia de gripe, 11 de mayo 2009.
364. OMS, Human infection with new influenza A (H1N1) virus: clinical observations from a school-associated outbreak in Kobe, Japan, Weekly Epidemiological Record; 2009, 84, 237–248.
365. Novel Swine-Origin Influenza A (H1N1) Virus Investigation Team, Emergence of a Novel Swine-Origin Influenza A (H1N1) Virus in Humans, New England Journal of Medicine: 2009;360:2605-15.
366. OMS, Human infection with new influenza A (H1N1) virus: clinical observations from Mexico and other affected countries, May 2009, Weekly Epidemiological Record; 2009, 84, 185 -196.
367. Hageman JC, Uyeki TM, Francis JS, et al. Severe community-acquired pneumonia due to Staphylococcus aureus, 2003-04 influenza season. Emerg Infect Dis 2006;12: 894-9.
368. McCullers JA. Insights into the interaction between influenza virus and pneumococcus. Clin Microbiol Rev 2006;19: 571-82.
369. Update: influenza activity United States, September 28, 2008–April 4, 2009, and composition of the 2009–10 influenza vaccine. MMWR Morb Mortal Wkly Rep 2009;58:369-74.
370. http://www.nins.nih.gov/disorders/reyes_syndromew/reyes_syndrome.htm
371. Novel Swine-Origin Influenza A (H1N1) Virus Investigation Team, Emergence of a Novel Swine-Origin Influenza A (H1N1) Virus in Humans, New England Journal of Medicine: 2009;360:2605-15.372 minsa guia influenza 2009
372. Jesús Chacaltana Huarcaya, Alfredo Guerrero Benavides, Carlos Salcedo Espinoza, Patricia Gutierrez Montoya et al. Guía rápida para la atención hospitalaria de influenza (Incluye Influenza A H1N1y Estacional). Aprobado por: Comité Hospitalario para la Atención, Prevención de la Influenza pandémica. Hospital Nacional Daniel A. Carrión. Versión 26 de Junio 2009
373. Servicio Nacional de Sanidad Animal de Argentina. Se detecto el virus A (H1N1) humano en un establecimiento de producción porcina http://www.senasa.gov.ar/contenido.php?to=n&in=1411&ino=0&io=9916. Accesado 31/07/09
CORRESPONDENCIA
Fernando Osores Plenge
fosores@gmail.com