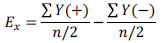Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista de la Sociedad Química del Perú
Print version ISSN 1810-634X
Rev. Soc. Quím. Perú vol.83 no.4 Lima Oct./Dec. 2017
TRABAJOS ORIGINALES
Implementación de una metodología analítica para la cuantificación de proteínas en la microalga Arthrospira platensis
Implementation of analytical methodology for quantification of proteins in Arthrospira platensis microalgae
Leenin Flores Ramos*a, Anthony Ruiz Sotoa
a Laboratorio de Análisis Instrumental - Laboratorio de Biotecnología Acuática, Instituto del Mar del Perú, Esquina Gamarra y General Valle s/n Chucuito Callao , Callao, Perú, lflores@imarpe.gob.pe
RESUMEN
La metodología analítica implementada para la cuantificación de proteínas en la microalga Arthorspira platensis por espectrofotometría UV-Vis fue el método de Hartree-Lowry. Para lograr este objetivo, se optimizaron los factores de extracción de proteínas por el método estadístico de diseño de superficie de respuesta de Box Behnken y se obtuvieron los siguientes resultados de los parámetros implementados: la linealidad, donde se reportó un coeficiente de determinación (R2) mayor a 0,995, en el estudio de repetibilidad un coeficiente de variación menor al 2 %, un porcentaje de recuperación entre 94 % y 101 %, los límites de detección y cuantificación fueron 2,36 μg/mL y 7,86 μg/Ml, respectivamente y el estudio de la robustez demostró que el método analítico tuvo mayor sensibilidad al cambio del peso de la muestra y al cambio del volumen de reactivo C respecto al resto de factores evaluados.
Palabras clave: Espectrofotometría, microalga, Box Behnken, Youden-Steiner.
ABSTRACT
The Hartree-Lowry method was developed and implemented for quantification of protein in Arthorspira platensis microalgae by spectrophotometry UV-Vis. In order to achieve this goal, the protein extraction factors were optimized by Box Behnken design of response surface and the following results of the implementation parameters were obtained: the linearity showed a coefficient of determination (R2) greater than 0.995, a coefficient of variation less than 2% for repeatability, a percentage recovery within 94% to 101%, the limit of detection and limit of quantification were 2.36 μg/mL and 7.86 μg/mL respectively, and the robustness showed that some parameters such as sample weight and volume of reagent C presented high influence in the evaluated factors.
Key words: Spectrophotometry, microalgae, Box Behnken, Youden-Steiner.
INTRODUCCIÓN
Actualmente, las microalgas han sido ampliamente estudiadas por sus numerosos contenidos de compuestos bioactivos, que son aprovechados para su uso comercial1, por ejemplo las microalgas: Dunaliella salina para la producción de β-caroteno, Haematococcus pluvialis para la producción de Astaxantina, Schizochytrium sp. para la producción de ácido docosahexaenoico (DHA), etc.
En particular la microalga Arthrospira platensis es producida por la industria como suplemento alimenticio en diferentes países como Estados Unidos, China, Cuba, India, Tailandia, México, Chile y Sud África, debido a su alto contenido de proteínas 50-70 % y aminoácidos esenciales2. La cuantificación de proteínas es esencial no solo para garantizar la calidad e inocuidad de los alimentos, sino también para facilitar su comercio3.
Los laboratorios analíticos han desarrollado diversas técnicas para la cuantificación de proteínas, por ejemplo el método normalizado Kjeldahl que se aplica en diferentes matrices como cerveza, leche, pan, etc.4 y métodos espectrofotométricos como Lowry, Biuret, Bradford y BCA. Por otro lado, el método de Hartree-Lowry (modificación subsecuente del método de Lowry) es un método espectrofotométrico utilizado para la cuantificación de proteínas debido que presenta ventajas tales como su sensibilidad, límite de detección del orden de 15 μg de proteína, precisión y fácil implementación5.
De acuerdo a normas vigentes, las metodologías no normalizadas deben ser validadas (Resolución Nº 0008-2003/INDECOPI-CRT), por ello, la Asociación de las Comunidades Analíticas (AOAC Internacional), Eurachem, el Codex Alimentarius, Farmacopea de los Estados Unidos de América (USP), ICH, ISO y otros organismos internacionales, recomiendan la implementación del método analítico evaluando los parámetros de desempeño como: veracidad, precisión, especificidad, linealidad, límite de detección, límite de cuantificación, sensibilidad y robustez6.
El objetivo del presente trabajo es implementar el método analítico de Hartree-Lowry para la cuantificación de proteínas en la microalga Arthrospira platensis por espectrofotometría UV-Vis.
PARTE EXPERIMENTAL
Muestra
La microalga Arthrospira platensis liofilizada fue proveída por la Sala de Procesos – Laboratorio de Biotecnología Acuática, IMARPE.
Reactivos
Albúmina de suero bovino BSA (Sigma-Aldrich®), hidróxido de sodio P.A. (Merck), tartrato sodio y potasio tetrahidratado P.A. (Merck), carbonato de sodio anhidro P.A. (Merck), sulfato de cobre pentahidratado P.A. (Merck), reactivo del fenol según Folin-Ciocalteau (Merck).
Equipos
Espectrofotómetro UV-Visible Varian Cary 50, Agitador Vórtex ZX Classic VELP Scientífica, balanza analítica Sartorius MSU225S-000-DU, centrífuga refrigerada Eppendorf Centrifuge 5702R, baño de ultrasonido Branson 2510, campana extractora de gases LABCONCO, baño maría MRC BH-200 y baño maría Eyela SB 2000.
Solución patrón de proteínas
Se pesó 30,0 mg de albúmina de suero bovino, se transfirió a una fiola de 50 mL y se añadió 25 mL de agua ultrapura, se sonicó por 5 min y finalmente se enrazo con agua ultrapura para obtener una concentración de 600 μg/mL.
Extracción de proteínas
Se pesaron 5,0 mg de microalga liofilizada y se añadió 5 mL de hidróxido de sodio, luego se sonicó en baño de ultrasonido por 15 min y se calentó en baño maría para realizar la hidrólisis. Se centrifugó la muestra a 4 ºC por 10 min a 3500 rpm y se extrajo 200 μL del sobrenadante para el análisis de proteínas.
Método analítico
Las proteínas extraídas mediante hidrólisis alcalina y las soluciones estándares fueron cuantificadas por el método de Hartree-Lowry, el cual consistió en la adición de 0,9 mL de reactivo A (hidróxido de sodio 0,5 N, carbonato de sodio anhidro 10 % y tartrato de sodio y potasio tetrahidratado 0,2 %), seguido de una incubación a 50 ºC durante 10 minutos, luego la adición de 0,1 mL de reactivo B (hidróxido de sodio 0,1 N, sulfato de cobre pentahidratado 1 % y tartrato de sodio y potasio tetrahidratado 2 %) y finalmente la adición de 3 mL de reactivo C (reactivo del fenol según Folin-Ciocalteau: Agua 2:15 v/v). Se midió las absorbancias en el espectrofotómetro a una longitud de onda de 650 nm.
Optimización de los parámetros de extracción de proteínas
El método estadístico utilizado para la optimización fue el diseño de superficies de Box Behnken7 y los resultados fueron procesados con el software Minitab 16. Se optimizó la variable respuesta Absorbancia con los siguientes factores, sugeridos por Arredondo y Voltolina8, Concentración de hidróxido de sodio (CNaOH), tiempo de extracción (t) y temperatura (T). Los niveles de los factores se muestran en la Tabla 1. Es necesario mencionar que los valores optimizados fueron los usados en todas las pruebas de implementación.
Parámetros de la implementación
Linealidad
Se construyó la curva de calibración por duplicado en el rango de concentración de 30- 300 μg/mL usando 10 niveles (30, 60, 90, 120, 150, 180, 210, 240, 270 y 300 μg/mL) y empleando la solución patrón albúmina suero de bovino (BSA).
Límite de detección (LOD) y límite de cuantificación (LOQ)
Estos parámetros se obtuvieron de los valores obtenidos de la pendiente (m) y el error estándar del intercepto (sb) de la curva de calibración de acuerdo a las siguientes fórmulas: LOD = 3* sb/ m y LOQ = 10* sb/ m.
Porcentaje de recuperación
Se evaluó el porcentaje de recuperación usando tres niveles de concentración de fortificación (Ca) correspondientes al 25 %, 50 % y 75 % de la concentración estimada del analito en la muestra (Cu, 80 μg/mL) de acuerdo a la siguiente formula: %R = ((Cf-Cu) /Ca) x 100 % donde Cf es la concentración del analito en la muestra fortificada.
Repetibilidad
Se evaluó la repetibilidad analizando nueve muestras independientes bajo las siguientes condiciones: un mismo analista, mismo laboratorio, intervalos de tiempos cortos y los mismos equipos.
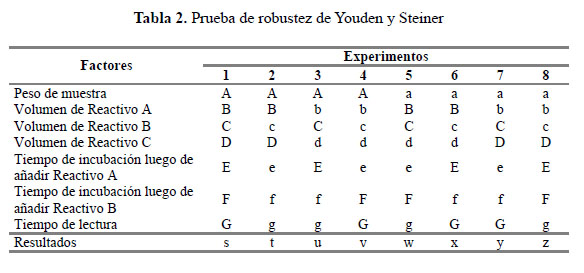
Robustez
Se aplicó la metodología de Youden-Steiner9 y se seleccionaron los siguientes factores, de acuerdo al criterio del laboratorio, Peso de muestra: A = 5 mg, a = 7 mg; Volumen de Reactivo A: B = 900 μL, b = 1000 μL; Volumen de Reactivo B: C = 100 μL, c = 150 μL; Volumen de Reactivo C: D = 3mL, d = 4mL; Tiempo de incubación luego de añadir Reactivo A: E = 10 min, e = 5 min; Tiempo de incubación luego de añadir Reactivo B: F = 0 min, f = 10 min y el Tiempo de lectura: G = instante, g = después de 30 min.
Los efectos fueron calculados con la siguiente formula:
Donde Ex son los efectos del factor x, ΣY (+) es la suma de los resultados factor x en el extremo superior del intervalo definido, ΣY (-) es la suma de los resultados del factor x en el extremo inferior del intervalo definido y n es el número total de experimentos.
RESULTADOS Y DISCUSIÓN
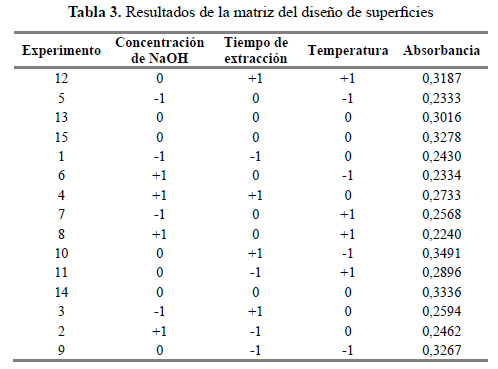
Optimización de los parámetros de extracción de proteínas:
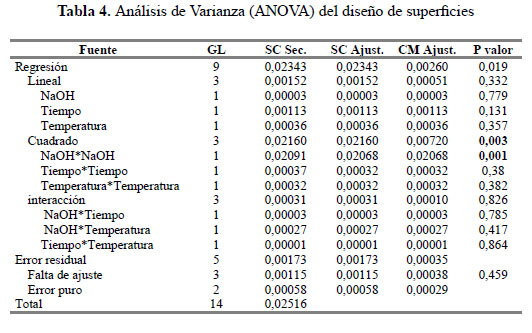
Los resultados del diseño de superficies se muestran en la Tabla 3.
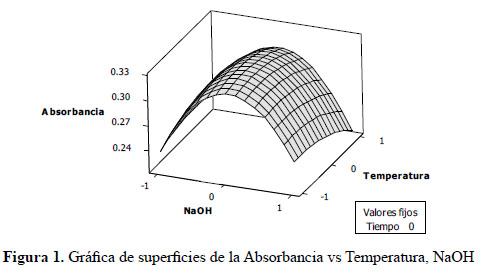
Al graficar la Absorbancia vs la Concentración de NaOH y la Temperatura se puede apreciar una tendencia cuadrática en la superficie debido a la curvatura formada en los niveles del factor concentración NaOH.
El análisis de varianza del diseño de superficies (Tabla 4) confirma que el modelo cuadrático es significativo (p-valor = 0,003) para un nivel de significancia (α) igual a 0,05. Los efectos de las interacciones y términos lineales no fueron significativas (p-valor > α = 0,05). De otro lado, los efectos de los términos cuadráticos fueron significativos solo en el caso de la concentración de NaOH (p-valor = 0,001 < α = 0,05). La ecuación de regresión resultante fue la siguiente:
Absorbancia = 0,321 – 0,002CNaOH + 0,012t – 0,007T + 0,003CNaOHt –
0,008CNaOHT +0,002tT– 0,075CNaOH2 + 0,009t2 – 0,009T2
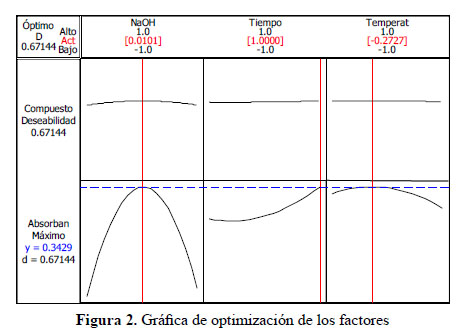
Al optimizar la ecuación con el software se obtuvieron las condiciones óptimas de extracción de proteínas (Figura 2): concentración de NaOH = 0,5 N [0,0101], tiempo = 60 min [1,0000] y temperatura = 75 ºC [-0,2727].
Linealidad
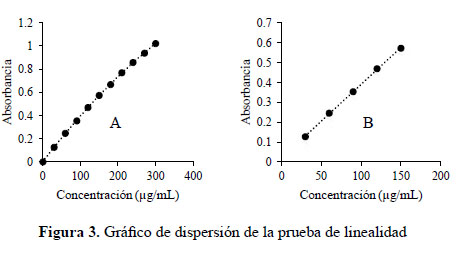
Al graficar los resultados de la prueba de linealidad (Figura 3) se observa que la curva de calibración no presenta linealidad en todo su rango experimental (Figura 3A), debido a ello se seleccionó el intervalo de trabajo de 30-150 μg/mL (5 niveles) donde se observa linealidad (Figura 3B).
Para validar si el modelo es de regresión lineal en el intervalo de trabajo de 30-150 μg/mL se verificaron cuatro supuestos estadísticos:
1.- Supuesto de linealidad
El coeficiente de determinación (R2) 0,9995, superó el valor de referencia R2 ≥ 0,995, usualmente aceptados en los laboratorios como criterio de aceptación10, 11, asegurando el supuesto de linealidad.
2.- Supuesto de independencia de los residuos
El estadístico de Durbin-Watson presentó un valor de 2,33 cercano al valor referencial 2 indicando independencia o incorreción de los residuos.
3.- Supuesto de residuos constantes (Homocedasticidad)
La prueba estadística de homocedasticidad contrasta la hipótesis nula de que variable respuesta tiene varianzas iguales en cada nivel del factor. La prueba de Bartlett presentó un p-valor = 0,951 > α = 0,05, indicando que las varianzas de los residuos no presentan diferencias significativas, asegurando el supuesto de homocedasticidad.
4.- Supuesto de normalidad de residuos
La prueba estadística de normalidad contrasta la hipótesis nula de que la variable tiene una distribución normal. El estadístico Kolmogorov - Smirnov presentó un p-valor = 0,090 > α = 0,05, por lo tanto los residuos presentan una distribución normal.
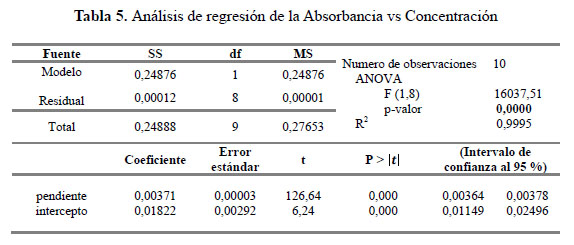
La Tabla 5 muestra los estadísticos de la regresión del modelo lineal.
El análisis de varianza (ANOVA) de regresión muestra que los coeficientes de regresión son significativamente diferentes de cero (p-valor < α =0,05).
El intervalo de confianza al 95 % de la pendiente fue [0,00364 - 0,00378]. Finalmente el modelo de regresión fue:
Absorbancia = 0.01822 + 0.00371 *[Concentración]
Límite de detección (LOD) y límite de cuantificación (LOQ)
En el rango de trabajo de la curva de calibración de 30 a 150 μg/mL, el sb= 0,00292 y el m= 0,00371, por lo tanto el LOD= 2,36 μg/mL y el LOQ= 7,86 μg/mL. Estos valores se encuentran en el orden con lo reportado por Hartree5.
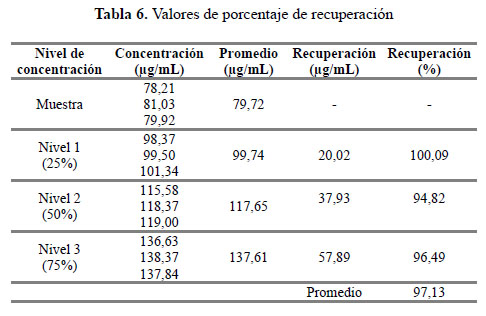
Porcentaje de recuperación
Los resultados (Tabla 6) muestran que el promedio de los porcentajes de recuperación para las concentraciones estudiadas es 97,13 % y se encuentra dentro del rango de aceptación (90 – 107 %) de acuerdo al Manual del Codex Alimentarius12, por lo que se puede aseverar que el efecto de la matriz no es significativo.
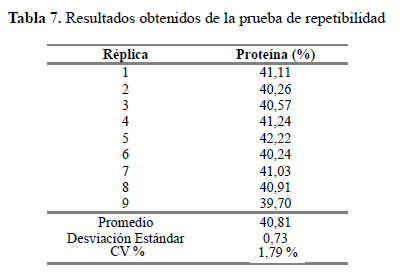
Repetibilidad
La Tabla 7 muestra la precisión bajo condiciones de repetibilidad, el resultado fue un coeficiente de variación (CV % = 1,79 %) y se encuentra dentro del límite de referencia establecido9 (CV % < 5,3 %).
Robustez
La Tabla 8 muestra los resultados de los tratamientos de acuerdo al diseño factorial de Youden- Steiner.
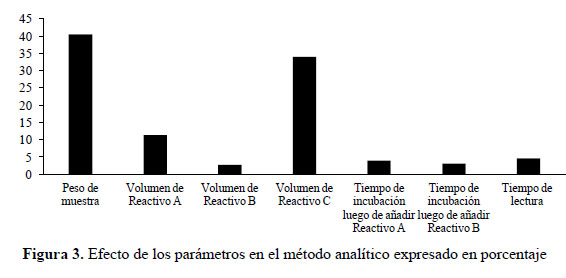
La Figura 4 muestra la proporción de los efectos, donde los factores más sensibles del método analítico fueron el peso de la muestra y el volumen de reactivo C. Posiblemente, al incrementar el peso de muestra no se logre una completa extracción de proteínas y puede disminuir el resultado final. El exceso del volumen de reactivo C, Folin-Ciocalteau, puede disminuir el pH y generar que el óptimo de absorbancia disminuya y alterar el resultado final.
Los factores menos sensibles (más robustos) fueron el volumen de Reactivo A, volumen de eactivo B, tiempos de incubación y el tiempo de lectura.
CONCLUSIONES
Se logró optimizar las condiciones de extracción e implementar la metodología analítica para la cuantificación del proteínas en la microalga Arthrospira platensis por espectrofotometría UV-Vis.
Las condiciones óptimas de extracción de proteínas en la microalga Arthrospira platensis fueron la concentración de NaOH = 0,5 N, tiempo = 60 min y temperatura = 75 ºC.
Los parámetros obtenidos de la implementación fueron: linealidad presentó un coeficiente de determinación de 0,9995; el límite de detección fue 2,36 μg/mL y el límite de cuantificación fue 7,86 μg/mL; el porcentaje de recuperación fue 97,13 %; la precisión en condiciones de repetibilidad del método analítico presentó un CV % de 1,79 % y la robustez demostró que el método analítico es sensible al cambio en el peso de muestra y volumen de reactivo C.
AGRADECIMIENTO
Los autores agradecen al laboratorio de Sala de Procesos por producir la muestra para nuestras pruebas y al Instituto del Mar del Perú por el apoyo brindado para la ejecución de este trabajo.
BIBLIOGRAFÍA
1. Priyadarshani I, Rath B. Commercial and industrial applications of microalgae – A review. J Algal Biomass Utln. 2012; 3(4): 89-100. [ Links ]
2. Rojas E, Ávila M, Parada G. Aplicación de estrategias nutricionales y su efecto en el crecimiento en el cultivo continuo de Spirulina (Arthrospira platensis). Lat Am J Aquat Res. 2012; 40(3): 763-771. [ Links ]
3. Moore JC, DeVries JW, Lipp M, Griffiths JC, Abernethy DR. Total Protein Methods and Their Potential Utility to Reduce the Risk of Food Protein Adulteration. Compr Rev Food Sci Food Saf. 2010; 9: 330-357. [ Links ]
4. AOAC International. Official Methods of Analysis of AOAC International. Rockville Maryland USA: AOAC International; 2016. ISBN: 0-935584-87-0. [ Links ]
5. Hartree EF. Determination of protein: a modification of the Lowry method that gives a linear photometric response. Anal Biochem. 1972; 48(2):422-7. [ Links ]
6. INDECOPI. Aprueban Guía para la validación de Métodos de Ensayo y las Directrices para la Implementación y Evaluación de métodos de Ensayo Sensoriales. [Internet]. El Peruano, 01 de Febrero de 2003, págs. 238363-238366. [consultado 15 jun 2017]. Disponible en: http://www.inacal.gob.pe/inacal/images/docs/acreditacion/requisitospara- solicitar-acreditacion/laborarios-ensayo-calibracion/guiaValidacion.pdf [ Links ]
7. Box G, Draper NR. Empirical model-building and response surfaces. Hoboken, New Jersey: Wiley; 1987. ISBN-10: 047471810339. [ Links ]
8. Arredondo B, Voltolina D. Métodos y herramientas analíticas en la evaluación de la biomasa microalgal. La Paz, Baja California Sur: Centro de Investigaciones Biológicas del Noroeste; 2007. ISBN: 968-5715-51-3. [ Links ]
9. AEFI. Validación de Métodos Analíticos. Barcelona: Asociación Española de Farmacéuticos en la Industria; 2001. [ Links ]
10. EPA. Method 6020B: Inductively Coupled Plasma - Mass Spectrometry. [Internet] SW- 846 Update V; 2014. ¬[Consultado el 12 ago 2017]. Disponible en: https://www.epa. gov/sites/production/files/2015-12/documents/6020b.pdf [ Links ]
11. Srinivas CS, Puranik SB. HPLC Method Development and Validation for Dissolution of Vancomycin Hydrochloride from the Capsules. IJPPR. 2015; 4(2): 301-314. [ Links ]
12. FAO. Codex Alimentarius Commision Procedure Manual. Twenty-first edition. Roma: Food and Agriculture Organization of the United Nations; 2010. [ Links ]
Recibido el 23-01-17
Aprobado el 11-10-17