INTRODUCCIÓN
Los venenos de las serpientes son una mezcla de distintas proteínas, las cuales le han permitido sobrevivir a lo largo del tiempo, ya que, mediante las mismas, han desarrollado un mecanismo de defensa y captura hacia su presa. En este veneno destacan las fosfolipasas A2 (E.C. 3.1.1.4), la cual hidroliza a los fosfolípidos en la posición n-2 del glicerol, liberando lisofosfolípidos y ácidos grasos3,8. Además, existen otras proteínas, tales como; metaloproteasas, serino proteasas, L-aminoácido oxidasas, hialuronidasas y proteína similar a trombina7, las cuales permiten que el veneno se difunda en el tejido, generando diversos efectos biológicos. La superfamilia de fosfolipasas A2 (PLA2) presenta una clasificación de acuerdo a su origen, secuencias aminoacídicas, estructura y función, pudiendo ser; secretoras, citosólicas, lisosomales, dependientes o independientes de calcio3.
En el Perú, la serpiente Bothrops atrox es la más peligrosa y causante de la mayoría de envenenamientos. No obstante, hay mucho interés en los componentes no sólo para generar un antiveneno sino porque presenta ciertas aplicaciones clínicas. Por ello, se realizó un estudio proteómico de tres serpientes peruanas del género Bothrops donde concluyen que la PLA2 es la segunda en abundancia en el veneno7. Además, se han estudiado a las PLA2 en las serpientes, L. muta6 y B. atrox5. Además, se caracterizó por primera vez a la miotoxina del veneno en B. brazili17. No obstante, aún no se ha investigado a las isoformas de PLA2 ácidas en B. atrox ni su efecto en la miotoxicidad. Por ello, tal situación conduce a explorar en mayor profundidad este tipo de efecto biológico.
Las PLA2 pertenece al grupo II, a su vez tienen dos subgrupos: Asp49(D49) y Lys49(K49). Dentro de cada subgrupo, hay a su vez isoformas pudiendo ser; Asp49(D49) ácidas1,16,19,20, Asp(D49) básicas10,12y Lys49 (K49) básicas10,12, las cuales tienen diversos efectos biológicos. La isoforma básica ha sido la más investigada, siendo altamente miotoxica5,10,12, en tanto que a la isoforma ácida también se le atribuye diversos efectos biológicos; miotóxica1,2,16,18, hemolítica indirecta16, inhibidora de agregación plaquetaria1,16,18 e hipotensiva14,20. No obstante, de todos ellos, el efecto miotóxico es el más grave ya que puede dañar irreversiblemente el tejido muscular y dar lugar a amputaciones. Además, se ha reportado que la sinergia entre las miotoxinas D49 y K49, potencian el efecto miotóxico12.
Por otro lado, la predicción estructural teórica terciaria de la isoforma de PLA2 ácida puede permitir entender la relación estructura-función durante el envenenamiento. Dentro de este panorama, la presente investigación estuvo enfocada en aislar a la primera isoforma de PLA2 ácida (BaPer-PLA2a), conocer sus propiedades bioquímicas, moleculares, estructural y fundamentalmente su relación en el proceso miotóxico, el cual presenta gran importancia clínica, teniendo en cuenta que esta acción biológica es la menos sensible a la neutralización con el antiveneno comercial.
PARTE EXPERIMENTAL
Muestras
El veneno se obtuvo de la serpiente peruana Bothrops atrox “jergón” de la zona de Alto Marañón, departamento de Amazonas, mantenida en cautiverio en el serpentario “Oswaldo Meneses” - UNMSM. El veneno extraído fue liofilizado y conservado a 0°C, hasta su empleo.
Purificación de BaPer-PLA2a
Se resuspendió 120 mg de veneno liofilizado en acetato de amonio 50 mM a pH 6,4, se centrifugó a 4000 rpm por 20 min para eliminar restos insolubles. El sobrenadante fue aplicado a una columna cromatográfica de intercambio aniónico DEAE-Sephadex-A50 (16,5 cm x1,8 cm) equilibrada con buffer de elución acetato de amonio 50 mM a pH 6,4 a un flujo de ~12 mL/h. Para eluir las proteínas ligadas a la resina se cambió el buffer isocrático a uno que contenía NaCl 0,3 M y 0,6 M. Aquellas fracciones que mostraron actividad enzimática, fueron juntadas y concentradas para aplicarlas a una columna cromatográfica de exclusión molecular Sephadex-G75 (58 cm x 1,5 cm), equilibrada con el buffer acetato de amonio 50 mM a pH 6,4 a un flujo de ~14 mL/h, colectando fracciones de 2 mL en un equipo colector (Pharmacia LKB SuperFrac). Finalmente, se empleó un sistema cromatográfico automatizado de presión media-NGC (Bio-Rad), utilizando una columna de intercambio anionico ENrichQ (5x50 mm), equilibrada con el buffer de elución de acetato de amonio 50 mM a pH 6,4 y se generó una gradiente de NaCl 0,1-1,0 M. Esta última corrida fue realizada a un flujo de 0,8 mL/min, colectando fracciones de 0,5 mL.
Cuantificación de proteínas
Se realizó mediante dos formas; por absorbancia de luz UV a 280 nm con el espectrofotómetro Spectronic Genesys5 (Spectronic Ins) y por el método de Lowry11 empleando albúmina bovina como proteína estándar.
Actividad enzimática
Se determinó la actividad enzimática de la fosfolipasa A2 ácida empleando el método descrito por Vidal y Stoppani22. Se utilizó el sustrato en una relación (4:5) de yema de huevo y buffer Tris-HCl 10 mM (pH 7.4 y CaCl2 10 mM), respectivamente. Se empleó 1,5 mL de sustrato con 100 μL de veneno crudo o 50μL proteína purificada. La mezcla fue pre-incubada en un baño maría a 37°C por 15 min y se midió el retardo de coagulación de la yema de huevo a 100°C. La actividad específica se expresó como el tiempo de retardo en la coagulación por minuto y miligramo de proteína (U/mg).
Electroforesis en gel de poliacrilamida con dodecil sulfato de sodio (PAGE-SDS)
Se procedió de acuerdo a la metodología descrita por Laemmli9. La muestra fue incubada a 100°C y se preparó un gel acrilamida al 15 %. Se corrió en condiciones reductoras (0,5% β-mercaptoetanol) y no reductoras a 100V en un sistema electroforesis Mini-PROTEAN Tetra Cell, 4-Gel System (Bio-Rad). La tinción del gel fue con azul brillante de Coomassie R-250 por 15 min y la revelación de las bandas se obtuvo por decoloración con una mezcla de metanol, etanol, ácido acético glacial y agua en una relación de 2:1:5:6,5, respectivamente. Se empleó como patrón de bandas proteicas Page Ruler Prestained Protein Ladder (Thermo Scientific).
Actividad miotóxica
Se empleó ratones de la línea Balb/c (18-22g, n=3 por grupo) del Instituto Nacional de Salud (INS), Lima-Perú. Los cuidados éticos de los animales fueron en base; International Guiding Principles for Biomedical Research Involving Animals (1985), by the Council for International Organizations of Medical Sciences (COIMS). En un primer grupo se inyectó en el músculo gastrocnemius, la enzima purificada BaPer-PLA2a (30 μg/100 μL). En un segundo grupo fue inyectada la PLA2 básica miotóxica (20 μg/100μL). En un tercer grupo, la combinación entre la BaPer-PLA2a y la PLA 2 básica miotóxica (5 μg + 20 μg/100 μL) y como control negativo se empleó 100 μL de solución salina. Posterior a las tres horas de la inoculación, se colectó sangre de la vena facial que fue recibida en tubos de 1,5 mL, conteniendo 40 μL de citrato de sodio al 3,8 %. La sangre fue centrifugada a 2000 rpm por 10 min para la liberación del plasma con creatina quinasa (CK). Se utilizó el kit CK-NAC FS* (DiaSys, GmbH-Germany), donde se empleó 40 μL de plasma con 1 mL del kit. La lectura de absorbancia fue a 340 nm, realizada en un espectrofotómetro Genesys5 (Spectronic Ins.) y la actividad creatina quinasa (CK) fue expresada en (U/L).
Diseño de cebadores
Los cebadores se diseñaron a partir de un alineamiento múltiple de las secuencias de PLA2 ácidas depositadas en el Gen Bank correspondiente a las especies: B. moojeni (HQ327311.1), B. neuwiedi pauloensis (GQ850458.1), B. asper (MF319968.1), B. jararacussu (AY145836.1), B. diporus (JQ661499.1; JQ661500.1) y de B. erythromelas (DQ359953.1). Los cebadores fueron diseñados manualmente tomando en cuenta las regiones no traducidas (UTRs) ubicados en los extremos del gen (F1BAPLA2 -R1BAPLA2) y sintetizados por el fabricante (Invitrogen).
Aislamiento de ARN total
Se empleó muestras de veneno liofilizado de B. atrox y siguiendo el protocolo del fabricante TRIzolTM Reagent (Invitrogen): Se agregó 1ml de TRIzol a 40 mg de muestra y se homogenizó por 5 min. Después, se agregó 0,2 mL de cloroformo, se incubó a temperatura ambiente por 3 min y se centrifugó a 12.000xg por 15 min. Luego, se transfirió la fase acuosa a otro tubo, se agregó 0,5 mL de isopropanol, se incubó por 10 min a temperatura ambiente y se centrifugó a 12.000xg por 10 min. Se descartó el sobrenadante y el pellet fue lavado con un 1ml de etanol al 75 %. De inmediato, se descartó el etanol y se secó el pellet por inversión. Finalmente, el ARN total fue resuspendido en 50 μL de agua ultra pura (Invitrogen) libre de ARNasa y se dejó incubar en un AccuBlock Digital Dry Bath (Labnet) a 58°C por 15 min. La cuantificación se realizó en un equipo Nanodrop Lite Spectrofotometre (Thermo Scientific), para calcular la concentración y calidad del ARN total.
Síntesis de ADNc
En la síntesis se siguió el protocolo del fabricante SuperScriptTM III Reverse Transcriptase (Invitrogen). Se partió de 6,66 ng de ARN total, luego se agregó 400 ng de oligo (dT)18-20, 9,2 μL de agua miliQ, 1 μL de mix dNTPs 10 mM c/u. La mezcla se incubó a 65°C por 5 min y un 1 min en frío, se centrifugó brevemente y se agregó 4 µL de buffer First-Strand (5X), 1 µL de DTT 0.1 M, 1 μL de SuperScriptTM III RT (200 units/ μL). Después, la mezcla se homogenizó y se incubó a 50°C por 45 min. Posteriormente, para la inactivación de la reacción se incubó a 70°C por 15 min. La cuantificación se realizó en un equipo Nanodrop Lite Spectrofotometre (Thermo Scientific), para calcular la concentración y calidad de ADNc.
Amplificación de la región codificante de la PLA2 ácida por PCR
La amplificación del gen de la PLA2 por reacción en cadena de la polimerasa por sus siglas en inglés (PCR) fue realizada en un termociclador Mastercycler (Eppendorf). La reacción tuvo 100 ng de ADNc, agua mili Q, dNTPs 0.2 mM, MgCl2 2 mM, tampón buffer con KCl 1X, 0.4 µM de cada cebador forward (5´-ATGAGGACTCTCTGGATAGTGA-3´), reverse (5´-TTAGCATGGCTCTGCCTTCT-3´) y 0,3 μL de Taq Polimerase Recombinant (5 U/ μL). Las condiciones del amplificado fueron: desnaturalización inicial a 95°C por 3 min, desnaturalización a 95°C por 40 seg, alineamiento a 54°C por 45 seg y extensión 72°C por 1 min, por 33 ciclos y extensión final a 72°C por 6 min. El producto de PCR fue corrido en un gel de agarosa (1,2 %) con buffer TBE 1X en un sistema de electroforesis (Cleaver Scientific Ltd), y el amplicón fue estimado con el Ladder GenRuler 100 pb DNA (Thermo scientific). Luego, el gel fue teñido con GelRed (0,01 %) y observado en un UV Transilluminator/ microDoc (Cleaver Scientific Ltd). Los productos fueron secuenciados por EZ-Seq, realizado en la empresa Macrogen, Inc (Corea del Sur).
Análisis bioinformático
Para la identificación de la secuencia obtenida se usó las bases de datos disponibles en National Center for Biotechnology Information (https://www.ncbi.nlm.nih.gov/), UniProt (https://www.uniprot.org/) y para la identificación de estructuras homólogas se basó Protein Date Bank (www.rcsb.org). Se utilizó el programa (ClustalW) para alineamiento y generar la secuencia consenso de nucleótidos, de la cual se basó para obtener la estructura primaria de aminoácidos con Translate tool (https://web.expasy.org/translate/), Asimismo, se determinó el punto isoeléctrico con el programa ProtParam (https://web.expasy.org/protparam/). Por otro lado, también se emplearon los siguientes programas; Robetta (new.robetta.org) para el modelo comparativo, Modeller (https://salilab.org/modeller/) para la homología estructura y PyMol22 para el modelamiento tridimensional de BaPer-PLA2a.
RESULTADOS Y DISCUSIÓN
Purificación y determinación del peso molecular de BaPer-PLA2a.
La presente enzima purificada denominada BaPer-PLA2a, es la primera isoforma ácida en ser aislada del veneno de la especie B. atrox de Perú. En la primera etapa de purificación por intercambio aniónico, las proteínas fueron separadas por su carga. La enzima de interés eluyó después de adicionar NaCl 0,3 M, estando registrada en la caída del cuarto pico (figura 1A). El pool conteniéndola fue sometido a una segunda etapa de purificación por exclusión molecular, la cual permitió separar a la enzima por diferencia de pesos; la BaPer-PLA2a fue registrada en la caída del tercer pico (figura 1B). Posteriormente, se empleó el sistema cromatográfico automatizado de presión media -(NGC), empleando una gradiente de NaCl 0,1-1,0 M para separar a los contaminantes residuales, la cual permitió obtener a BaPer-PLA2a en el tercer pico (figura 1C), verificando que se encontró la enzima en el estado homogéneo, como lo indica en análisis por PADE-SDS (figura 1D). En la tabla 1 se muestra los procesos de purificacion, en la primera etapa de purificacion las unidades totales de actividad (UTA) se incrementaron en 37 % lo que indica la probable liberación de un inbihidor endógeno de dicha enzima. Finalmente, se logró obtener la enzima, a una purificación de 14,82 veces y un rendimiento de 3,39 %.
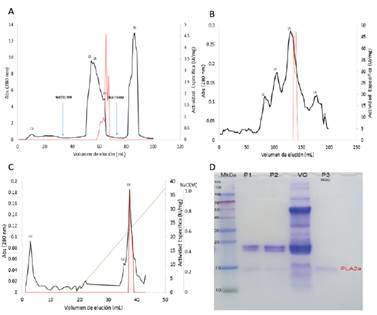
Figura 1 Cromatogramas de purificación. En (A) se muestra la primera etapa, la línea roja muestra la actividad enzimática específica. En (B) la segunda etapa, la actividad específica alcanza un valor de 45 (U/mg). En (C) la tercera etapa de purificación, en línea verde indica la gradiente de NaCl y la enzima en estudio se registra en el tercer pico, mientras que la línea roja indica la actividad específica de BaPer-PLA2a con un valor de 34,1 U/mg. En (D) Se muestra un PAGE-SDS, en condiciones no reductoras. P1 y P2 corresponden a muestras del segundo paso de purificación, en tanto que P3, después del paso por el sistema cromatográfico automatizado. Se llegó a obtener la proteína en su estado homogéneo, con peso de ~14,5 kDa. VC indica veneno total y (M) el marcador de peso molecular.
El uso combinado de sistemas cromatográficos ha sido la estrategia más eficaz para la purificación de las PLA2 en especies tales como: la PLA2 ácida en B. pauloensis1 la PLA2 básica miótoxica en B. atrox5 y PLA2 ácida en B. moojeni19.
El peso molecular de la enzima analizada por PAGE-SDS fue de ~14.5 KDa en condiciones no reductoras (figura 1D). Este resultado es compatible con otras PLA2 ácidas como; en B. pauloensis (15kDa)1, en B. moojeni (15,8kDa)18 y en B. asper (~14,5kDa)16. Por lo tanto, la enzima se trata de proteína de bajo peso molecular. Además, el tratamiento con β-mercaptoetanol sugiere que es monomérica, este resultado es compatible con otros trabajos1,14.
Tabla 1 Purificación de la enzima BaPer-PLA2a del veneno de Bothrops atrox.
| Etapas | Proteína Total | A.E. | U.T.A. | Rendimiento | Purificación | |||
| mg | % | (U/mg) | % | |||||
| Inicial: Veneno crudo | 120 | 100 | 2,3 | 276 | 100 | 1 | ||
| 1° : DEAE-Sephadex A50 | 52,98 | 44,15 | 7,14 | 378,27 | 137 | 3,1 | ||
| 2° : Sephadex G75 | 3,709 | 3,09 | 16,34 | 60,6 | 21,96 | 7,1 | ||
| 3° : ENrichQ | 0,275 | 0,229 | 34,1 | 9,377 | 3,39 | 14,82 | ||
A.E.: Actividad específica (U/mg de proteína), determinado por el método yema de huevo22
U.T.A: Unidades totales de actividad.
Actividad enzimática de BaPer-PLA2a
La medición de la actividad enzimática se realizó usando la prolongación del tiempo de coagulación de emulsión de yema de huevo de Vidal y Stoppani21, en este caso se obtuvo una actividad específica de 34,1 U/mg lo que equivale a una purificación de 14,82 veces comparado con el veneno total (tabla 1). Esto se debió a la hidrólisis de los fosfolípidos a nivel del enlace éster n-2, liberando lisofosfolípidos y ácidos grasos3,8.
Si bien existen métodos más sensibles para medir la actividad de PLA2, el método de yema de huevo permitió hacer el seguimiento rápido de la enzima en cada etapa de purificación. Adicionalmente, se realizó la medición de actividad con el sustrato fosfatidilcolina de la enzima purificada, obteniendo una actividad de 179,6 U/mg.
Mucho se ha discutido acerca de la validez del método enzimático de prolongación del tiempo de coagulación de la yema de huevo ya que se trata de un sustrato heterogéneo. Sin embargo, el método es absolutamente idóneo para fines prácticos teniendo resultados reproducibles. Por otro lado, el método por espectrometría usado con el sustrato fosfatidilcolina tiene una alta sensibilidad, pero el procedimiento es laborioso en términos de aplicación, aunque se requiere pocas cantidades de enzima para el ensayo4,15.
Actividad miotóxica de BaPer-PLA2a.
La enzima por sí sola no mostró actividad miotóxica al obtener valores iguales o menores al blanco durante la liberación de creatina qQuinasa (CK). Sin embargo, cuando es BaPer-PLA2a es combinada con la isoforma de PLA2 básica miotóxica; el valor inicial de la isoforma de PLA2 básica de 740 U/L±0,07 , aumenta a 915 U/L ± 0,065, lo que equivale a un incremento del 21,58 % de la actividad miotóxica (figura 2). La carencia de actividad miotóxica de BaPer-PLA2a se ha reportado en otras isoformas ácidas del grupo II19, no obstante, también se ha reportado que la isoforma de PLA2 ácida es capaz de producir una liberación alta o moderada de creatina quinasa (CK)1,16,18.
Un hecho relevante en esta investigación fue el hallazgo del incremento de la actividad miotóxica cuando se combinan las isoformas de PLA2 ácida y básica, lo que sugiere que durante el envenenamiento podría ocurrir un incremento del daño al tejido muscular por acción sinérgica de ambas isoformas. Asimismo, esto abre un camino para intentar no sólo bloquear a la PLA2 básica miotóxica, sino también a la enzima BaPer-PLA2a. Este resultado es compatible con lo reportado en la combinación de D49 y K4912.
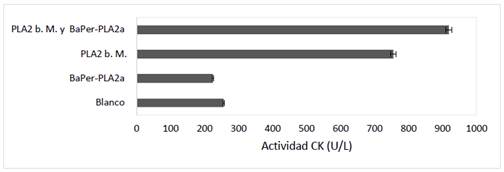
Figura 2 Actividad creatina quinasa (CK). La PLA2 básica miotóxica (20 μg/100 μL), la BaPer-PLA2a (30 μg/100 μL), la combinación de PLA2 básica miotóxica con BaPer-PLA2a (20 μg + 5 μg /100 μL) y el blanco 100 μL de solución salina.
Aislamiento de ARN total, síntesis de ADNc y amplificación por PCR
De 40 mg de veneno liofilizado, se aisló 77 ng/μL de ARN total, por otro lado, a partir de 65 mg tejido glandular, se obtuvo 1282,4 ng/μL de ARN total. Las glándulas del veneno representan una fuente primaria para obtención de altas concentraciones de ARN total1,16. No obstante, esto implica el sacrificio del espécimen que para efectos de la presente investigación no es óptimo y atenta contra los principios de conservación de la especie.
Para la síntesis de ADNc se obtuvo 541 ng/μL, a partir de veneno liofilizado y 762,5 ng/μL de tejido glandular. Por otra parte, en la amplificación del gen de BaPer-PLA2a se obtuvo un producto de ~480 pb (figura 3), tamaño que es comparable a otros amplificados de 448 pb en L. muta6, 417 pb de B. pauloensis1 y de 414 pb en B. moojeni19.

Figura 3 Electroforesis en agarosa 1 %. En C2 se muestra en el cuadro en blando el amplificado con un peso de ~ 480 pb. Siendo M el marcador en pares de bases (100-1000 pb).
Análisis bioinformático de la BaPer-PLA2a.
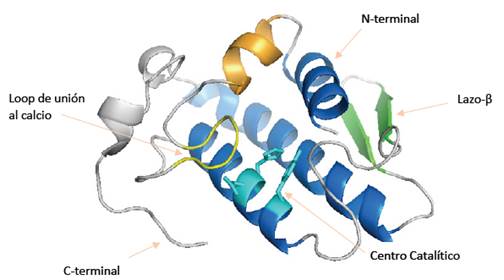
La estructura primaria de la enzima presenta 124 aminoácidos deducidos a partir de la secuencia de nucleótidos obtenida posterior a la amplificación. Los análisis por Blastp (NCBI) demostraron homología de la proteína deducidas con PLA2 ácidas depositadas en esta base de datos. Posteriormente, el alineamiento múltiple con las PLA2 ácidas de 11 especies del genero Bothrops mostró que la BaPer-PLA2a presenta una identidad entre 70-84 % con sus semejantes (figura 4). Asimismo, existe 83 % de identidad con la PLA2 ácida de B. asper de Colombia, esto puede ser debido a la cercanía taxonómica entre ambas especies amazónicas. La BaPer-PLA2a presentan 14 aminoácidos cisteínas altamente conservados, las cuales formarían los siete enlaces disulfuro, mientras que los cuatro aminoácidos en las posiciones (Tyr28, Gly30, Gly32 y Asp49) formarían el loop de unión al calcio y un centro catalítico constituido por los residuos (His48, Asp49, Tyr52 y Asp99) (figura 4). Además, se dedujo su punto isoeléctrico (pI) teórico de 4,41.
Esto permitió concluir que se trata de una isoforma de PLA2 ácida.
Estos resultados son similares a los reportados en B. insulares2, B. asper17, B. pauloensis1 y B. moojeni18,19. Además, son regiones altamente conservadas en las fosfolipasas A2 ácidas pertenecientes al grupo II de las serpientes de la familia Viperidae.
EL análisis bioinformático coloca en cercanía de congruencia de identidad a la secuencia proteica deducida con la proteína purificada en el presente trabajo, esto porque los cebadores fueron diseñados partiendo de la región conservadas de los UTR 5`y 3` de las secuencias de las PLA2 ácidas con mayor cercanía taxonómica a la especie estudiada.

Figura 4 El alineamiento múltiple de la estructura primaria de BaPer-PLA2a con otras fosfolipasas A2 ácidas de varias serpientes del género Bothrops. Las accesiones de las proteínas se muestran en la primera columna. Se muestra el número de aminoácidos, porcentaje de identidad y la especie de origen. El aminoácido de cisteína se muestra en color gris. El loop para la unión al calcio se muestra en color amarillo, en tanto que los aminoácidos del centro catalítico se muestran en color rojo-cian.
Modelamiento estructural de la BaPer-PLA2a
El modelamiento estructural se realizó en base a homología con las estructuras cristalizadas de la BthA-I-PLA2 de B. Jararacussu13,14 (PDB: 1ZLB, 1U73 y 1UMV), los cuales tiene una identidad del 70-75 % con la BaPer-PLA2a empleando el programa Modeller y generado en el programa PyMol22. La estructura construida (figura 5) presenta tres α-hélice, un lazo-β, un loop para la unión al calcio y un centro catalítico (His48, Asp49, Tyr52 y Asp99)20. Además, este modelo también fue reproducido por otras investigaciones como la de BmooPLA2 en B. moojeni21.
CONCLUSIONES
Esta investigación permitió purificar la primera isoforma ácida de PLA2 (BaPer-PLA2a) a partir del veneno de la serpiente B. atrox que habita en la selva peruana. Posee actividad enzimática sobre lipoproteínas de yema de huevo y fosfatidilcolina, pero carece de actividad miotóxica. Su peso molecular fue de ~14,5 kDa en condiciones no reductoras y su pI (4,41), calculado in silico. La enzima incrementa la actividad miotóxica en 21,58 % de su homóloga PLA2 básica, la cual es una miotoxina. Asimismo, se obtuvo una secuencia proteica deducida cercana a las características de la proteína purificada la cual pertenece al subgrupo (D49), presenta una estructura primaria de 124 aminoácidos, un centro catalítico formado por los residuos (His48, Asp49, Tyr52 y Asp99) y un loop para la unión al calcio. Finalmente, la enzima mostró una homología estructural teórica del 74,5 % con 1ZLB.
Por otro lado, la investigación demostró que la isoforma ácida potencia el efecto de miotoxicidad elevando los niveles de daño al tejido muscular, lo cual sugiere que es importante considerar a la BaPer-PLA2a durante el desarrollo de algún antiveneno, mientras que el análisis de la estructura primaria permitió, también, identificar regiones variables, las cuales podrían ser atribuidas al aumento de la miotoxicidad cuando se combinan con la isoforma básica. Finalmente, el modelo teórico estructural permitió una comprensión parcial de la enzima y dejando abierta la posibilidad de estudios posteriores.