Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Scientia Agropecuaria
versión impresa ISSN 2077-9917
Scientia Agropecuaria vol.6 no.1 Trujillo 2015
http://dx.doi.org/10.17268/sci.agropecu.2015.01.05
ARTÍCULOS ORIGINALES
Transferencia de algunos marcadores moleculares microsatélites de la familia Fabaceae en tarwi (Lupinus mutabilis Sweet)
Transference of some microsatellite molecular markers from Fabaceae family to Andean Lupin (Lupinus mutabilis Sweet)
Michelle C. Chirinos-Arias1,*; Jorge E. Jiménez2
1 Centro de Diagnóstico Molecular S.A.C, Calle Monterosa 270 oficina 503, Surco, Perú.
2 Laboratorio de Biotecnología, Programa de Cereales y Granos Nativos, Universidad Nacional Agraria La Molina, Lima, Perú.
Resumen
Con el objetivo de analizar la transferencia de 15 iniciadores microsatélites de la familia Fabaceae en Lupinus mutabilis Sweet "tarwi", escogidos por presentar transferibilidad entre especies y géneros, por su alto índice de contenido polimórfico (PIC) y alto grado de heterocigosidad observada y esperada. Se extrajo el ADN de 300 plantas de tarwi, se estandarizaron las condiciones PCR haciendo gradientes de los diferentes componentes del master mix. Para el screening se corrieron los iniciadores en gel de agarosa 3% con algunas muestras. Finalmente se amplificó la población y se corrieron en gel de poliacrilamida 6% por su mayor resolución. Sólo el 6,67% de iniciadores amplificaron pero resultaron ser monomórficos, por lo que no pueden usarse en caracterización molecular. Se propone ocho iniciadores microsatélites para tarwi, los cuales deben ser probados en condiciones de laboratorio.
Palabras clave: Lupinus mutabilis, familia Fabaceae transferencia, marcadores moleculares microsatélites.
Abstract
In order to analyze transferibility of 15 microsatellite primers from Fabaceae family to Lupinus mutabilis Sweet "andean lupine" chosen to present transferability between species and genera, by its high rate of polymorphic content (PIC) and high degree of observed and expected heterozygosity. DNA was extracted of 300 andean lupines plants, PCR conditions were standardized by gradients of master mix components. Primers for screening were run on 3% agarose gel with some samples. Finally population was amplified and run on 6% polyacrylamide gel for its highest resolution. Only 6.67% of primers were amplified, but they were monomorphic, so they cannot be used for molecular characterization. We proposed eight microsatellite primers for andean lupin wich should be probed in laboratory conditions.
Keywords: Lupinus mutabilis, Fabaceae family, transference, microsatellite molecular markers.
1. Introducción
La familia Fabaceae es la tercera más numerosa de las plantas con flores (Sato et al., 2010; Ceroni, 2003). Sin embargo, solo se han secuenciado o están casi secuen-ciados completamente pocos genomas pertenecientes a las siguientes especies modelo: Lotus japonica, Medicago truncaluta y Glycine max (Sato et al., 2010). A pesar de esta carencia de genomas secuenciados, se ha probado la transferibilidad de la información de una especie a otra (en el caso de los microsatélites), siendo ésta incluso entre género y género, un claro ejemplo son las secuencias EST-SSR (Expressed Sequence Tag-Simple Sequence Repeat) altamente transferibles en estudios moleculares con microsatélites (Sato et al., 2010; Bory et al., 2007; Gong et al., 2010; Liu et al., 2010).
Los estudios moleculares basados en estos marcadores se han realizado en L. luteus y L. angustifolia para los cuales se han diseñado iniciadores SSR (Simple Sequence Repeat) en base a las secuencias EST de Lotus japonica (Gonzalez et al., 2010). Lamentablemente no existen estudios moleculares en L. mutabilis Sweet, excepto el realizado por Jiménez (2006) usando iniciadores AFLPs.
Existen dos clases de marcadores genéticos: los morfológicos y los moleculares (Tanksley, 1983; Azofeifa, 2006; Ferreira & Grattapaglia, 1998), siendo estos últimos los más confiables y utilizados en la actualidad (Sparks, 2007; Azofeifa, 2006). Un marcador molecular es una secuencia de ADN fácilmente detectable cuya herencia puede ser monitoreada de manera sencilla. Éstos se basan en el análisis de las diferencias que se dan entre individuos dentro de pequeñas secuencias de ADN (Azofeifa, 2006; Ferreira & Grattapaglia, 1998). Algunas ventajas de los marcadores moleculares, en comparación con los morfológicos, son: la facilidad en la elaboración de mapas genéticos, ya que permiten analizar una gran cantidad de loci polimórficos. Por otro lado, los marcadores moleculares son generalmente neutros y usualmente presentan herencia codominante, por lo que contienen mayor información genética. Además, su análisis se puede realizar en cualquier estadío de desarrollo utilizando cualquier muestra de células o tejido vivo. También es necesario recalcar que dentro de los marcadores moleculares se menciona la existencia de dos tipos: las proteínas (principalmente las isoenzimas) y los marcadores de ADN. Dentro de estos últimos tenemos a los ISSR, RAPD, AFLP, RFLP, SSR, etc. (Azofeifa, 2006).
Siendo necesario que este tipo de marcadores de ADN cumplan dos cualidades para su aplicación en mejoramiento genético: a) deben permitir la inequívoca diferenciación de los progenitores y ser transmitido en forma estable a la progenie (Paterson et al., 1992). b) deben generar abundante información, dentro de un lapso de tiempo prudente y a un nivel de costo razonable.
Los marcadores microsatélites (SSR) tienen muchas ventajas debido a que son altamente polimórficos, robustos y co-dominantes. Además debido a su transferencia entre diferentes especies es usado en estudios de conservación genética (Moreira et al., 2012).
El análisis de la transferencia de inicia-dores microsatélites permitirá caracterizar molecularmente la especie para su posterior mejoramiento genético. Siempre y cuando los iniciadores identificados sean polimórficos. Pues iniciadores monomórficos no se pueden utilizar en análisis de variabilidad genética.
Por tal motivo, este estudio propone probar la transferencia de algunos iniciadores de la familia Fabaceae en L. Mutabilis Sweet "tarwi" y determinar si el porcentaje de transferencia es lo suficiente recomendable para continuar con estos análisis o si es tan bajo que se recomendaría diseñar inicia-dores específicos para el tarwi (el cual tendría que secuenciarse) o en todo caso realizar estudios moleculares con otros marcadores de ADN que permitan realizar la caracterización molecular.
2. Materiales y métodos
Siembra de accesiones
Se sembraron 300 semillas de tarwi correspondientes al banco de germoplasma del Instituto de Innovación Agraria (INIA). Las cuales se mantuvieron bajo condiciones de tinglado, se las regó dos a tres veces por semana. Se recolectaron las dos primeras verdaderas hojas a los 15 días de la siembra. Luego se secaron las muestras en sílica gel. Una vez secas, las hojas se trituraron y se colocaron en microtubos de 1,5 ml para la extracción de ADN. Todos estos pasos fueron realizados en condiciones de asepsia, usando en todo momento guantes y alcohol al 70%.
Extracción y cuantificación del ADN
Se extrajo el ADN de las 300 muestras siguiendo el método micro CTAB (Cetil Trimethyl Ammonium Bromide) indicado por Doyle y Doyle (1987) modificado en el Laboratorio de Biotecnología del Programa de Cereales y Granos Nativos.
Iniciadores microsatélites
Considerando que los estudios con marcadores Microsatélites requieren de información previa del genoma para diseñar los iniciadores SSR, que la especie en estudio no se encuentra entre las leguminosas que están siendo secuenciadas (por tanto no existe información previa de su genoma) y que el diseño de iniciadores demanda tiempo y dinero. Se realizó un screening de iniciadores SSR (Tabla 1) provenientes de la familia Fabaceae (escogidos por presentar transferibilidad entre especies y géneros, por su alto índice de contenido polimórfico y alto grado de heterocigosidad observada y esperada).

Estandarización de la amplificación por PCR
Las muestras de ADN se diluyeron a una concentración uniforme de 10 ng/μl con agua ultrapura. Las temperaturas de alinea-miento de los iniciadores microsatélites de la familia Fabaceae escogidos se tomaron de la literatura. Sin embargo, también se calcularon usando Tm CALCULATOR NEB Software de New England Biolabs (disponible en https://www.neb.com/tools-and-resources/interactive-tools/tm-calculator). Con los datos de estas temperaturas, se empezó a probar las condiciones de PCR. Al no producirse la amplificación con las temperaturas de alineamiento citadas en la literatura ni con la que nos brindó el software. Se procedió a bajar la temperatura dos, cuatro y seis grados centígrados.
Por último se fue subiendo la temperatura dos y cuatro grados hasta llegar a un máximo de 65 °C, con el fin de encontrar la temperatura correcta de hibridación de los iniciadores.
El mix de PCR estuvo compuesto por ADN (20 ng/reacción), iniciadores forward y reverse (1μM), MgCl2 (2mM), dNTP (0,25mM), Taq DNA Polimerasa Fermentas (0,5U), buffer Taq (1x) y completando con agua grado PCR para un volumen final de 10uL. Se incluyeron controles negativos a los cuales se le agregó agua en lugar de ADN y controles positivos a los cuales se le agregó el ADN de la planta de la familia Fabaceae en el que fue previamente reportado.
Se probaron diferentes protocolos de amplificación para cada uno de los 15 iniciadores estudiados usando las tempe-raturas de alineamiento de la literatura y del software (Tabla 2).
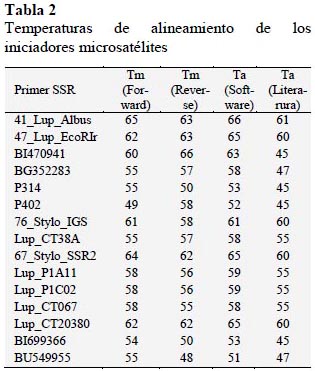
Las condiciones de PCR fueron de 94 ºC por 3 minutos, seguido de 35-45 ciclos de 94 ºC por 1 minuto, temperatura de alineamiento por 1 minuto y 72 ºC por 2 minutos, con una amplificación final de 72 ºC por 10 minutos.
El número de ciclos se obtuvo de la literatura y se amplió unos diez ciclos más para asegurar la amplificación del producto de PCR o para descartar la hibridación del iniciador con el ADN, según sea el caso. Para mejorar la amplificación se usó el método descrito por Gonzalez et al. (2010), que incluyó cinco ciclos de PCR en la cual la temperatura se disminuyó 1 °C (touch-down), hasta llegar a la temperatura de alineamiento, seguido por 35 ciclos de 94 ºC por 1 min, temperatura de alineamiento por 2 min y 72 ºC por 1 min, con una amplificación final de 72 ºC por 10 min. Para evitar la formación de dímeros y productos inespecíficos y con el fin de mejorar el tamizado de iniciadores micro-satélites, se usó Taq polimerasa hot start (activación a 95°C por 10 minutos) y se corrieron las muestras en un termociclador en tiempo real Eco Illumina por ser de mayor resolución.
Por último se utilizaron coadyuvantes para PCR como el DMSO (Dimetilsulfóxido) y el BSA (Albúmina de Suero Bovino), hasta estandarizar las condiciones de PCR. Una vez estandarizadas, se amplificó toda la población usando el termociclador Eppendorf MasterCycler.
Post PCR
Para el tamizado de iniciadores se emplearon geles de agarosa al 3,0% y se corrieron solo 10 muestras. Con los iniciadores escogidos se corrió la población en estudio en gel de acrilamida 6% por su mayor resolución y por el mayor número de muestras con el fin de encontrar polimorfismo.
Diseño de iniciadores microsatélites
Se utilizó la base de datos del NCBI (National Center for Biotechnology Information) para buscar las secuencias conservadas en la familia Fabaceae, para lo cual se utilizó el conjunto de secuencias de estudios poblaciones y filogenéticos (PopSet). Se priorizaron los datos que contenían a L. mutabilis y se hizo un alineamiento de las secuencias usando el software BLAST (Basic Local Alignment Search Tool). Se escogieron los alinea-mientos con puntuaciones mayores a 80, las secuencias que tuvieron mayor iden-tidad y un valor de E (valor esperado) menor a 1e-4 por ser considerado con un error menor al 0,01%. Para la búsqueda de microsatélites se usó el software SSRIT- Simple Sequence Repeats identification Tool de Gramene Ssrtool disponible en (http://archive.gramene.org/db/markers/ssrtool). Se diseñaron iniciadores usando el software Primer 3 Plus (disponible en http://www.bioinformatics.nl/cgi-bin/primer3plus/primer3plus.cgi) siendo el tamaño óptimo del iniciador de 20 bases, su temperatura de fusión de 55 a 63 ºC, el porcentaje de GC óptimo de 50% y se verificó que contenga al microsatélite encontrado. Por último se usó Primer Blast para cerciorarnos que no se formen productos inespecíficos y que la secuencia solo se ancle en la región de interés (http://www.ncbi.nlm.nih.gov/tools/primer-blast/).
3. Resultados y discusión
Se observó que solo los iniciadores 41_Lup_Albus (Mace et al., 2008) y P 402 (Gong et al., 2010) presentaron bandas en el gel de agarosa (3%). El primero con una temperatura de alineamiento (Ta) de 60 °C y el segundo con una Ta de 45 °C, datos experimentales que coinciden con lo reportado por Mace et al. (2008) y Gong et al. (2010) respectivamente. Pero, cuando se hizo la corrida en gel de acrilamida para el iniciador 41_Lup_Albus. Se observó que sólo algunas muestras (bulks) amplificaban en regiones de aproximadamente 200 bp y 300 bp.
Según Parra et al. (2010), quién desarrolló marcadores microsatélites para L. luteus y su transferibilidad a otras especies de Lupinus, el rango de tamaño de los amplicones para este género está entre 162 bp (el menor) y 371 bp (el mayor). Por lo que las bandas observadas podrían corresponder a marcadores microsatélites. Sin embargo, el hecho que algunas muestras no amplifiquen (incluso en las posteriores repeticiones) a pesar de tener buena calidad de ADN, parece indicar que se trataría de productos inespecíficos. Esto se comprobó por PCR en tiempo real, en la cual los productos inespecíficos usual-mente tienen un ciclo umbral o threshold cycle (Ct) temprano. Mientras que, si son dímeros de iniciadores amplifican tardíamente. En ambos casos, las curvas de amplificación no siguen el patrón usual (línea base - curva exponencial - curva de plateau).
En este caso se observó una amplificación temprana (alrededor del quinto ciclo) de dos de las tres muestras, lo que correspondería a productos inespecíficos. A pesar que se usó una ADN Polimerasa Hot Start que evita su formación. Por lo que este iniciador no es recomendable en el estudio de estas muestras.
Por otro lado, Mace et al. (2008) describe un amplicón de 188 bp en L. albus para este iniciador. Es decir de menor contenido de pares de bases (bp) que las bandas halladas y la presencia de tres alelos. A diferencia de Gonzalez et al. (2010) cuyo trabajo describe dos alelos. Además el hecho que su motivo repetido sea AAATC (2) dificulta el anclaje, ya que se recomienda motivos cortos y de mayor repetición en estudios los cuales usen iniciadores microsatélites desarrollados para otras especies (Gong et al., 2010), razón por la cual este iniciador no es adecuado para las muestras de L. mutabilis usadas en este estudio.
A pesar que se ha probado que algunos iniciadores microsatélites pueden amplificar en otras especies para lo cual no fueron desarrollados y que incluso son transferibles exitosamente entre género (Moreira et al., 2012). Este iniciador que amplifica en una especie del mismo género del tarwi, como lo es L. albus, no lo hace con el L. mutabilis. "Quizás la diferencia en el tamaño del genoma de las especies, la fuerza con la que se produce la hibridación y la temperatura de alineamiento (Ta) pueden haber influido en la amplificación" (Mace et al., 2008).
Por otro lado, el iniciador P 402 de motivo TC (10) de Pisum sativum L. amplificó en las muestras de tarwi. Se observaron bandas entre 230 bp y 310 bp. En el caso de la arveja el tamaño del alelo encontrado fue de 352 bp (Gong et al., 2010). Lo que guarda concordancia con estos hallazgos, por lo que los productos observados corresponderían a segmentos micro-satélites. Sin embargo, en la corrida en gel de poliacrilamida de las 300 muestras, se obtuvieron bandas monomórficas, por lo que no se puede realizar un estudio de variabilidad genética o caracterización molecular con los datos obtenidos.
Los demás iniciadores no fueron visibles en gel de agarosa. Aunque el iniciador Lup_CT38A amplificó en muestras chilenas de L. mutabilis en el estudio de Gonzalez et al. (2010), no amplificó con las muestras de este estudio. A pesar de que se siguió el mismo protocolo y luego se varió las condiciones en busca de las óptimas, como se describe en la sección anterior. Este mismo autor coincide con nuestros resultados para los iniciadores Lup_P1A11, Lup_P1C02, Lup_CT067 y Lup_CT20380 que a pesar de amplificar el ADN de L. albus no amplifica las muestras chilenas de L. mutabilis. Según Mace et al. (2008) esto se podría explicar por el bajo porcentaje de amplificación de iniciadores microsatélites observado en las leguminosas que corresponden solo al 31%.
Cabe recalcar que muchos de los microsatélites probados no presentaban el motivo (AG)n que ha demostrado estar ampliamente distribuido en el genoma de Lupinus sp. (Sbabou et al., 2010), lo que explicaría que no se produjera amplificación. Lamentablemente ninguno de los iniciadores microsatélites se utilizó en el análisis de variabilidad genética de tarwi, por ser monomórficos. Se debe aclarar que los controles negativos como es de esperar no amplificaron. Mientras que, los controles positivos amplificaron con el ADN de las plantas con las cuales fueron descritas previamente, a excepción de la variedad de tarwi chileno.
Estudios como el de Sun et al. (2012) desarrollaron 300 microsatélites de los cuales 117 (39%) amplificaron y solo 44 fueron polimórficos (14,67%). Estos iniciadores fueron diseñados especialmente para la especie y tuvieron éxito. Sin embargo, el porcentaje de marcadores polimórficos es bajo. Por lo que se recomendaría trabajar con más iniciadores para probar su transferibilidad en tarwi, ya que en este estudio la cantidad de marcadores microsatélites usada fue baja.
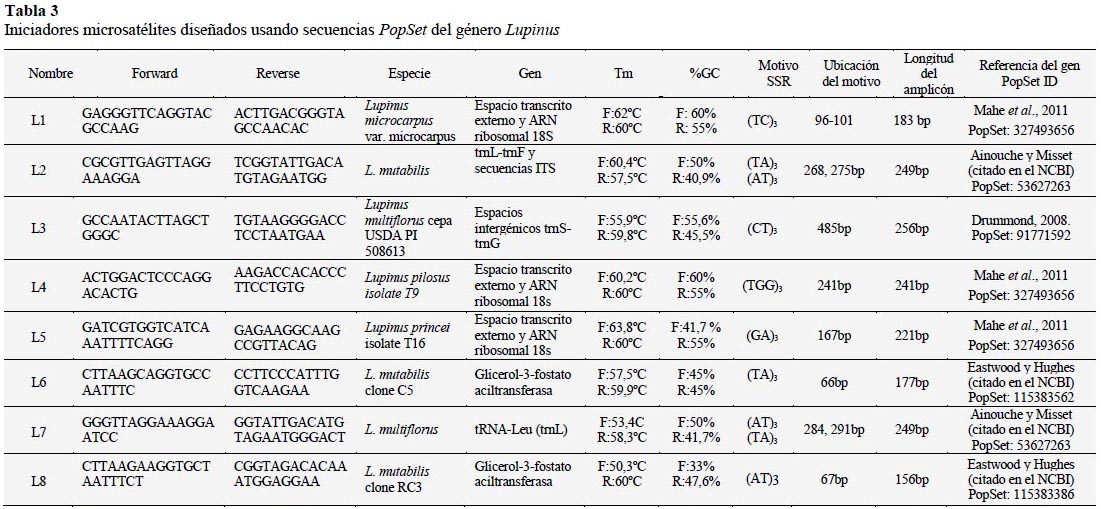
Por otro lado, usando herramientas bioinformáticas se propone ocho inicia-dores que pueden ser usados en estudios posteriores. Para ello se usaron las secuencias disponibles en el NCBI que eran conservadas entre especies y que había sido previamente reportadas en filogenética mediante los datos PopSet. Además se usaron datos de la familia Fabaceae para lo cual nos enfocamos en el género Lupinus, ya que hay mayor probabilidad que en ellas se encuentren iniciadores que anclen en tarwi. Solo se encontraron 16 resultados y se trabajaron con ellos. Las secuencias microsatélites encontradas se corroboraron con Gramene Ssrtool. En la Tabla 3 se muestra los iniciadores escogidos. De ellos, se recomienda que el contenido de GC% sea entre 40-60%. Sin embargo, usando el software Primer 3 Plus, se obtuvo un iniciador L8 con un valor muy bajo de GC, por lo que se debería dar prioridad a los otros iniciadores propuestos. Por otro lado, las temperaturas de fusión de ambos iniciadores deberían ser cercanas y diferenciarse en 5 ºC. Lo que también es un problema para el iniciador L8, ya que la variación entre los iniciadores es mayor. Sin embargo, se recomienda probarlo ya que el motivo es diferente al L6, aunque pertenecen al mismo gen.
4. Conclusiones
La temperatura de alineamiento es el principal factor a encontrar para la estandarización de la PCR.
Para ello es necesario no solo guiarse de la literatura, si no usar programas bioinfor-máticos que faciliten hallar dicha tempe-ratura. De los 15 iniciadores microsatélites solo el iniciador P 402 de Pisum sativum "arveja", fue transferible de esta especie a Lupinus mutabilis "tarwi". Aunque ambas especies pertenecen a la familia Fabaceae, este iniciador mostró ser completamente monomórfico para las muestras en estudio, por lo que no se recomienda en estudios de variabilidad genética o caracterización molecular. Según Mace et al. (2008) el porcentaje de transferibilidad de los iniciadores micro-satélites en la familia Fabaceae es bajo (31%). En esta investigación se observó transferencia del 6,67% de los iniciadores, pero sin evidencia de polimorfismo. Se recomienda probar más microsatélites dentro de la familia Fabaceae lo cual daría mayor oportunidad de encontrar micro-satélites que puedan ser usados en tarwi y con polimorfismo como se ha evidenciado en otros trabajos. Otra alternativa es la secuenciación del genoma del tarwi, para el desarrollo de iniciadores específicos para esta planta. Así como estudios moleculares usando otros marcadores de ADN como ISSR, RAPD, entre otros que ayuden en estudios de caracterización molecular para facilitar el estudio de duplicados en el banco de germoplasma y la determinación de la variabilidad genética para el mejoramiento genético.
Agradecimientos
Al Programa de Cereales y Granos Nativos de la Universidad Nacional Agraria La Molina por el financiamiento de los materiales y reactivos usados en la investigación.
5. Referencias bibliográficas
Ainouche, A.; Misset, MT. Lupinus phylogeny based on the trnL-trnF and ITS sequences. National Center for Biotechnolgy Information. Disponible en: http://www.ncbi.nlm.nih.gov/popset/?term=53627263.
Azofeifa, A. 2006. Uso de marcadores moleculares en plantas: aplicaciones en frutales del trópico. Agronomía Mesoamericana 17: 221-242.
Bory, S.; Da Silva, D.; Risterucci, A.M.; Grisoni, M.; Besse, P.; Duval, M.F. 2008. Development of microsatellite markers in cultivated Vanilla: Polymorphism and transferability to other vanilla species. Scientia Horticulturae 115: 420–425.
Ceroni, A. 2003. Distribución de las leguminosas de la parte alta de la cuenca Gallega Morropón- Piura. Ecología Aplicada 2(1): 9-13.
Doyle, J.J.; Doyle, J.L. 1987. A rapid DNA isolation procedure from small quantities of fresh leaf tissues. Phytochem Bull. 19:11-15.
Drummond, C.S. 2008. Diversification of Lupinus (Leguminosae) in the western New World: derived evolution of perennial life history and colonization of montane habitats. Mol. Phylogenet. Evol. 48: 408-421.
Eastwood, R.J.; Hughes, C.E. Phylogeny of Lupinus. National Center for Biotechnolgy Information. Disponible en: http://www.ncbi.nlm.nih.gov/popset/?term=115383562.
Ferreira, M.; Grattapaglia, D. 1998. Introducción al uso de marcadores moleculares en el análisis genético. Editorial Embrapa Cenargen. Brasil.
Gong, Y.M.; Xu, S.C.; Mao, W.H.; Hu, Q.Z.; Zhang, G.W.; Ding, J.; Li, Y.D. 2010. Developing new SSR markers from ESTs of pea. Journal of Zhejiang University-Science B 11: 702-707.
Gonzalez, L.B.; Straub, S.C.; Doyle, J.J.; Ortega, P.E.; Garrido, H.E.; Butler, I.J. 2010. Development of microsatellite markers in Lupinus luteus (Fabaceae) and cross - species amplification in other lupine species. American Journal of Botany 97(8): 72-74.
Jiménez, J. 2006. Biodiversity of traditional seed propagated crops cultivated in Peruvian highland. Tesis Ph.D. University of Silesia. Polonia.
Liu, Y.L.; Li, Y.H.; Zhou, G.A.; Uzokne, N.; Chang, R.Z.; Chen, S.Y.; Qiu, L.J. 2010. Development of soybean EST-SSR markers and their use to assess genetic diversity in the Subgenus Soja. Agricultural Sciences in China 9: 1423-1429.
Mace, E.S.; Varshney, R.K.; Mahalakshmi, V.; Seetha, K.; Gafoor, A.; Leeladevi, Y.; Crouch, J.H. 2008. In silico development of simple sequence repeat markers within the Aeschynomenoid/Dalbergoid and Genistoid clades of the Leguminosae family and their transferability to Arachis hypogaea, groundnut. Plant Science 174: 51–60.
Mahe,F.; Pascual,H.; Coriton,O.; Huteau,V.; Navarro-Perris,A.; Misset, M.T.; Ainouche, A. 2011. New data and phylogenetic placement of the enigmatic Old World lupin: Lupinus mariae-josephi H. Pascual. Genet. Resour. Crop Evol. 58: 101-114.
Moreira, P.A.; Sousa, S.A.; Oliveira, F.A.; Araújo, N.H.; Fernandes, G.W.; Oliveira, D.A. 2012. Characte-rization of nine transferred SSR markers in the tropical tree species Enterolobium contortisiliquum (Fabaceae). Genet. Mol. Res. 11: 3729-3734.
Paterson, A.H.; Tankley, S.D.; Sorrells, M.E. 1992. DNA markers and plant improvement. Adv. Agron. 36: 39-90.
Sato, S.; Isobe, S.; Tabata, S. 2010. Structural analyses of the genomes in legumes. Current Opinion in Plant Biology 13: 146–152.
Sbabou, L.; Brhada, F.; Alami, I.T.; Maltouf, A.F. 2010. Genetic Diversity of Moroccan Lupinus Germplasm Investigated using ISSR and AFLP Markers. Internat. Journal of Agricultural & Biology 12: 26-32.
Sparks, D. 2007. Advances in Agronomy. Editorial Academic Press. United Kingdom.
Sun X.L.; Yang, T.; Guan, J.P.; Ma, Y.; Jiang, J.Y.; Cao, R.; Burlyaeva, M.; Vishnyakova, M.; Semenova, E.; Bulyntsev, S.; Zong, X.X. 2012. Development of 161 novel EST-SSR markers from Lathyrus sativus (Fabaceae). American Journal of Botany 99: 379-390.
Tanksley, S. 1983. Molecular markers in plant breeding. Plant Molecular Biology Reporter 1: 3-8.
* Autor para correspondencia
E-mail: michelle.christine16@gmail.com (M.C. Chirinos-Arias).
Recibido 10 septiembre 2014
Aceptado 28 febrero 2015














