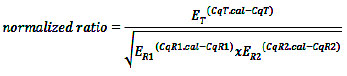Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Scientia Agropecuaria
versión impresa ISSN 2077-9917
Scientia Agropecuaria vol.7 no.spe Trujillo oct. 2016
http://dx.doi.org/10.17268/sci.agropecu.2016.03.11
ARTÍCULOS ORIGINALES
Expresión de genes quitinasas de Beauveria bassiana frente a sustratos de Fusarium oxysporum y Peronospora variabilis
Chitinases genes expression of Beauveria bassiana against Fusarium oxysporum and Peronospora variabilis substrates
Maryam Paredes*; Oscar Nolasco; Rosalyn Acuña; Ana I. F. Gutiérrez
Laboratorio de Bioquímica y Biología Molecular. Facultad de Ciencias Naturales y Matemática. Universidad Nacional Federico Villarreal, Jr. Río Chepén 290, El Agustino, Lima – Perú.
Resumen
El hongo filamentoso Beauveria bassiana es el agente de control biológico más utilizado en la actualidad, ya que es muy conocido por su actividad entomopatógena y por su referencia como hongo antagonista. El mod o de infección de B. bassiana es a través de la degradación de la quitina presente en la cutícula del insecto o pared celular de hongos patógenos cuyo mecanismo de acción involucra la actividad de las enzimas y los genes de quitinasas. La expresión de dos genes de quitinasas; el gen de la familia 18.4 (XM_008603039.1) y el gen CHIT1 (EU828354.1) de B. bassiana, se han evaluado mediante RT-qPCR a partir de RNA extraídos a los 0, 4 y 7 días, de cultivos líquidos inducidos con diferentes fuentes de carbono: quitina coloidal 1,8%, laminarina 0,1%, pulverizado de F. oxysporum 0,1% y pulverizado de hojas infectadas con P. variabilis 0,1%. La expresión relativa frente a genes referenciales de actinina y tubulina muestran que en el día 7 la expresión del gen CHIT1 incrementó en 1,87 ± 0,35 veces en los medios con F. oxysporum, mientras que el gen quitinasa de la familia 18.4 presentó mejor expresión en los medios con P. variabilis siendo su incremento de 5,88 ± 1,35 veces. Este incremento de la expresión de los genes quitinasa de la familia 18.4 (XM_008603039.1) y CHIT1 (EU828354.1) de B. bassiana sugiere que estas enzimas tienen una expresión diferencial en el desarrollo y tipo de cultivo.
Palabras claves: Quitinasa, Beauveria bassiana, Peronospora variabilis, Fusarium oxysporum, RT-qPCR.
Abstract
The Beauveria bassiana filamentous fungus is the most biological control agent frequently used today due to its entomopathogenic activity and antagonist fungi capacity. B. bassiana infects the insects through the degradation of the chitin present in the cuticle, also; affects the cell wall of pathogenic fungi. This mechanism involves enzymatic activities and activation of Chitinase genes. Gene expression of the family 18.4 (XM_008603039.1) and CHIT1 (EU828354.1) of B. bassiana, were evaluated, by RT-qPCR at 0, 4 and 7 days. The assay consists of four media with different carbon sources: colloidal chitin 1.8%, laminarin 0.1%, pulverized of F. oxysporum 0.1% and pulverized leaves infected with P. variabilis 0.1%. Relative expression using actinin and tubulin as referential genes, showed that CHIT1 gene expression increased 1.87 ± 0.35 times on day 7 in the medium with F. oxysporum, while chitinase gene 18.4 family presented the best expression in the medium with P. variabilis increasing 5.88 ± 1.35 times on day 7. This increase in the expression of 18.4 family chitinase genes (XM_008603039.1) and CHIT1 (EU828354.1) of B. bassiana suggests that these enzymes have differential expression in the developing and type of culture.
Keywords: Chitinase, Beauveria bassiana, Peronospora variabilis, Fusarium oxysporum, RT-qPCR.
1. Introducción
Las especies del género Beauveria actualmente son estudiadas por su importancia como agentes de control biológico de plagas de insectos y hongos fitopatógenos en diversos cultivos de importancia económica en aplicaciones a campo abierto (Rehner y Buckley, 2005). Beauveria bassiana actúa como parásito oportunista de hábitos patogénicos y saprófitos sobre diversos fitopatógenos limitando su actividad mediante la producción de enzimas quitinasas, glucanasas, proteasas y lipasas que son importantes para la invasión y degradación del blanco fitopatógeno (Peteira et al., 2011; Flórez et al., 2005).
Diversas enzimas han sido anotadas en estudios de genomas de B. bassiana pero su función no ha sido demostrada (Xiao et al., 2012); sin embargo, en un estudio reciente realizado por Valero y su equipo (2016) han proporcionado una visión general del actual conocimiento de los genes que contribuyen a la virulencia del hongo entomopatógeno Beauveria bassiana. En el proceso infectivo, diversas enzimas deben cumplir una función muy importante dependiendo del tipo de sustrato proporcionado por el fitopatógeno. El hongo infecta el insecto u hongo patógeno por la penetración directa de la cutícula o pared celular empleando diferentes enzimas. Las quitinasas son importantes para facilitar cada etapa de la infección del hongo, desde el proceso de germinación de la conidia, crecimiento de la hifa y hasta la penetración de esta en el organismo afectado (Ownley et al., 2010; González et al., 2010). La expresión de genes quitinasas en algunos hongos tales como Trichoderma sp. es inducida por medio de paredes celulares (cutícula), por quitina coloidal o por la ausencia de carbono, por otro lado, la presencia de altas concentraciones de glucosa o de glicerol inhibe su expresión (Felse y Panda, 1999). Dentro de los genes de quitinasa, un gen de quitinasa (Bbchit1) de Beauveria bassiana fue aislado y clonado por Fang et al. (2005). Ellos propusieron que la sobreexpresión de este gen mejora de forma significativa la virulencia de B. bassiana.
En el Perú, uno de los cultivos que está cobrando importancia es Chenopodium quinoa "quinua", el cual es un cultivo vital en la evolución socioeconómica del poblador andino. El problema de plagas y enfermedades en el bioma andino es latente; se acentúa más por el uso desmesurado e irracional de pesticidas orgánicos que alteran el equilibrio ecológico con secuelas muy negativas en la sociedad y el medio ambiente. Este cultivo está siendo afectado principalmente por dos plagas que están causando grandes pérdidas económicas en el sector agrario: la primera, la polilla Eurysacca melanocampta "q’hona q’hona" que es la principal plaga de quinua, y la segunda Peronospora variabilis, un oomycete conocido como "Downy mildew", que afecta principalmente al follaje disminuyendo su rendimiento, siendo la enfermedad más dañina de la quinua en Argentina, Bolivia, Colombia, Ecuador y Perú (Choi et al., 2010; Gandarillas et al., 2014). El objetivo de este trabajo fue comparar el nivel de expresión de genes de quitinasas Beauveria bassiana cuando es cultivado con diferentes fuentes de carbono y enfrentado con pulverizado de hojas infectadas con Peronospora variabilis y con sustrato de Fusarium oxysporum usando la técnica de cuantificación RT- qPCR de ARNm. Los resultados de este trabajo proporcionará datos para futuros ensayos de investigadores y su aplicación en el campo de la biotecnología orientada al control biológico de Peronospora variabilis con Beauveria bassiana.
2. Materiales y métodos 2.1 Cultivo del hongo
La cepa de Beauveria bassiana usada en este estudio fue obtenida del Servicio Nacional de Sanidad Agraria (SENASA- PERÚ) con código de cepa CCB-LE 262. Las conidias de Beauveria bassiana, adheridas a granos de arroz, fueron resuspendidas en medio mínimo, contadas en cámara de Neubauer y mediante diluciones sucesivas se sembraron 0, 10 y 100 conidias por placa para obtener un cultivo monospórico en agar papa dextrosa (PDA) cultivados por 5 días. Esta cepa fue replicada en medio liquido de enriquecimiento (medio mínimo suplementado con glucosa 4% (p/v), extracto de levadura 1% (p/v) y bactopeptona 1% (p/v)), el cultivo creció por 4 días hasta la fase media logarítmica con la finalidad de tener una población en desarrollo y condiciones óptimas de nutrición (Pham et al., 2009; Havukkala et al., 1993).
2.2 Cultivo en medios de inducción
Los medios de cultivo de análisis se prepararon a partir de medio mínimo (MM) (para 100 mL de MM se agregó 0,6 g de NaNO3; 0,052 g de KCL; 0,202 g de KH2PO4 y 0,102 g de MgSO4, y 40 µL de elementos trazas propuesto por Fernandes et al. (2012)), suplementada con la muestra de análisis. Los suplementos de los medios evaluados fueron: quitina coloidal 1,8% (Q), laminarina 0,1% (L), pulverizado de F. oxysporum 0,1% (F), hojas de quinua pulverizadas infectadas con P. variabilis 0,1% (P) y hojas sanas pulverizadas de quinua 0,1% (H).
La preparación de quitina coloidal se realizó de acuerdo al método propuesto por Castro et al. (2011).
Concentraciones de 107 conidias x mL-1, determinadas por conteo al microscopio en cámara de Neubauer, fueron colocadas en cada uno de los medios a evaluar (MM, Q, L, F, P, H), El cultivo se desarrolló a una agitación constante de 130 rpm durante los días de análisis a temperatura entre 25 °C y 27 °C.
2.3 Extracción de ARN y síntesis de cDNA
ARN total fue extraído del micelio colectado en los días 0, 4 y 7 de cada uno de los medios cultivados (previamente el micelio fue triturado en un mortero estéril en presencia de nitrógeno líquido hasta quedar reducido a polvo) usando el kit "High Pure RNA isolation" (ROCHE), siguiendo las indicaciones del fabricante. La concentración de ARN fue medida con el fluorómetro Qubit® 2.0 con el kit "Qubit™ RNA Assay". El cDNA fue sintetizado usando el kit "Maxima First Strand cDNA Synthesis" para RT-qPCR de Thermo Scientific. Finalmente, todos los ARNs aislados y los cDNAs fueron almacenados a -20°C antes de su uso.
2.4 Expresión de genes quitinasas por PCR en tiempo real (qPCR)
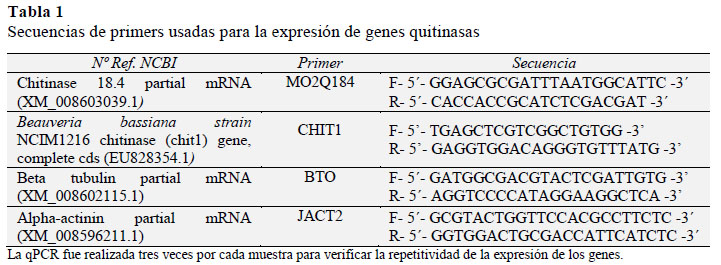
La cuantificación de los niveles de expresión de genes quitinasas fue realizada usando la técnica de PCR en tiempo real (qPCR) del cDNA sintetizado a partir del ARN extraído del micelio a 0, 4 y 7 días de cultivo de los 6 tratamientos descritos previamente. La qPCR fue realizada utilizando el Sistema de PCR en tiempo Real LightCycler® 96 (Roche). El volumen final de la reacción fue de 20 µL y contenía la siguiente mezcla de reacción: 10 µL de SYBR Green 2X Master mix (Roche), 125 nM de primer forward, 125 nM de primer reverse, agua libre de nucleasas y 5 μL de cDNA de la muestra (100 ng).
Los primers fueron diseñados de acuerdo a las secuencias reportadas en la base de datos NCBI (http://www.ncbi.nlm.nih.gov) y son mostradas en la Tabla 1. La secuencia de los productos de amplificación se confirmó por secuenciamiento estándar en ambas direcciones realizado por la compañía MACROGENE. Para la normalización de expresión de genes se utilizaron los genes de actinina y tubulina de B. bassiana como genes de referencia o housekeeping. De la misma manera el medio mínimo fue utilizado como calibrador para los medios suplementados con laminarina, quitina coloidal y F. oxysporum, y el calibrador para el medio con pulverizado de hojas infectadas con P. variabilis fue el medio que contenía medio mínimo con hojas sanas de quinua. La cuantificación se llevó a cabo en base al valor del ciclo umbral (Ct o Cq) registrado en el software LightCycler® 96 de Roche en base al modelo de cuantificación relativa normalizada con la fórmula siguiente (Pfaffl, 2004):
Dónde ET: eficiencia del gen target; ER: eficiencia del gen de referencia; CqT: ciclo umbral del gen target; CqR: Ciclo umbral del gen de referencia; Cal: calibrador de estudio.
3. Resultados y discusión
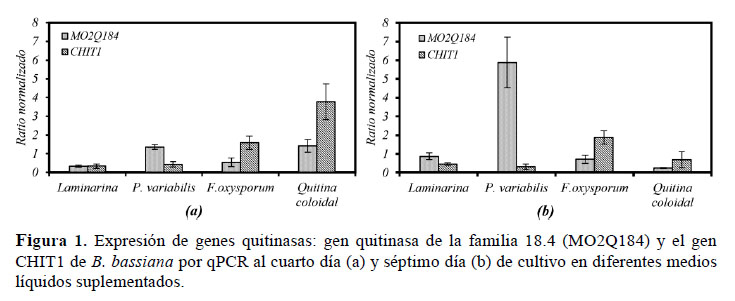
La cuantificación de la expresión del gen quitinasa de la familia 18.4 (XM_008603039.1), codificado con el primer MO2Q184, y del gen CHIT1 (EU828354.1) fue realizada por RT-qPCR, donde la expresión fue similar a los genes de referencia actinina y tubulina a nivel basal de expresión (día cero). Los resultados de la expresión son mostrados en la Figura 1 (a y b) para los genes MO2Q184 y CHIT1 en el cuarto y séptimo día, respectivamente.
Mayorga et al. (2012) mencionan que la expresión de genes de quitinasas en microorganismos es controlada por medio de un sistema inductor-represor, en donde, la quitina y/o subproductos derivados pueden participar como inductores, y que la glucosa funciona como represores del sistema. Ellos confirmaron lo expuesto anteriormente en su análisis de expresión de genes quitinasas en Lecanicillium lecanii. En nuestro estudio, el gen CHIT1 presentó niveles de expresión altos en comparación con el gen quitinasa de la familia 18.4 (MO2Q184) en medio con quitina coloidal, sugiriendo que quitina coloidal actúa como mejor inductor de genes quitinasas en B. bassiana. El gen quitinasa de la familia 18.4 (MO2Q184), al séptimo día de cultivo, se expresa entre cinco a seis veces más que los genes de referencia utilizados (actinina y tubulina) en el medio que contiene pulverizado de hojas infectadas con P. variabilis (Figura 1b). Cuando el medio suplementado con F. oxysporum fue analizado, los niveles de expresión del gen CHIT1 fue entre uno y dos veces mayor que los genes referenciales al séptimo día de cultivo (Figura 1b), mientras en el medio suplementado con quitina coloidal, su expresión es cuatro veces más que los genes de referencia al cuarto día de crecimiento del hongo (Figura 1a), pero disminuye al séptimo día de cultivo (Figura 1b). Estudios en la producción de quitinasas por B. bassiana URM2915, URM2930 y URM4548 realizados por Svedese et al. (2013) indican que la expresión es mayor durante los últimos días de incubación en el medio que contiene la cutícula de insecto como única fuente de carbono. El efecto potenciador de la cutícula o de la pared celular en la producción de quitinasa sugiere que esta enzima puede ser inducida específicamente por algún componente que es encontrado en la cutícula o pared celular.
En los dos genes evaluados en este artículo la expresión no presenta diferencias significativas al cuarto día de cultivo en laminarina (Figura 1a), sin embargo, tres días después séptimo día de evaluación) la expresión del gen quitinasa de la familia 18.4 es casi una vez más que los genes referenciales actinina y tubulina (Figura 1b). Es importante mencionar que los genes quitinasa se expresan desde el inicio de la evaluación, y que el cambio de expresión se debe al cambio de medio (sustrato o fuente de carbono), sugiriendo que existe una regulación de la expresión de dichos genes por el tipo de medio de cultivo utilizado.
4. Conclusiones
El nivel de expresión de quitinasas con Peronospora variabilis es mejor observada con el gen quitinasa de la familia 18.4.
Este incremento de la expresión de los genes quitinasa de la familia 18.4 (XM_008603039.1) (MO2Q184) y CHIT1 (EU828354.1) de B. bassiana sugiere que esta enzima está presente en ciertas etapas de desarrollo del hongo. La actividad enzimática de quitinasas de B. bassiana deberá ser analizado para relacionar su actividad entomopatógena o de micoparasitismo con la expresión de genes.
Agradecimientos
Los autores agradecen al Programa Nacional de Innovación para la Competitividad y Productividad (PNICP) y al Laboratorio de Bioquímica y Biología Molecular de la Facultad de Ciencias Naturales y Matemática de la Universidad Nacional Federico Villarreal por el Convenio N°189-FINCyT-IB-2013.
Referencias
Castro, R.; Álvarez, A.; Machado, E.; Mendoza, M.; Gómez, R.; García, P. 2011. Caracterización de una quitinasa extracelular producida por Serratia sp. BIOMI-363706 usando quitina coloidal como sustrato. Sociedad Química del Perú, 77(2): 101-108. [ Links ]
Choi, Y.; Danielsen, S.; Lubeck, M.; Hong, S.; Delhey, R.; Shin, H. 2010. Morphological and Molecular Characterization of the Causal Agent of Downy Mildew on Quinoa (Chenopodium quinoa). Mycopathologia 169: 403-412. [ Links ]
Fang, W.; Leng, B.; Xiao, Y.; Jin, K.; Ma, J.; Fan, Y.; Feng, W.; Yang, X.; Zhang, Y.; Pei, Y. 2005. Cloning of Beauveria bassiana Chitinase Gene Bbchit1 and Its Application to Improve Fungal Strain Virulence. Applied and Environmental Microbiology 71: 363–370. [ Links ]
Felse, A.; Panda, T. 1999. Regulation and cloning of microbial chitinases genes. Applied and Environmental Microbiology 51: 141-151. [ Links ]
Fernandes, E. G.; Valério, H. M.; Feltrin, T.; Van Der Sand, S. T. 2012. Variability in the production of extracellular enzymes by entomopathogenic fungi grown on different substrates. Brazilian Journal of Microbiology 43(2): 827-833. [ Links ]
Flórez, M.; López, J.; Valencia, A. 2005. Actividad de α-amilasas del hongo entomopatógeno Beauveria bassiana cultivado en medio líquido. Revista Colombiana de Entomología 31 (2):123 – 126. [ Links ]
Gandarillas, A.; Saravia, R.; Plata, G.; Reinaldo Quispe, R.; Ortiz, R. 2014. Principales plagas y enfermedades de la quinua. Capítulo Numero 2.6. In: Bazile D. et al. (Editores), "Estado del arte de la quinua en el mundo en 2013": FAO (Santiago de Chile) y CIRAD, (Montpellier, Francia): 227 – 256. [ Links ]
González, I.; Infante, D.; Peteira, B.; Martínez, B.; Arias, Y.; González, N.; Miranda, I. 2010. Caracterización bioquímica de aislamientos de Trichoderma spp. promisorios como agentes de control biológico. I. expresión de actividad quitinasa. Revista Protección Vegetal [online] 25(1): 58-63. [ Links ]
Havukkala, I.; Mitamura, C.; Hara, S.; Hirayae, K.; Nishizawa, Y.; Hibi, T. 1993. Induction and Purification of Beauveria bassiana Chitinolytic Enzymes. Journal of Invertebrate Pathology 61(1): 97-102. [ Links ]
Mayorga, L.; Calderón, E.; Gutiérrez, A.; Gonzáles, R.; Azaola, A.; Barranco, E. 2012. Characterization and expression of the chitinase chit II gene from Lecanicillium lecanii in solid-state fermentation. Revista Mexicana de Ingeniería Química 11(1): 97-104. [ Links ]
Ownley, B.H.; Gwinn, K.D.; Vega, F.E. 2010. Endophytic fungal entomopathogens with activity against plant pathogens: ecology and evolution. BioControl 55: 113-128. [ Links ]
Peteira, B.; Gonzáles, I.; Arias, Y.; Fernández, A.; Miranda, I.; Martínez, B. 2011. Caracterización bioquímica de seis aislamientos de Beauveria bassiana (BALSAMO) Vuillemin. Revista de Protección Vegetal 26(1): 16 – 22. [ Links ]
Pfaffl, M.W. 2004. Quantification Strategies in Real-Time PCR. A-Z of Quantitative PCR 1: 89-113. [ Links ]
Pham, T.A.; Kim, J.J.; Kim, S.G.; Kim, K. 2009. Production of Blastospore of Entomopathogenic Beauveria bassiana in a Submerged Batch Culture. Mycobiology 37(3): 218-224. [ Links ]
Rehner, S.; Buckley, E. 2005. A Beauveria phylogeny inferred from nuclear ITS and EF1-α sequences: evidence for cryptic diversification and links to Cordyceps teleomorphs. Mycologia 97 (1): 84 – 98. [ Links ]
Svedese, V.; Vieira, P.; Pereira, J.; Mesquita, L.; Luna, E.; Figueiredo, A. 2013. Pathogenicity of Beauveria bassiana and production of cuticle-degrading enzymes in the presence of Diatraea saccharalis cuticle. African Journal of Biotechnology 12(46): 6492 –6497. [ Links ]
Valero-Jiménez, C.A.; Wiegers, H.; Zwaan, B.J.; Koenraadt, C. J. M.; Kan, J. A. L. van. 2016. Genes involved in virulence of the entomopathogenic fungus Beauveria bassiana. Journal of Invertebrate Pathology 133: 41 – 49. [ Links ]
Xiao, G.; Ying, S-H.; Zheng, P.; Wang, Z-L.; Zhang, S.; Xie, X-Q.; Shang, Y.; St. Leger, R.; Zhao, G-P.; Wang, C.; Feng M-G. 2012. Genomic perspectives on the evolution of fungal entomopathogenicity in Beauveria bassiana. Scientific Reports 2, 483. DOI: 10.1038/srep00483.
* Corresponding author
E-mail: maryam.paredes.a@gmail.com (M. Paredes)
Received May 13, 2016.
Accepted July 18, 2016.