Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Scientia Agropecuaria
versión impresa ISSN 2077-9917
Scientia Agropecuaria vol.10 no.1 Trujillo ene./mar. 2019
http://dx.doi.org/10.17268/sci.agropecu.2019.01.05
ARTÍCULOS ORIGINALES
Densidad estomática, contenido de clorofila y relación filogenética en 17 cultivares de Musa spp.
Stomatal density, chlorophyll content and phylogenetic relationship in 17 cultivars of Musa spp.
Scott David Cruz Arévalo1; Ricardo Fernando Romero Meza1 ORCID iD https://orcid.org/0000-0002-3915-3309; Ángel Virgilio Cedeño Moreira1 ORCID iD https://orcid.org/0000-0002-6564-5569; Ángel Vinicio Verdosoto Valencia1; María Fernanda Peñafiel Jaramillo2 ORCID iD https://orcid.org/0000-0002-5369-016X; Hayron Fabricio Canchignia Martínez3,* ORCID iD https://orcid.org/0000-0003-1195-5446
1 Facultad de Ciencias Agropecuarias, Carrera de Ingeniería Agropecuaria, Universidad Técnica Estatal de Quevedo, Los Ríos-Ecuador.
2 Facultad de Ciencias Ambientales, Carrera de Ingeniería Ambiental, Universidad Técnica Estatal de Quevedo, Los Ríos-Ecuador.
3 Facultad de Ciencias Agrarias, Carrera de Agronomía, Universidad Técnica Estatal de Quevedo, Laboratorio de Microbiología Molecular del Departamento de Biotecnología, km 1.5 a Santo Domingo de los Tsáchilas, EC. 120501, Los Ríos-Ecuador.
Resumen
El objetivo del trabajo se enfatizó en estudiar los caracteres estomáticos, clorofila y relación filogenética en 17 cultivares de Musa spp. (AAB; ABB; AAA; AA), del Banco de germoplasma de la Universidad Técnica Estatal de Quevedo. Los experimentos se ejecutaron en el Laboratorio de Microbiología y Biología Molecular. Se empleó dos métodos de evaluaciones morfométricos y fisiológicos de hoja: a) De tejido foliar de 25 cm2 se evaluó la densidad estomática abaxial y adaxial en (e/mm2) y b) De discos foliares de 1,3 cm2 se determinó el contenido de clorofila Total. En caracterización molecular por RAPD se emplearon cinco oligonucleótidos. Se determinaron diferencias estadísticas significativas (p ≤ 0,05) para las variables, con resultados de mayor densidad estomática abaxial en Barraganete de 233 e/mm2 y su relación directamente proporcional entre densidad estomática y longitud, donde a mayor densidad menor su longitud. El contenido de clorofila de 4,61 mg/g en Guineo de seda. El árbol filogenético generó dos grandes Grupos a su accesión genómica. El Sub-grupo A reunió a nueve accesiones al genoma (AAB), Sub-grupo B con cinco accesiones de (AAA), destacando el Sub-grupo C al contener al cultivar Orito del genotipo (AA) y Subgrupo D con las accesiones (AAB y ABB).
Palabras clave: Musa spp.; densidad estomática; abaxial; adaxial; clorofila; RAPD.
Abstract
The objective of the work was to study stomatal characters, chlorophyll and their phylogenetic relationship in 17 cultivars of Musa spp. (AAB; ABB; AAA; AA), of the Germplasm Bank of la Universidad Técnica Estatal de Quevedo. The experiments were carried out in the Laboratory of Microbiology and Molecular Biology. Two methods of morphometric and physiological leaf evaluations are used: a) From leaf tissue of 25 cm2 the abaxial and adaxial stomatal density was evaluated in (e/mm2) and b) From leaf disks of 1,3 cm2, was determined the total chlorophyll content. In molecular characterization by RAPD, were used five oligonucleotides. Significant statistical differences were determined (p ≤ 0,05) for the variables, with results of higher abaxial stomatal density in Barraganete of 233 e/mm2 and its directly proportional relationship between stomatal density and length, where the greater the density, the shorter its length. The chlorophyll content of 4,61 mg/g in Guineo seda. The phylogenetic tree generated two large groups at its genomic accession. Sub-group A brought together nine accessions to the genome (AAB), Sub-group B with five accessions of (AAA), highlighting Subgroup C containing the cultivar Orito of the genotype (AA) and Sub-group D with the accessions (AAB and ABB).
Keywords: density stomatal; abaxial; adaxial; chlorophyll; RAPD.
1. Introducción
El cultivo de banano (Musa AAA) y plátano (Musa AAB) es de importancia mundial y constituye la mayor actividad agrícola de países tropicales (Jooste et al., 2016). Durante el año 2017 el Ecuador exportó 5, 333 toneladas (AEBE, 2018), del punto de vista socioeconómico, el plátano y banano genera fuentes de trabajo estables y transitorias (AEBE, 2018). La mayoría de estos cultivares tienen origen en dos especies silvestres: M. acuminata Colla y M. balbisiana Colla, donde por poliploida e hibridación se generó las variedades cultivables (Simmonds y Shepherd, 1955). Las enfermedades fúngicas que reducen la productividad entre un 30 a 50% de su fruta en Musa spp., es la Sigatoka Negra (SN) causado por Mycosphaerella fijiensis (Churchill, 2011), Fusarium oxysporum f. sp. cubense. (Foc4) (Ploetz, 2015).
El inicio de infección por M. fijiensis, se sitúa por encima de las aberturas estomáticas con la germinación de ascosporas (Fingolo et al., 2012). La infección primaria ocurre en el envés de la hoja y ápice por mayores densidades estomáticas, donde el hongo tiene alta probabilidad de infiltración (Washington et al., 1998). Las mayores densidades estomáticas en cultivares de Musa spp., son abaxiales y clasificándolas como anfiestomáticas (Putra et al., 2012). Estos son variables entre cultivares de Musa spp., donde le proporciona a la planta un medio para ajustarse a cambios ambientales (Nascimento et al., 2015). Teniendo diferentes grados de resistencia a SN, relacionados con la concentración de clorofila (Rodríguez y Cayón, 2008) y su capacidad fotosintética (Asmar et al., 2013). El aumento de temperatura se vincula al cierre estomático con la disminución del contenido de clorofila ocasionando la reducción en productividad (Djanaguiraman et al., 2018). Donde la variabilidad del contenido de clorofila total respondería al crecimiento vegetativo y su capacidad fotosintética (Hasanah y Sembiring, 2018). El empleo de marcadores morfológicos son limitantes para el estudio de evolución y taxonomía de Musa spp., por su diversidad genética existente (Robinson y Galán, 2012). Cultivares con distintos comportamientos agronómicos que son influenciados por factores genéticos, variaciones medios ambientales, temperatura y fertilización (Zuo et al., 2018). Los marcadores moleculares de ADN son aplicados en bancos de germoplasma de Musa spp, verificando altos niveles de similitud genética entre cultivares (Ude et al., 2002). El genotipado por marcadores moleculares de ADN proporciona una mayor resolución para el establecimiento de relaciones filogenéticas (Čížková et al., 2015).
Los estudios a diversidad genética en Musa sp., son aplicados al polimorfismo de longitud de fragmentos de restricción (RFLP), que ha derivado al agrupamiento para genomas BB, ABB y AAB (Hapsari et al., 2015). Los marcadores moleculares de variantes de PCR, como (RAPD) de las siglas en ingles ADN polimórfico amplificado al azar, son utilizados en verificar la estabalidad genética al proceso de multiplicación in vitro de Simmondsia chinensis (jojoba) (Kumar et al., 2011). Además, los RAPD son empleados en estudios de diversidad genética de Moringa oleífera (moringa), produciendo bandas polimórficas y reproducibles (Saini et al., 2013). Nuevos estudios en Musa acuminata están enfocados en identificación y caracterización de MicroRNAs (miRNAs) un rol importante en la resistencia a infecciones a patógenos (Song et al., 2018), donde los miRNA169 están involucrados al desarrollo radicular (Sorin et al., 2014), resistencia a enfermedades (Li et al., 2017). El desarrollo de marcadores SCARs (Sequence-Charaterized Amplified Regions) están desarrollados para identificar cultivares de Musa AAA Cavendish susceptible a F. oxysporum f. sp. cubense (Foc4) (Wang et al., 2018).
La existencia de un conjunto de material genético de cultivares de Musácea en Ecuador, ha requerido la necesidad de estudiar sus colecciones existentes y preservarlos en bancos germoplasma ex situ. El objetivo del trabajo se ejecutó para caracterizar el Banco de germoplasma ex situ de Musa spp., de la Finca Experimental "La María", UTEQ, Quevedo-Ecuador, empleando caracteres morfológicos sobre densidad estomática, fisiológicos al contenido de clorofila y su caracterización molecular por RAPD al estudio de su diversidad genética.
2. Materiales y métodos
El trabajo se desarrolló en en el Laboratorio de Microbiología y Biología Molecular de la UTEQ, localizado en el Campus Universitario "Manuel Haz Álvarez" en el km 1,5 vía Quevedo – Santo Domingo. Sus coordenadas geográficas son 01° 01" de latitud Sur y 79° 47" de longitud Occidental.
Material genético
Se analizaron 17 cultivares de Musa spp., del Banco de germoplasma enmarcado dentro del Proyecto de Investigación "Improving productivity and resilience for the rural poor throught enhanced use of crop varietal diversity in integrated production and pest management (IPPM)". Localizado en la Facultad de Ciencias Pecuarias, de la UTEQ. Campus Finca Experimental "La María", km 7 vía QuevedoEl Empalme (Tabla 1).
Densidad estomática
De los 17 cultivares de Musa spp., se determinó la densidad y longitud estomática, siguiendo la metodología descrita por Hernández et al. (2006), muestreando tejido foliar de 25 cm2 en su estado de floración. El tejido vegetal se preservó en un tampón FAA (Formol 75%, Etanol 10% y Ácido Acético glacial 15%). Se realizaron cortes epidérmicos de la hoja desprendiendo la cutícula abaxial y adaxial, con hojillas de afeitar. Las muestras se ubicaron al centro de la cámara Newbauer, adicionando 5 µl de agua para evitar la deshidratación del tejido epidérmico y añadiendo el cubre objeto, observados por un microscopio óptico Olympus (ocular 10X y objetivo 40X). Se contabilizaron los estomas por los 25 cuadros de 0,20 mm2 en (e/mm2) empleando la cámara Newbauer, realizando un patrón de conteo de cinco cuadrantes y registrado con fotografías con el programa ISCapture Professional Imaging.
Contenido de clorofila
La determinación de clorofila se empleó el método de extracción con etanol descrito por Wintermans y De Mots (1965), se recuperó discos foliares de 1,3 cm2 de la zona media de la tercera hoja emitida a la hoja cigarro en la etapa de formación de la bellota. Se maceró el disco foliar en un nortero con 4 ml del tampón que contenía etanol frio a 98% + MgCO3 (0,5 g L-1). Se transfirió a tubos falcon de 13 ml; enjuagando el mortero con un mismo volumen de la solución para completar 8 ml y centrifugado a 3000 rpm por 5 min, el sobrenadante se examinó por absorbancia de 649 y 665 nm en un espectrofotómetro "UNICO" modelo 1205. A partir de esos datos se calcularon las concentraciones de clorofila a (Cla), clorofila b (Clb) y clorofila total (ClT), según las siguientes formulas: Cla = [(13,7 x A665) (5,76 x A649)] x V/PD; Clb = [(25,8 x A649) (7,6 x A665)] x V/PD; ClT = [(6,10 x A665) + (20,04 x A649)] x V/PD. Donde: V = volumen final extracto etanólico (8,0 ml); PD = peso seco disco foliar (g).
Extracción de ADN genómico y determinación de los RAPD
La extracción de ADN de los 17 cultivares de Musa spp., se realizó siguiendo las intrusiones del fabricante por el kit QuickStart Protocol (QIAGEN), partir de 1 gr de tejido foliar se pulverizó con N2 lÃquido. Se verificó la calidad del ADN, con 2 µl de ADN y 1 µl del tampón de carga de Blue Juice (10X) (Invitrogen), en gel de agarosa al 1% y teñidos en bromuro de etidio. Se emplearon los oligonucleótidos decámeros con mayor polimorfismo en Musáceas reportados por Nadal et al. (2009), considerando las cinco combinaciones de mayor número de bandas polimórficas (Tabla 2).
Los componentes para el mix de la PCR, se realizó un volumen de 20 µL: ADN genómico 2 µL; 4 µL de buffer 5x Cl2 Mg; 1 µL de DNTP; 0,2 µL de TAQ pol; 1 µL de los iniciadores (sentido y antisentido); 10,8 µL de agua ultra-pura. Se emplearon los reactivos de PROMEGA. El perfil térmico empleado en PCR: 94 °C por 8 min; 40 ciclos a 94 °C por 1 min; 37 °C por 2 min a 72 °C por 2 min; extensión final de 72 °C por 10 min. Los productos de PCR amplificados se verificaron por electroforesis en gel de agarosa al 2,5% y teñidos en bromuro de etidio, a 70 V por 150 min., y documentado por Technologies Thermo Fisher Scientific. La información binaria de (0 y 1), se reemplazó por las letras (A y T) de los perfiles generados por RAPD. La historia evolutiva se infirió empleando el método UPGMA, el árbol filogenético se construyó empleando la distancia evolutiva de Kimura 2-parametro con el programa MEGA 6.06 (Tamura et al., 2013). Los grupos de cada nodo se fortalecieron por un análisis de muestreo con (Bootstrpping, 3000 repeticiones) para un dendrograma estadísticamente robusto.
Análisis de datos
Se registró la densidad estomática abaxial y adaxial en los cultivares de Musáceas, con 17 tratamientos los que contenían cuatro replicas experimentales y cada una con cinco unidades experimentales. Al contenido de clorofila se analizó con tres replicas y cuatro unidades experimentales. Aplicando un DCA los valores a cada condición están representados con la desviación estandar promedio individual (±), los tratamientos fueron sujetos al aná-lisis de varianza por ANOVA, y separados por procedimiento de comparación múltiple de Tukey SD, al nivel de significancia de (P≤0.05), empleando Statgraphics.
3. Resultados y discusión
Densidad estomática abaxial
Los estudios de morfología estomática de los cultivares de Musa spp., del Banco de Germoplasma, de clones: Orito; Williams; Vinces; Barraganete; Curare; Limeño se ilustran en la (Figura 1). Verificando diferencias significativas entre los cultivares en la densidad estomática abaxial Figura 2, en los cultivares a M. balbisiana se observó la mayor densidad de (233 y 199 e/mm2) en los clones Barraganete y Cuatrofilos, caso contrario considerando al Maqueño morado y Manzano como los cultivares de menor densidad estomática con (128 y 139 e/mm2). Estos rangos en densidades estomáticas coincide con los cultivares de accesión a los genomas (AAB y ABB) de clones a plátano África y Cachaco con (215 y 133 e/mm2) (Hernández et al., 2006). En los cultivares a M. acuminata se observó la mayor densidad de (185 y 174 e/mm2) en Orito y Guineo de seda. En todos los cultivares de Musa spp., se verificaron densidades estomáticas superiores en la superficie abaxial, mecanismo que ayudaría a prevenir la pérdida de agua por estar menos expuesto a radiación solar (Martin y Glover, 2007). Las menores densidades estomáticas de (120 y 122 e/mm2) se reflejan en Guineo jardín y Vinces (Figura 2).
Esta disminución en densidad estomática de estos clones posibilita una mejor adaptación a estrés hídrico. La densidad estomática abaxial es tan variable que son influenciados por el porcentaje y cambios de exposición de luz (azul con 269 y roja con 200 e/mm2) en Lactuca sativa L (Wang et al., 2016).
Densidad estomática adaxial
En densidades estomáticas adaxiales se determinaron diferencias significativas entre los cultivares Figura 2, observando en M. acuminata el mayor número de estomas de 58 e/mm2 en Williams. Considerando con índices promedio entre (42 y 50 e/mm2) en los cultivares de Guineo jardín, Filipino, Orito y Guineo de seda.
En M. balbisiana, las mayores densidades estomáticas con (34, 41 y 43 e/mm2), en Dominico gigante, Cuatrofilos y Manzano. Esta característica se considera como un parámetro que regularía un grado de tolerancia a M. fijiensis. Donde clones de plátano con resistencia a la enfermedad presentan menor densidad estomática adaxial en sus hojas (Hernández et al., 2006). Los clones a M. balbisiana y sus densidades estomáticas no superaron los 43 e/mm2, esto posibilita la capacidad de adaptación en condiciones de sequía, cuando las tasas de evapotranspiración son más altas. Las especies vegetales con disminuciones en densidades estomáticas incrementan su resistencia, limitando el exceso de transpiración (Lawson y Blatt, 2014). En los plátanos como: Barraganete, Limeño, Dominico verde, negro y Hartón resultaron con bajas densidades estomáticas adaxiales que no superan los 14 e/mm2, parámetro importante en la ecofisiología de las plantas para mitigar el estrés hídrico, esta característica permite controlar la perdida de agua por transpiración y la ingesta de CO2 (Alvarez et al., 2018).
Longitud estomática abaxial y adaxial
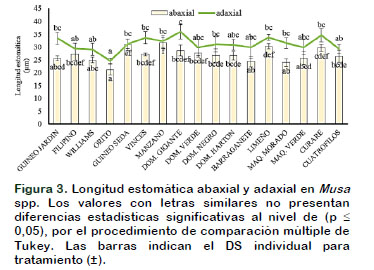
Las características morfométricas a longitud estomática abaxial, se encontró diferencias significativas entre los cultivares a M. acuminata para el clon Guineo de seda mostró la mayor longitud estomática de 30,86 µm. En M. balbisiana los plátanos como el Manzano y Limeño se observan longitudes de 31,52 y 30,65 µm (Figura 3). Los cultivares como: (Williams, Guineo de jardín, Barraganete, Cuatrofilos, Maqueño morado y verde) se observó la disminución entre (23,91 y 26,37 µm), este aspecto morfológico del estoma le puede conferir una ventaja de adaptabilidad en ambientes de alta irradiación, donde estomas de menor tamaño pueden abrir y cerrar más rápidamente, siendo más eficientes en los procesos de fotosíntesis y respiración (Hetherington y Woodward, 2003).
En longitud adaxial se observaron diferencias significativas entre los cultivares, donde M. balbisiana se verifica la mayor longitud estomática de 35,9 µm en el clon Dominico gigante y los cultivares a M. acuminata de 33,56 µm clon Vinces (Figura 3). Concordando con lo observado por Sumardi y Wulandar (2010), en los cultivares Ambon warangan y Kluthuk susu al genoma (AAA y ABB) con longitudes de (33,88 y 32,03 µm). Los reportes a disminución longitudinal estomática adaxial de 24,56 µm, se observó en el clon Orito de accesión de genoma (AA), carácter morfológico vinculado con el nivel de ploidía. Sumardi y Wulandar (2010) observan en los cultivares Penjalin (AA) y Kluthuk warangan (BB) longitudes estomáticas adaxiales de (27,41 y 26,48 µm). Esto demuestra que existe una relación directamente proporcional entre la densidad estomática y la longitud del estoma, postulando que los cultivares de mayor densidad de estomática es menor su longitud.
Contenido de Clorofila Total
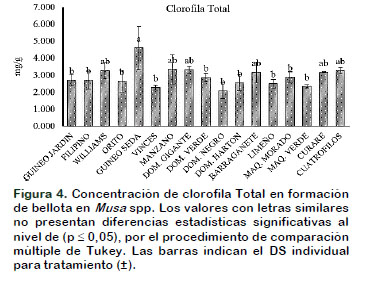
En todos los cultivares de Musa spp del Banco de germoplasma se reportaron niveles bajos en clorofila Total, por el daño causado al tejido foliar por incidencia de M. fijiensis. El análisis al contenido de clorofila se observó diferencias significativas entre los cultivares donde el Guineo de seda reportó el mayor contenido de 4,61 mg/g (Figura 4). La reducción de la concentración de clorofila por M. fijiensis coincide con el segundo estadio de la enfermedad, donde la degradación de las células del mesófilo y daños al tejido foliar ocasionan la disminución en los procesos de transpiración y fotosíntesis (Cayón, 2001). Otro parámetro a considerar en la disminución al contenido de clorofila especies vegetales es por efecto del aumento de la temperatura cuando estos encuentran entre 30/36 °C (Kumari et al., 2015), donde los cultivares de Musa sp., se desarrollaron a 31 °C promedio.
El Banco de Germoplasma estaba dirigido a estudiar los procesos de interacción de los distintos cultivares y su grado de tolerancia a problemas fitopatológicos. Donde los niveles de clorofila Total no superando los 3,34 mg/g en los cultivares de M. balbisiana (Figura 4). Además, la existencia de una población 16000 nematodos por gramo de raíces a Meloidogyne spp., cabe resaltar que la presencia de M. incognita en banano ocasiona la disminución del contenido de clorofila Total a 7,2 mg/g y su eficiencia fotosintética (Devrajan et al., 2003).
Análisis de polimorfismo por PCR-RAPD y su diversidad genética
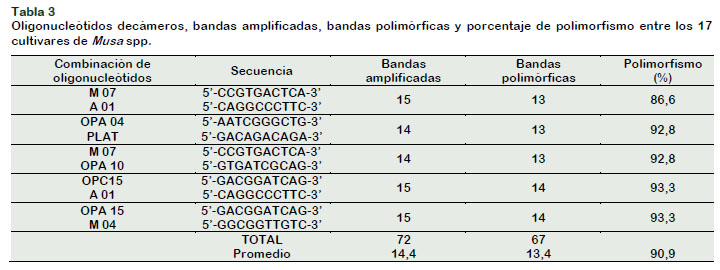
De cinco combinaciones de iniciadores se generó 72 bandas amplificadas y 63 bandas polimórficas en los 17 cultivares de Musa spp. El patrón generado por los marcadores RAPD mostró un rango de 14 productos amplificados por la combinación (OPA04 PLAT), 15 bandas amplificadas con (M07 A01), definiendo de 86,6 a 93,3% bandas polimórficas entre las 17 accesiones de bananos y plátanos estudiados (Tabla 3).
La temperatura de alineamiento de los oligonucleótidos decámeros se ajustó a 37 °C, esto coincide con temperaturas de alineamiento de los oligonucleótidos 35 °C a 45 °C en trabajos de aplicación de RAPD para estudios en Musa spp y sus relaciones filogenéticas en accesiones genómicas de cultivares AA, AAB y BB (Phothipan et al., 2005).
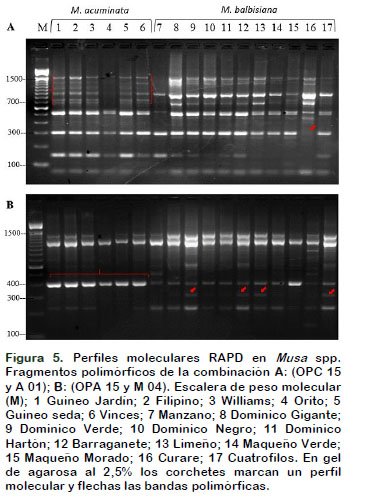
El empleo de la combinación (OPC 15 con A01) ha derivado un perfil de bandeo único entre 700 y 1500 pb, para los cultivares de M. acuminata. En el cultivar de plátano Curare pude ser distinguido de otros cultivares por la ausencia de la banda de 300 pb (Figura 5A). La amplificación por la combinación (OPA 15 con M 04) en todos los cultivares a M. acuminata se verificó la banda muy marcada de 400 pb. Y en los cultivares a M. balbisiana como: (Dominico verde, Barraganete, Limeño y Cuatrofilos) originó un producto de amplificación específico de 300 pb (Figura 5B).
Las combinaciones de los oligonucleótidos (OPC 15 con A 1) y (OPA 15 con M 04) han resultado ser exitosos para poder revelar e identificar perfiles genéticos específicos entre los cultivares de M. acuminata y M. balbisiana. Obteniendo 14 marcadores polimórficos entre las 17 accesiones de banano y plátano. Esto coincide con Nadal et al. (2009) donde la combinaciones de los oligonucleótidos encontraron 10 bandas polimórficas en cultivares de M. balbisiana y M. acuminata.
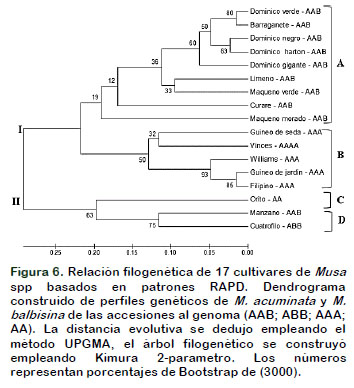
Los resultados de los perfiles genéticos obtenidos por RAPD, generó el árbol filogenético en base al modelo Kimura 2Parametros donde su dendrograma se fortaleció por un Bootstrap de 3000 repeticiones. Esto procedió a la formación de dos Grupos principales y cuatro subgrupos para las relaciones evolutivas de las accesiones al genoma de Musáceas (AAB; ABB; AAA; AA). El Grupo I, reunió al Sub-grupo A con nueve accesiones para el genoma (AAB) a M. balbisiana, para los cultivares (Dominico verde, Barraganete, Dominico negro, Dominico hartón y Dominico gigante), de mayor homología genética. Y con otra segunda ramificación con los cultivares (Limeño, Maqueño verde, Curare, Maqueño morado) (Figura 6). El Sub-grupo B con cinco accesiones para el genoma (AAA) a M. acuminata de mayor relación filogenética para los cultivares (Guineo de seda, Vinces, Williams, Guineo de jardín y Filipino). La accesión al genoma (AAA) corresponden a cultivares susceptibles a M. fijiensis, demostrando la eficiencia de la combinación de oligonucleótidos decámeros en el agrupamiento filogenético del Banco de germoplasma. Esto difiere a los resultados obtenidos por Phothipan et al. (2005), ellos no llegan a clarificar las diferencias a su agrupamiento de Musáceas entre los genomas ABB y AA.
El Grupo II reunió al Sub-grupo C, que se destaca al mantener al cultivar Orito como el único perfil genético para la accesión del genoma (AA). El Sub-grupo D están relacionados filogenéticamente a cultivares de plátano como el Manzano y Cuatrofilo para la accesión del genoma (AAB y ABB) (Figura 6). Estos resultados demostraron ser claros para la identificación de cultivares comerciales e híbridos de Musa, en base a su información genética. La selección de los cinco oligonucleótidos decámeros empleados en la técnica de biología molecular RAPD permitió afianzar los resultados de agrupamiento en base a sus perfiles genéticos, diferenciándose de los estudios a caracteres morfológicos, donde el fenotipo varía en respuesta al factor medio ambiental donde se desarrolla.
4. Conclusiones
La mayor densidad estomática abaxial se verifica en M. balbisiana clon Barraganete y la menor longitud estomática adaxial el cultivar Orito de genoma (AA) con 24,56 µm., vinculado con el nivel de ploidía, este aspecto le puede conferir una ventaja de adaptabilidad en ambientes de alta irradiación. Al empleo de marcadores RAPD es una herramienta útil para caracterizar la diversidad genética en M. acuminata y M. balbisiana, coinciden con la clasificación morfológica existente. El dendrograma mostró dos grupos, donde el Sub-grupo A reunió el 52% al genoma (AAB), el Sub-grupo B reúne el 29% al genoma (AAA). El cultivar Orito con accesión de genoma (AA) se encuentra agrupado alejada del resto de las accesiones analizadas.
Agradecimientos
Se agradece a Carmita Suárez Capello, al facilitar el material genético de estudio del Banco de germoplasma de Musa spp, por Bioversity International: (IPPM) y financiado por el Proyecto FOCICYT 4: (Actividad nematicida a Radopholus similis por rizobacterias nativas y comerciales del género Pseudomonas spp) otorgado por la Universidad Técnica Estatal de Quevedo (2016-2017).
Referencias bibliográficas
AEBE. 2018. Asociación de Exportadores de Banano Del Ecuador. PROCARSA. Disponible en: http://banano.ebizor.com/2017-fue-bueno-paraexportacion-de-banano-desde-ecuador/ [ Links ]
Alvarez, A.; Morales, R.; Corrales, R.; Avendaño, C.; Rubio, H. Villareal, F. 2018. Stomatal characterization, chlorophyll concentration and their relation with biomass production of Bouteloua Curtipendula. Agronomia Mesoamericana 29(2): 251–261. [ Links ]
Asmar, S.; Castro, E.; Pasqual, M.; Pereira, F.; Soares. J. 2013. Changes in leaf anatomy and photosynthesis of micropropagated banana plantlets under different Silicon Sources. Scientia Horticulturae 161: 328–332. [ Links ]
Cayón, G. 2001. Estimación del desarrollo de las raíces a partir de los caracteres de los brotes en banano y plátano (Musa spp.). INFOMUSA 10(2): 12–17. [ Links ]
Churchill, A. 2011. Mycosphaerella fijiensis, the black leaf streak pathogen of banana: progress towards understanding pathogen biology and detection, disease development, and the challenges of control. Molecular Plant Pathology 12(4): 307–328. [ Links ]
Čížková, J.; Hřibová. E.; Christelová, P.; Van den Houwe, I.; Häkkinen, M.; Roux, N.; Swennen, R.; Doležel. J. 2015. Molecular and cytogenetic characterization of wild musa species. PLOS ONE 10(8): 1–19. [ Links ]
Devrajan, K.; Rajendran, G. y Seenivasan. N. 2003. Nutrient status and photosynthetic efficiency of banana (Musa sp.) influenced by Meloidogyne incognita infected with Pasteuria penetrans. Nematology 31: 197–200. [ Links ]
Djanaguiraman, M.; Boyle, D.; Welti, R.; Jagadish, S.; Prasad. C. 2018. Decreased photosynthetic rate under high temperature in wheat is due to lipid desaturation oxidation, acylation, and damage of organelles. BMC Plant Biology 18(55): 1–17. [ Links ]
Fingolo, E.; Catharina, E.; Braga, J.; Vieira, A.; Moura, M.; Kaplan. M. 2012. The natural impact of banana inflorescences (Musa acuminata) on Human Nutrition. Anais da Academia Brasileira de Ciencias 84(4): 891–898. [ Links ]
Hapsari, L.; Wahyudi, D.; Azrianingsih, R.; Arumingtyas, E. 2015. Genome identification of bananas (Musa L.) from East Java Indonesia assessed with PCR-RFLP of the internal transcribed spacers nuclear ribosomal DNA. International Journal of Biosciences (IJB) 7(3): 42–52. [ Links ]
Hasanah, Y.; Sembiring. M. 2018. Role of elicitors in chlorophyll content and stomatal density of soybean cultivars by foliar application. Journal of Agronomy 17(2): 112–117. [ Links ]
Hernández, F., Portillo, M.; Navarro, C.; Rodríguez, M.; Velazco. J. 2006. Stomatal density in plantains materials (Musa AAB, AAAB and ABB) susceptible and resistant to black sigatoka (Mycosphaerella fijiensis, Morelet). Rev. Fac. Agron. 23: 291–296 [ Links ]
Hetherington, D.; Alistair, M.; Woodward.F. 2003. The role of stomata in sensing and driving environmental change. Nature 424: 901. [ Links ]
Jooste, A., Wessels, N.; van der Merwe. M. 2016. First report of banana bunchy top virus in banana (Musa spp.) from South Africa. Plant Disease 100(6): 1251. [ Links ]
Kumar, S.; Mangal, M.; Dhawan, A.; Singh, N. 2011. Assessment of genetic fidelity of micropropagated plants of Simmondsia Chinensis (Link) Schneider using RAPD and ISSR markers. Acta Physiologiae Plantarum 33(6): 2541–2545. [ Links ]
Kumari, D.; Bode, S.; Ottosen, C.; Rosenqvist. E. 2015. Wheat cultivars selected for high Fv/Fm under heat stress maintain high photosynthesis, total chlorophyll, stomatal conductance, transpiration and dry matter. Physiologia Plantarum 153:284–298. [ Links ] Lawson, T.; Blatt, M. 2014. Stomatal size, speed, and responsiveness impact on photosynthesis and water use efficiency. Plant Physiology 164(4): 1556– 1570. [ Links ]
Li, Y.; Zhao. S.; Li. J.; Hong. X.; Wang, H.; Long. X.; Ju, Y.; Xue. Z.; Yuan, Z.; Yang, N.; Fan, J.; Huang. F.; Ming W. 2017. Osa-MiR169 negatively regulates rice immunity against the blast fungus Magnaporthe oryzae. Frontiers in Plant Science 8(January): 1–13. [ Links ]
Martin, C.; Glover. B. 2007. Functional aspects of cell patterning in aerial epidermis. Current Opinion in Plant Biology 10(1): 70–82. [ Links ]
Nadal, R.; Manzo, G.; Orozco, J.; Orozco, M.; Guzmán, S. 2009. Diversidad genética de bananos y plátanos (Musa spp.) determinada mediante marcadores RAPD. Revista Fitotecnia Mexicana 32(1): 1–7. [ Links ]
Nascimento, L.; Pacheco, H.; Goulart, K.; Correa, C.; Scherer, R.; Almeida, D.; Guerra. M. 2015. Light-emitting diodes (LED) increase the stomata formation and chlorophyll content in musa acuminata (AAA) ‘Nanicão Corupá’ in vitro plantlets. Theoretical and Experimental Plant Physiology 27(2): 91–98.
Phothipan, S.; Benchamas, S.; Kawit, W. 2005. Genetic relationship among bananas in AA AAB and BB groups using random amplified polymorphic DNA (RAPD) and sequence related amplified polymorphism (SRAP) techniques. Kasetsart Journal 710: 703–710. [ Links ]
Ploetz, C. 2015. Fusarium wilt of banana. Phyto-pathology 105(12): 1512–1521. [ Links ]
Putra, E.; Zakaria, W.; Abdullah, N.; Saleh. G. 2012. Stomatal morphology, conductance and transpiration of Musa sp. Cv. rastali in relation to magnesium, boron and silicon availability. American Journal of Plant Physiology 7(2): 84–96. [ Links ]
Robinson, J.; Galán. V. 2012. Plátanos y Bananas. Mundi-Prensa. 345 pp. [ Links ]
Rodríguez, P.; Cayón. G. 2008. Efecto de Mycosphaerella fijiensis sobre la fisiología de la hoja de banano. Agronomia Colombiana 26(2): 256– 265. [ Links ]
Saini, R.; Saad, K.; Ravishankar, G.; Giridhar, P.; Shetty. N. 2013. genetic diversity of commercially grown Moringa oleifera Lam. cultivars from India by RAPD, ISSR and cytochrome P450-based markers. Plant Systematics and Evolution 299(7): 1205–1213. [ Links ]
Simmonds, N.; Shepherd. K. 1955. The taxonomy and origin of the cultivated Bananas. Botanical Journal of the Linnean Society: 302–312. [ Links ]
Song, S.; Xu, Y.; Huang. D.; Ashraf, M.; Li, J.; Hu, W.; Zhiqiang, J.; Zeng, C.; Tang, F.; Xu. B.; Zeng, H.; Li, Y.; Xie, J. 2018. Identification and characterization of mirna169 family members in banana (Musa acuminata L.) that respond to Fusarium oxysporum f. sp. cubense infection in banana cultivars. Peer J: 2–17. [ Links ]
Sorin, C.; Declerck, M.; Christ, A.; Blein, T.; Ma, L.; Lelandais, C.; Njo, M.; Beeckman, T.; Crespi, M.; Hartmann, C. 2014. A MiR169 isoform regulates specific NF-YA targets and root architecture in Arabidopsis. New Phytologist 202(4): 1197–1211. [ Links ]
Sumardi, I.; Wulandar. M. 2010. Anatomy and morphology character of five indonesian banana cultivars (Musa spp.) of different ploidy level. Biodiversitas 11(4): 167–175. [ Links ]
Tamura, K.; Stecher, G.; Peterson, D.; Filipski, A.; Kumar, S. 2013. MEGA6: Molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution 30(12): 2725–2729. [ Links ]
Ude, G.; Pillay, M.; Nwakanma, D. y Tenkouano. A. 2002. Genetic diversity in Musa acuminata Colla and Musa balbisiana Colla and some of their natural hybrids using AFLP markers. Theoretical and Applied Genetics 104(8): 1246–1252. [ Links ]
Wang, F.; Xia, L.; Xu, C.; Niu, Y.; Liu, W.; Zeng, L.; Zhou, J.; Hu. B. 2018. Development of a mitochondrial SCAR marker related to susceptibility of banana (Musa AAA Cavendish) to Fusarium oxysporum f. sp. cubense race 4. Notulae Botanicae Horti Agrobotanici Cluj-Napoca 46(2): 509–516. [ Links ]
Wang, J.; Lu, W.; Tong, Y.; Yang, Q.; Luna. A. 2016. Leaf morphology, photosynthetic performance, chlorophyll fluorescence, stomatal development of lettuce (Lactuca sativa L.) exposed to different ratios of red light to blue light. Frontiers in Plant Science 7: 1–10. [ Links ]
Washington, J.; Cruz, J.; López, J.; Fajardo. M. 1998. Infection studies of Mycosphaerella fijiensis on banana and the control of black sigatoka with chlorothalonil. Plant Dis. 82: 1185–1190. [ Links ]
Wintermans, J.; De Mots, A. 1965. Spectrophotometric characteristics of chlorophylls a and b and their phenophytins in ethanol. Biochimica et Biophysica Acta Biophysics Including Photosynthesis 9(2): 448–453. [ Links ]
Zuo, C. Deng, G.; Li, B.; Huo. H.; Li, C.; Hu, C.; Kuang, R.; Yang, Q.; Dong, T.; Sheng, O.; Yi, G. 2018.
Germplasm screening of Musa spp. for resistance to Fusarium oxysporum f. sp. cubense tropical race 4 (Foc TR4). European Journal of Plant Pathology 4: 1–6.
Received August 7, 2018.
Accepted February 15, 2019.
* Corresponding author
E-mail: hcanchignia@uteq.edu.ec (H. Canchignia).