Introducción
La quinua (Chenopodium quinoa Willd) es un importante cultivo nativo andino (Graf et al., 2016), originaria del Ande Sudamericano (Nuñez, 2015; Vidueiros et al., 2015), fue domesticada hace más de 7000 años (Bazile et al., 2016a). Ha sido protegida y conservada por las poblaciones indígenas de América del Sur (Bojanic, 2011). Está adaptada para crecer en una gran variedad de sistemas agrícolas, particularmente en la meseta andina a más de 3500 m.s.n.m. y ha desarrollado tolerancia a varios tipos de estrés abiótico como a la helada, sequía, salinidad (Ruiz, 2016) y a diferentes tipos de suelo (Bazile et al., 2016a). Sin embargo, presenta baja tolerancia al calor (Bazile et al., 2016a), pero buenas cualidades nutricionales (Ahumada et al., 2016); contiene entre 13,81 y 21,9% de proteínas, más aún la calidad de estas son del tipo albúmina y globulina (Nuñez, 2015). Es considerada como el único alimento vegetal que contiene todos los aminoácidos esenciales y es rica en vitaminas, minerales, ácidos grasos y fibra (Avalos, 2016; FAO, 2018), lo que podría resolver problemáticas de seguridad alimentaria (Bazile et al., 2016b). En las últimas décadas ha surgido el interés por cultivar la quinua fuera de Sudamérica (Bazile y Baudron, 2014; Ramírez et al., 2016). Esto, debido a su riqueza nutricional, así como a su habilidad para adaptarse a un amplio rango de condiciones agroecológicas y a su alto nivel de resistencia en ambientes marginales (Iqbal, 2015). En el año 2013, a la quinua se le otorgó un alto perfil como cultivo con el potencial de crecer en importancia en la agricultura mundial (Ruiz et al., 2016).
La habilidad adaptativa a diferentes ambientes del cultivar dentro de una especie radica en gran medida a su diversidad genética (Gilchrist et al., 2006). En Sudamérica el mejoramiento genético de la quinua se ha enfocado en desarrollar variedades de semillas grandes, de tipo dulce y amarga, con un mayor potencial de rendimiento (Zurita-Silva et al., 2014). Se han realizado estudios sobre los efectos del estrés de sequía temprana en quinua (Morales et al., 2011) y sobre el impacto del estrés hídrico y salino en las plantas de quinua (Aly et al., 2018). Sin embargo, no hay estudios realizados a la fecha que se enfoquen a profundidad sobre los genes que son resistentes a la sequía en esta especie. Por otro lado, la quinua es un alotetraploide con un número básico de cromosomas de 9 (2n = 4x = 36), con un tamaño genómico de aproximadamente 1,5 Gbp (Kolano et al., 2016), que exhibe herencia disómica para la mayoría de los caracteres cualitativos y es principalmente autógama (Ward, 2000). En las plantas superiores el estudio de herencia monoparental y biparental se ha basado en estudios de genes del cloroplasto, dado que por su haploidía presenta una sola copia (Mulo et al., 2009). El objetivo de este trabajo fue identificar los genes relacionados con la tolerancia a la sequía en la quinua (Chenopodium quinoa Willd).
Materiales y métodos
La presente investigación fue conducida en el Invernadero del Centro Experimental y Laboratorio Multipropósito del Instituto Nacional de Innovación Agraria (INIA), distrito de La Molina, departamento de Lima, Perú. Localizado a 12° 4' 36,03'' S y 76° 56' 42,857'' O con una altitud de 112 m.s.n.m.
Tabla 1 Lista de variedades de plantas
| N° | Código | Nombre | Región | País |
| 1 | cq01 | Red head | USA | |
| 2 | cq02 | INIA 431 - Altiplano | Puno | Perú |
| 3 | cq03 | Illpa INIA | Puno | Perú |
| 4 | cq04 | INIA 415 - Pasankalla | Puno | Perú |
| 5 | cq05 | Salcedo INIA | Puno | Perú |
| 6 | cq06 | INIA 420 - Negra Collana | Puno | Perú |
| 7 | cq07 | Blanca de Junín | Junín | Perú |
| 8 | cq08 | Huancayo | Junín | Perú |
| 9 | cq09 | INIA 433 - Santa Ana/AIQ/FAO | Junín | Perú |
| 10 | cq10 | Hualhuas | Junín | Perú |
| 11 | cq11 | Compuesto A | Junín | Perú |
| 12 | cq12 | Compuesto B | Junín | Perú |
| 13 | cq13 | Chucuito | Lima | Perú |
| 14 | cq14 | Compuesto Tolerante a Sequía Rojo | Puno | Perú |
| 15 | cq15 | Compuesto Tolerante a Sequía Amarillo | Puno | Perú |
| 16 | cq16 | Compuesto Tolerante a Sequía Rosado | Puno | Perú |
| 17 | cq17 | Compuesto Tolerante a Sequía Anaranjado | Puno | Perú |
| 18 | cq18 | Compuesto Tolerante a Sequía Blanco grano grande | Puno | Perú |
| 19 | cq19 | Compuesto Tolerante a Sequía Blanco grano pequeño | Puno | Perú |
| 20 | cq20 | Misquinua | Puno | Perú |
| 21 | cq21 | Compuesto Tolerante a Sequía Purpura | Puno | Perú |
| 22 | cq22 | Compuesto Tolerante a Sequia Rosado medio | Puno | Perú |
| 23 | cq23 | Compuesto Tolerante a Sequía negro | Puno | Perú |
| 24 | cq24 | Quillahuaman | Cusco | Perú |
| 25 | cq25 | Amarillo Marangani | Cusco | Perú |
| 26 | cq26 | Amarillo Sacaca | Cusco | Perú |
| 27 | cq27 | INIA 433 - Santa Ana/AIQ/FAO | Cusco | Perú |
| 28 | cq28 | INIA 431 - Altiplano | Cusco | Perú |
| 29 | Cq29 | Salcedo INIA | Cusco | Perú |
| 30 | cq30 | Illpa INIA | Lima | Perú |
| 31 | cq31 | INIA 415 -Pasankalla | Cusco | Perú |
| 32 | cq32 | INIA 420 - Negra Collana | Cusco | Perú |
| 33 | cq33 | Blanca de Junín | Cusco | Perú |
| 34 | cq34 | Hualhuas | Cusco | Perú |
| 35 | cq35 | Huancayo | Cusco | Perú |
| 36 | cq36 | Kancolla 1 | Puno | Perú |
| 37 | cq37 | Kancolla 2 | Puno | Perú |
| 38 | cq38 | Kancolla 3 | Puno | Perú |
| 39 | Cq39 | Kancolla 4 | Puno | Perú |
| 40 | cq40 | Real Boliviana Amarillo | Puno | Bolivia |
| 41 | cq41 | Real Boliviana Rojo | Puno | Bolivia |
El material vegetal utilizado fue de la especie Chenopodium quinoa Willd, obtenida de la Colección Nacional de Germoplasma de Quinua del Perú (Tabla 1). El experimento se condujo en invernadero bajo iguales condiciones de temperatura, humedad e iluminación. Las macetas utilizadas eran de 22 cm de profundidad x 29 cm de diámetro, el suelo utilizado fue esterilizado y estuvo conformado por abono orgánico y vermiculita (~ 280 g/maceta).
Se cultivaron 41 variedades de Chenopodium quinoa Willd, cinco semillas de cada variedad en una maceta, se realizó seis réplicas (30 plantas por accesión). Las plantas recibieron el primer día una irrigación de aproximadamente (200 ml/bote), Además se añadió, cada 48 horas, una solución de agua hidropónica (100 ml/bote). 27 días después de la siembra, de las seis macetas de cada variedad, se seleccionaron al azar tres macetas para ser expuestas a una sequía completa (tratamiento) durante dos semanas. Luego de ese periodo se reanudó el riego regular. Las tres macetas restantes (control) continuaron con el sistema de riego.
La clasificación de las 41 variedades como tolerantes o susceptibles a la sequía se basó en la comparación del índice de contenido clorofila (ICC) del control y del tratamiento de cada variedad. El ICC fue medido en prefloración, floración y posfloración utilizando el medidor MC-100 CCI (instrumentos Apogee). Se realizaron tres comparaciones de cada variedad: control de prefloración-posfloración y sequía posfloración.
Secuenciación de ARN
El ARN total fue extraído a partir de las hojas usando el reactivo reagent(r) TRI, siguiendo el protocolo de la compañía proveedora. Sin embargo, en el paso final del protocolo se realizó una precipitación adicional con acetato de sodio pH 5,2. Las librerías se prepararon a partir de ARN total utilizando el kit TruSeq Strandedm RNA (Illumina) siguiendo las recomendaciones del fabricante. La secuenciación se realizó con el sistema NovaSeq 6000 (Illumina) por 200 ciclos (2 x 100 pb).
Análisis diferencial de expresión génica
Los adaptadores fueron seleccionados y retirados junto con los tramos de baja calidad usando el programa Trimmomatic v0.36 (Bolger et al., 2014), con los parámetros siguientes SLINGWINDOWS: 4: 15 y MINLEN: 75. Las librerías limpias se evaluaron con el programa FastQC (Wingett y Andrews, 2018) y se mapeó al genoma de referencia de Chenopodium quinoa Cq_PI614886_V1_pseudomolecule (Jarvis et al., 2017) utilizando el alineador STAR v2.7a (Dobin et al., 2013) con parámetros estándar. Las alineaciones ordenadas por coordenadas (BAM) se utilizaron para producir una tabla de conteo utilizando el Programa featureCounts del paquete Subread v1.6.4 (Liao et al., 2013) con parámetros estándar.
El análisis de expresión génica diferencial se realizó con el paquete estadístico R usando la librería bioconductor edgeR v3.26.5 (Robinson et al., 2010; McCarthy et al., 2012). Se realizó un filtrado inicial de los genes de recuento bajo (logCPM <= 1 en menos de 3 bibliotecas) antes de la normalización del recuento con la media recortada de los valores M (TMM). La estimación de dispersión, el ajuste del modelo binomial negativo y la prueba de expresión diferencial se realizaron utilizando el método de cuasi likelihood (QLF). Se seleccionaron resultados significativos utilizando un umbral de FDR <= 1e-2 y LFC > = 1. Se produjeron mapas de calor (Heat maps) de genes expresados diferencialmente con el programa Complex Heamap v2.0.0 (Gu et al., 2016).
Análisis funcional
Los genes seleccionados fueron anotados funcionalmente con la herramienta en línea InterProScan v5.0 (InterPro, 2019).
Resultados y discusión
Selección de plantas e índice de contenido de clorofila
En la Tabla 2 se muestran las variedades de Chenopodium quinoa Willd clasificadas como tolerantes y susceptibles a la sequía. Las variedades evaluadas presentaron una respuesta fisiológica diferente a la sequía, principalmente durante el proceso posterior a la floración; cq01, fue la variedad más susceptible, disminuye su contenido de clorofila al sexto día de estrés por sequía y recupera su nivel de clorofila 14 días después de recibir nutrientes y agua. Ello se debe a la capacidad de la quinua en recuperar rápidamente su nivel fotosintético anterior y su área foliar después de un período de sequía (Jacobsen, 2009). Mientras que, cq05 y cq37 muestran mejor respuesta a la exposición a la sequía, Según Issa et al. (2019) la tolerancia de la quinua a la sequía sea el resultado de su capacidad para mantener el estado de salud celular. Similar resultado fue reportado por INIA (2013) para la variedad cq05; sin embargo, Apaza et al. (2013) reportaron moderada tolerancia a la sequía acq05 y tolerante a la sequía a cq37. Mientras que, Al-Naggar et al. (2017) y Iqbal et al. (2018) reportaron los genotipos CICA-17 y, 2-Want e IESP como tolerantes a la sequía respectivamente.
Tabla 2 Variedades tolerantes y susceptibles a la sequia
| Tolerante | Susceptible | ||
| cq03 | Ilpa INIA | cq01 | Red head |
| cq04 | INIA 415- Pasankalla | cq02 | INIA 431-Altiplano |
| cq05 | Salcedo INIA | cq07 | Blanca de Junín |
| cq06 | INIA 420 -Negra Collana | cq08 | Huancayo |
| cq12 | Compuesto B | cq09 | INIA 433 - Santa Ana /AIQ/FAO |
| cq23 | Compuesto tolerante a sequía | cq10 | Hualhuas |
| cq27 | INIA 433 - Santa Ana /AIQ/FAO | cq11 | Compuesto A |
| Cq29 | Salcedo INIA | cq13 | Chucuito |
| Cq30 | Ilpa INIA | cq14 | Compuesto Tolerante a Sequía Rojo |
| Cq31 | INIA 415-Pasankalla | cq15 | Compuesto Tolerante a Sequía Amarillo |
| Cq32 | INIA 420-Negra Collana | cq16 | Compuesto Tolerante a Sequía Rosado |
| Cq33 | Blanca de Junín | cq17 | Compuesto Tolerante a Sequía Anaranjado |
| Cq34 | Hualhuas | cq18 | Compuesto Tolerante a Sequía Blanco grano grande |
| Cq35 | Huancayo | cq19 | Compuesto Tolerante a Sequía Blanco grano pequeño |
| Cq36 | Kankolla 1 | cq20 | Misiquina |
| Cq37 | Kankolla 2 | cq21 | Compuesto tolerante purpura |
| Cq38 | Kankolla 3 | cq22 | Compuesto tolerante |
| Cq40 | Real Boliviana amarillo | cq24 | Quillahuaman |
| cq25 | Amarillo marangani | ||
| cq26 | Amarillo Sacaca | ||
| cq28 | INIA431 Altiplano | ||
| cq39 | Kankolla 4 | ||
| cq41 | Real Boliviano rojo | ||
Tabla 3 Librerías de las tres variedades
| Librería | Total de lecturas (PE) | Trimmed | Mapedo | Mapeado (%) | Asignado | Asignado (%) |
| s1 | 21235460 | 15958645 | 13195695 | 82,69% | 22010142 | 83,40% |
| s2 | 26042624 | 17496768 | 15207256 | 86,91% | 25870676 | 85,06% |
| s3 | 21359320 | 23116067 | 15634594 | 67,64% | 22739986 | 72,72% |
| s4 | 22487349 | 19291454 | 17032021 | 88,29% | 28937511 | 84,95% |
| s5 | 18934513 | 19080219 | 15951328 | 83,60% | 25563908 | 80,13% |
| s6 | 20138341 | 19107699 | 15765109 | 82,51% | 26643179 | 84,50% |
| s7 | 17652281 | 16883653 | 13686491 | 81,06% | 22531919 | 82,31% |
| s8 | 20785782 | 21192538 | 18304907 | 86,37% | 30238746 | 82,60% |
| s9 | 25020732 | 17311826 | 8832200 | 51,02% | 10043666 | 56,86% |
| s10 | 24709355 | 21110813 | 17642316 | 83,57% | 29490980 | 83,58% |
| s11 | 22510468 | 21071159 | 18805370 | 89,25% | 30170053 | 80,22% |
| s11_b | 23463593 | 19528151 | 8691231 | 44,51% | 10393715 | 59,79% |
| s12 | 18440773 | 18869351 | 16250968 | 86,12% | 26759953 | 82,33% |
| s13 | 23263746 | 21047103 | 18311306 | 87,00% | 30081114 | 82,14% |
| s14 | 24915800 | 21931420 | 19467683 | 88,77% | 32451811 | 83,35% |
| s15 | 20059027 | 20337430 | 17879369 | 87,91% | 29700693 | 83,06% |
| s16 | 24172363 | 21864578 | 19560180 | 89,46% | 32772462 | 83,77% |
| s17 | 21520144 | 23053829 | 19743744 | 85,64% | 32880582 | 83,27% |
| s18 | 22306438 | 21186853 | 18632001 | 87,94% | 32290632 | 86,65% |
| s19 | 20974163 | 18248062 | 16266915 | 89,14% | 27218929 | 83,66% |
| s20 | 24269140 | 21458480 | 19084316 | 88,94% | 32580420 | 85,36% |
| s21 | 17847231 | 19303327 | 17384596 | 90,06% | 29927863 | 86,08% |
| s21_b | 21781546 | 19968566 | 16421097 | 82,23% | 27245013 | 82,96% |
| s22 | 19525463 | 23243950 | 20674874 | 88,95% | 36178582 | 87,49% |
| s23 | 23236139 | 18135367 | 15452969 | 85,21% | 26373832 | 85,34% |
| s24 | 20074005 | 22134633 | 19736612 | 89,17% | 34089813 | 86,36% |
| s25 | 14365584 | 19725364 | 17502019 | 88,73% | 30140595 | 86,11% |
| s26 | 20460588 | 19052930 | 16487966 | 86,54% | 27492948 | 83,37% |
| s27 | 20075552 | 18780641 | 15308324 | 81,51% | 25771809 | 84,18% |
| s28 | 22344813 | 2196843 | 1915365 | 87,19% | 2979298 | 77,77% |
| s29 | 22788852 | 15967918 | 12131351 | 75,97% | 18757880 | 77,31% |
| s30 | 25126159 | 20322328 | 18340595 | 90,25% | 31130610 | 84,87% |
| s31 | 21081252 | 18255301 | 15913717 | 87,17% | 26926513 | 84,60% |
| s32 | 21249826 | 12071401 | 8701460 | 72,08% | 13835950 | 79,50% |
| s33 | 20623528 | 17598152 | 15397344 | 87,49% | 26155554 | 84,94% |
| s34 | 18807678 | 17427587 | 15242037 | 87,46% | 25953891 | 85.,14% |
| s35 | 21664660 | 21624342 | 18328768 | 84,76% | 31425594 | 85,73% |
| s36 | 22350282 | 24566008 | 21931894 | 89,28% | 37919484 | 86,45% |
Tecnología de secuenciación de ARN (RNA-seq)
En total se produjeron y secuenciaron 12 librerías (Tabla 3) por variedad (seis en prefloración, tres controles posfloración y tres sequías posfloración). En promedio, se produjeron 21 millones de lecturas emparejadas por librería (42 millones de lecturas) y alrededor del 10% de las lecturas se eliminaron después del recorte de calidad. De los 19 millones de lecturas emparejadas por biblioteca restante, el 83,5% se asignó al genoma de referencia del C. quinoa y el 82,1% de ellas se asignaron a una característica anotada en el genoma de referencia de las variedades en estudio.
De los ~ 44 mil genes que se encuentran en el genoma de C. quinoa (Zou et al., 2017), se mantuvieron aproximadamente 24 mil después del filtrado de genes de bajo conteo.
El número de genes expresados diferencialmente se resume en la Tabla 4. Las comparaciones de los cambios comunes en PRE-CPOS y PRE-DPOS se muestran en la Tabla 5.
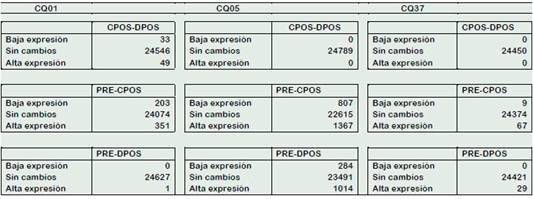
En cq01 se encontraron 554 genes expresados diferencialmente (DEG) (351 regulados al alza y 203 de baja regulación) en la comparación PRE - CPOS, mientras que solo se encontró un gen (regulado al alza) en la comparación PRE - DPOS.
Tabla 5 Comparación de los genes en los cambios comunes en PRE-CPOS y PRE-DPOS
B: Baja expresión
A: Alta expresión
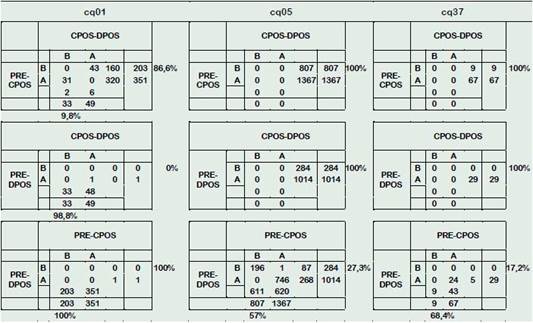
En la comparación CPOS - DPOS, se encontraron 82 DEG (33 regulados al alza en DPOS y 49 regulados a la baja). La comparación de los DEG de PRE-CPOS y PRE-DPOS mostró que en ambas situaciones los genes afectados son completamente diferentes y todos los cambios que ocurren naturalmente bajo riego regular después de la floración se neutralizan cuando se exponen a la sequía.
En cq05 se encontró un total de 2 174 DEGs entre CPOS y PRE (1367 regulados al alza y 807 de baja regulación) mientras que 1298 DEGs se encontraron entre DPOS y PRE (1014 regulados al alza y 284 de baja regulación). No se encontraron diferencias entre DPOS y CPOS. Similar a cq01, la comparación entre los DEG en los grupos PRE-CPOS y PRE-DPOS mostró que el 57% de los DEG en PRE-CPOS no se expresan diferencialmente en PRE-DPOS, lo que sugiere que la mayoría de los cambios naturales que se producen en la hoja después de la floración se neutralizan por exposición a la sequía. El 43% restante mantuvo un patrón de expresión similar, a excepción del gen AUR62002318 que se reguló rápidamente bajo condiciones de riego regular y se redujo bajo sequía. Este gen es un análogo de gen resistente a la enfermedad que contiene un dominio de unión a ADN B3, 2 regiones ricas en leucina y un dominio NB-ARC. Este dominio regula la muerte celular programada y es capaz de enlazar nucleótidos de adenosina o guanina (Giménez et al., 2006). Finalmente, el 73% de los DEG bajo tratamiento de sequía también se encontraron en condiciones de riego normales. Los resultados obtenidos indican que 355 genes son parte de la respuesta real a la sequía en cq05, mientras que los 943 genes restantes también son parte del desarrollo natural de la planta. Este resultado está en contraste con cq01, donde ninguno de los cambios que ocurrieron naturalmente se mantuvo en la respuesta a la sequía.
En cq37 se encontró un total de 76 DEG (nueve de baja regulación y 67 regulados al alza) en el análisis PRE-CPOS; mientras que, se encontraron 29 genes regulados al alza en el análisis PRE-DPOS. No se encontraron DEG entre los grupos DPOS y CPOS, similar resultado se obtuvo en cq05. Lo que sugiere que ambos grupos tienen patrones de expresión similar. En cq05, la gran mayoría de los cambios de expresión (68%) bajo riego normal se neutralizaron cuando se expusieron a la sequía. En cq37, 24 de los 29 (83%) genes regulados al alza en la comparación PRE-DPOS también se encontraron regulados al alza bajo riego regular. Estos resultados sugieren que la mayoría de los cambios causados por la floración bajo riego regular no ocurren cuando se exponen a la sequía. Además, solo cinco genes (AUR62037809, AUR62000271, AUR62037807, AUR62042825 y AUR62009791) tuvieron un cambio en su patrón de expresión como consecuencia de la exposición a la sequía. Tres de los cinco genes eran tiolasas con actividad de transferencia de acilo prevista, mientras que los dos restantes eran una proteína de unión a fosfatidiletanolamina y una oxidorreductasa dependiente de NADP.
En todas las variedades analizadas se observó un patrón similar, donde los cambios transcripcionales normales que siguen a la floración bajo riego regular se ven interrumpidos casi por completo por la exposición a la sequía. En cq01 este cambio fue completo y ninguno de los cambios de expresión que ocurrieron naturalmente se encontró bajo privación de agua. Mientras que, en cq05 y cq37, entre el 32% y el 43% de los cambios naturales no se vieron afectados por el estrés hídrico. Además, en las variedades tolerantes, entre el 73% a 83% de los cambios observados en los grupos expuestos a la sequía también se observaron en los grupos de control, lo que sugiere que estos genes no respondieron a la sequía.
Las variedades estudiadas presentan perfil similar de evolución de ICC para el control y tratamiento, se esperaba que sus patrones de expresión también reflejen este comportamiento. Sin embargo, mientras cq01 (susceptible) bajo estrés por sequía tuvo pocos cambios transcripcionales en comparación con el control (estado vegetativo), cq05 como cq37 (tolerante) permitieron que una fracción (~ 37%) de los cambios transcripcionales permaneciera en su lugar y la mayor parte de su respuesta a la sequía (~ 78%) fue idéntica al control. Este patrón de expresión convergente como respuesta al estrés por sequía es una característica común de C. quinoa tolerante a la sequía (Claeys e Inze, 2013). El hecho de que la mayoría de los genes que no responden a la sequía en plantas tolerantes también se expresaron en la planta susceptible, sugiere que estos genes están controlados por un factor de expresión diferente en ambos grupos. Es posible que las regiones promotoras de estos genes hayan mutado y, por lo tanto, no responden a los mismos estímulos que en las plantas susceptibles. También, es posible que el factor de transcripción que controla estos genes se haya desactivado y, por lo tanto, no pueda producir estos cambios.
Finalmente, son 26 genes los que se comportaron de manera similar en las variedades tolerantes a la sequía (cq05 y cq37), 17 de los cuales se encontraron regulados positivamente después de la floración bajo riego regular y estrés por sequía; tres fueron regulados negativamente en las plantas de control, pero no en los expuestos a la sequía y seis en las plantas de control, pero no en los expuestos a la sequía. No se encontraron genes en común entre aquellos que cambiaron el patrón como consecuencia de la exposición a la sequía sola. Solo tres de los 26 genes se encontraron regulados al alza en las plantas de control de cq01. Sin embargo, los 26 genes se encuentran con niveles significativos de expresión en las tres variedades (logCPM promedio = 5,03).
Conclusiones
De las 41 variedades utilizadas en el presente estudio se concluye que, 18 variedades son tolerantes a la sequía (Ilpa INIA, INIA 415- Pasankalla, Salcedo INIA, INIA 420 -Negra Collana, Compuesto B, Compuesto tolerante a sequía, INIA 433 - Santa Ana /AIQ/FAO, Salcedo INIA, Ilpa INIA, INIA 415-Pasankalla, INIA 420-Negra Collana, Blanca de Junín, Hualhuas, Huancayo, Kankolla 1, Kankolla 2, Kankolla 3 y Real Boliviana amarillo) y 23 variedades son susceptibles a la sequía (Red head, INIA 431-Altiplano, Blanca de Junín, Huancayo, INIA 433 - Santa Ana /AIQ/FAO, Hualhuas, Compuesto A, Chucuito, Compuesto Tolerante a Sequía Rojo, Compuesto Tolerante a Sequía Amarillo, Compuesto Tolerante a Sequía Rosado, Compuesto Tolerante a Sequía Anaranjado, Compuesto Tolerante a Sequía Blanco grano grande, Compuesto Tolerante a Sequía Blanco grano pequeño, Misiquina, Compuesto tolerante purpura, Compuesto tolerante, Quillahuaman, Amarillo marangani, Amarillo Sacaca, INIA431 Altiplano, Kankolla 4 y Real Boliviano rojo). Siendo la variedad Red head la más susceptible a la sequía; mientras que, las variedades más tolerantes son Salcedo INIA y Kankolla 2.
Las tres variedades de quinua (Red head, Salcedo INIA y Kankolla 2) presentan 26 genes que se comportaron de manera similar en las variedades tolerantes a la sequía; tres de ellos ante la exposición a la sequía son regulados al alza y cinco genes (AUR62037809, AUR62000271, AUR62037807, AUR62042825 y AUR62009791) tienen un cambio en su patrón de expresión como consecuencia de la exposición a la sequía. Este estudio ha generado información que podrá ser utilizada en futuras investigaciones sobre genes de resistencia a sequía en la quinua. Sin embargo, se necesitan datos de secuenciación adicionales para explorar mutaciones no codificantes alrededor de estos genes, así como datos adicionales de plantas más tolerantes y susceptibles para determinar qué tan extendido es realmente este patrón de expresión convergente. Pocos genes son comunes en la respuesta tolerante a la sequía.