Introducción
La superficie de la tierra se encuentra cubierta por bosques tropicales (7%), albergando dos tercios de la diversidad floral del mundo (Estoque et al., 2019), muchos de ellos son proveedores de bienes maderables y no maderables, los cuales brindan servicios ecológicos (captura de carbono, regulación hídrica y biodiversidad). La actividad humana ha reducido las áreas de bosques, transformándolas en zonas para uso agrícola y urbano, incrementando la explotación de especies forestales y fragmentando las áreas silvestres (Ledig, 1992), modificando la estructura demográfica forestal, el hábitat e impactando en la diversidad inter e intraespecífica de las especies; esta se debe al aprovechamiento indiscriminado de las especies forestales lo que ha generado deforestación en varias regiones del planeta (Bala et al., 2007; Barber et al., 2014; Busch y Amarjargal, 2020; Estoque et al., 2019; Guariguata et al., 2008; Kanae et al., 2001; Nogherotto et al., 2013; Rasolofoson et al., 2015; Spracklen y Garcia-Carreras, 2015; Zou et al., 1995), reduciendo la diversidad de las especies por la explotación directa de las poblaciones localmente adaptadas.
De acuerdo al primer catastro mundial de árboles del 2017, en el planeta se ha reportado 60065 especies de las cuales 9600 se encuentran en la categoría amenazada, resultado de la deforestación y la sobre explotación de estas, por lo que en los últimos años ha empezado a tener importancia la conservación para el aprovechamiento sostenible de los bosques (Newton et al., 1999), el cual permite obtener beneficios económicos, ambientales y socio económico de los bosques como una estrategia de mitigación al cambio climático (Canadell y Raupach, 2008). Al respecto la diversidad genética en las especies forestales permite a corto plazo la adaptación de las especies al cambio ambiental y a largo plazo favorece a la adaptación de las comunidades a las diferentes disrupciones ambientales. A la fecha, se tiene reportes acerca de la variación intra-especifica de especies forestales como Cedrela odorata (Meliaceae) (Gillies et al., 1997), Gliricidia sepium y G. maculata (Chalmers et al., 1992), Quercus petrae (Bacilieri et al., 1994), Bertholletia excelsa (Baldoni et al., 2020), Populus deltoides (Chen et al., 2020), Dipteryx sp. (Garcia-Davila et al., 2020), Swietenia macrophylla (Gillies et al., 1999), Pinus leucodermis (Bucci et al., 1997) Pinus oocarpa (Díaz-Vargas, 2001).
La familia Leguminoseae alberga alrededor de 600 géneros y 13000 especies cosmopolitas. Cedrelinga cateniformis (Ducke) “Tornillo” se distribuye en la región tropical de Sudamérica (Ecuador, Perú, Colombia y Brasil), desde los 120 hasta 800 m.s.n.m.; en áreas con una precipitación anual de 2500 a 3800 mm y temperatura promedio de 23 a 38 °C; es considerada como una especie de sucesión secundaria, de amplia distribución ecológica (húmedo tropical, subtropical y seco tropical), con buen desarrollo en suelos con bajos niveles de fertilidad (Aróstegui y Díaz, 1992). A nivel de la madera, esta se encuentra catalogada con una densidad básica media (INIA, 2010), de fácil trabajabilidad, con buen acabado por lo que es utilizado en carpintería (Gonçalez y Gonçalves, 2001) y es considerada como un sustituto de las especies tropicales sobreexplotadas (Haag et al., 2020)
Esta especie ha sido priorizada para estudios de domesticación (Leakey y Simons, 1997), así también en la incorporación de sistemas agroforestales (con café, cítricos, banano y cacao) lo que ha permitido reducir los bajos niveles económicos de los productores forestales en Junín (IIAP, 2009). Debido a su rápido desarrollo juvenil ha permitido su masificación en plantaciones en Ucayali y Loreto (Baluarte y Alvarez, 2015; IIAP, 2009).
FAO (2018) señala que el Tornillo es la principal especie utilizadas como madera aserrada, con una participación del 9,2% en el mercado nacional, el 90% procede del bosque tropical, principalmente de regiones como Loreto, Madre de Dios y Ucayali. Los trabajos de reforestación en esta especie son limitados, por lo que identificar la variabilidad intra-específica en Tornillo permitirá implementar una adecuada estrategia para la reforestación con una amplia variabilidad genética a fin de evitar su erosión genética.
El desarrollo de los marcadores moleculares, a la fecha ha permitido realizar estudios en filogenia y diversidad en especies forestales. Los RAPDs (Random Amplified Polymorphic DNA) son de bajo costo, de fácil manejo, requiere bajas concentraciones de ADN y genera perfiles genéticos en corto tiempo. Son marcadores de amplia utilización en especies forestales (Bucci et al., 1997; Chalmers et al., 1992; Gillies et al., 1997; Nesbitt et al., 1997; Newton et al., 1999; Tijerino et al., 2016; Wang et al., 2011), los cuales han permitido realizar estudios de diversidad genética. La región oriental del Perú concentra una diversidad de especies forestales con potencial de aprovechamiento sostenible. En Tornillo, a la fecha no se tiene ningún tipo de marcador que permita realizar estudios de diversidad genética. El presente trabajo tiene como objetivo identificar la estructura poblacional de Tornillo en la región oriental del Perú.
Materiales y métodos
Material biológico
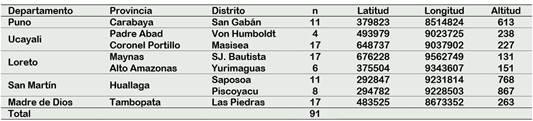
Los materiales de Tornillo fueron colectados en cinco departamentos de la región oriental del Perú (Loreto, Madre de Dios, Puno, San Martín y Ucayali) (Figura 1). Se registró la información dasométrica de los individuos seleccionados: altura total y comercial (Ht, Hc respectivamente), diámetro a la altura de pecho (DAP), rectitud de fuste y bifurcación, así mismo los individuos fueron georreferenciados (Tabla 1). Las hojas fueron colectadas en sobres de papel debidamente rotuladas y almacenadas en un recipiente hermético con silicagel, posteriormente las muestras fueron transportadas a la sede central del Instituto Nacional de Innovación Agraria (INIA) para la extracción de los ácidos nucleicos.
Extracción de ADN
La extracción de ADN se realizó de acuerdo con Cruz et al. (2019) el cual se basa en la metodología de Doyle y Doyle (1987) con modificaciones para la especie. Brevemente, las hojas fueron trituradas con nitrógeno líquido hasta la obtención de un polvo fino. 100 mg del polvo obtenido fue mezclado con buffer de extracción CTAB 2X (100 mM Tris-HCl, 1,4 M NaCl, 20 mM EDTA, 2% CTAB, 1% PVP) y 0,2% β-mercaptoetanol hasta la obtener una mezcla homogénea. Las muestras fueron incubadas a 65 °C por 60 minutos, posteriormente se adicionó cloroformo: alcohol isoamílico (24:1), el sobrenadante fue colectado y se adicionó buffer CTAB 10X (0,7 M NaCl, 20 mM EDTA, 100 mM Tris-HCl y 10% CTAB) y cloroformo: alcohol isoamílico (24:1). El sobrenadante obtenido fue precipitado con isopropanol frio, mezclándose suavemente para luego llevarlo a 20 °C por 1 hora. El pellet fue lavado sucesivamente con etanol (70 y 90 %) y resuspendido en agua libre de nucleasas, posteriormente se adicionó RNasa A (10 mg ml-1) incubándose a 37 °C por 3 horas. La integridad y calidad de los ADNs fueron evaluadas a través de una electroforesis en agarosa (1%) en buffer TBE (Tris-HCl 0,04 M; ácido EDTA 1 mM y ácido bórico 0,04 M; pH 8) a 90 V por 1 hora previa tinción con Gelred (Biotium®). La concentración y pureza del ADN fue realizada en un nanofotómetro NP80 (Implen®) donde se evaluó las relaciones A260/280 y A260/230.
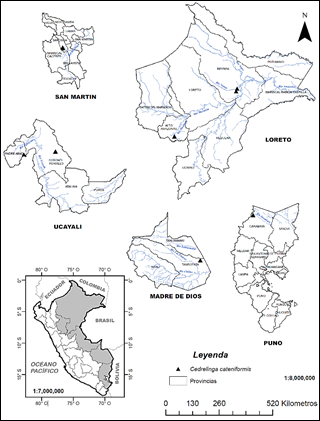
Figura 1 Zona de colecta de muestras de Tornillo realizada en cinco departamentos de la región oriental del Perú.
Caracterización genética
Se realizó una prueba para la identificación de los primers más polimórficos según lo propuesto por Goyal et al. (2014). El PCR se desarrolló en un volumen de 10 µl, se utilizó el kit Kapa HiFi Hotstart ReadyMix, primers (1 µM), ADN (2 ng) y agua libre de nucleasas. La amplifi cación se desarrolló en un termociclador Simplyone (Applied Biosystems™) siendo las condiciones: 94 °C por 4 min, 40 ciclos a 94 °C por 1 min, 37 °C por 45 s, 72 °C por 2 min y una extensión de 10 min a 72 °C.
Los fragmentos amplificados fueron separados a través de una electroforesis en agarosa (1 %) a 90 v en buffer TBE, la tinción se realizó con Gelred, el patrón de bandas fue visualizado en un transiluminador UV y las imágenes fueron almacenada en un fotodocumentador.
Análisis de datos
Los primers RAPDs más polimórficos generaron un perfil de patrones de bandas por individuo que fueron registrados en una matriz básica de datos como presencia y ausencia (1 y 0, respectivamente). El análisis descriptivo de los primers fue realizado a través de Infogen (Balzarini y Di Rienzo, 2004; 2016) evaluando, número de muestras duplicadas, proporción de loci polimórfico (PMF), contenido de informa ción polimórfica (PIC) (según Botstein et al., 1980), % de amplificación (AMP) y la probabilidad que dos individuos puedan compartir el mismo alelo al azar (PDICMA). A nivel poblacional se evaluó con GenAlex 6.5 (Peakall y Smouse 2016, 2012), número de alelos diferentes (Na), alelos efectivos (Ne), índice de Shannon (I), diversidad genética o Heterocigocidad esperada (He) y neutra de la población (uHe), el porcentaje de loci polimórfico, la distancia e identidad genética de Nei y la variación genética entre las poblaciones (PhiPT) a través de Análisis Molecular de Varianza (AMOVA), a la vez se realizó un análisis de correlación espacial entre la distancias genéticas y geográfica mediante de la prueba de Mantel. El análisis de disimilaridad se realizó mediante el coeficiente de Jaccard y el agrupamiento mediante el algoritmo de Neighbor Joining, así mismo se desarrolló el Análisis de Coordenadas Principales (PcoA) mediante el programa DARwin 6.5 (Perrier y Jacquemoud, 2006). La relación entre los grupos genéticos por localidades fue analizada a través del Árbol de Recorrido Mínimo (ARM) mediante Infostat (Di Rienzo et al., 2015).
Resultados y discusión
Diversidad genética
De los 44 primers evaluados se identificaron a los 5 más polimórficos (OPA02, OPA04, OPA12, OPA18 y OPF05) con patrones de bandas en el rango de 200 a 3000 pb. De los 96 marcadores identificados, OPA04 y OPA18 presentaron un mayor número de bandas polimórficas (20), seguido de OPA02 Y OPA12 (19) y OPF05 (18).
Los primers con el mayor contenido de amplificación (AMP) se registró para OPA04, OPA18, OPF05, OPA02 y OPA12 (53,57; 52,47; 48,78; 43,03 y 40,95 %, respectivamente). El promedio de las bandas polimórficas (47,86%) es superior a lo reportado por Hamrick y Godt (1990) para especies dicotiledóneas (44,8%). Los primers con mayor PIC fueron OPA18, OPA04, OPA12 y OPF05 (0,31; 0,30; 0,29 y 0,28 res pectivamente) estos marcadores superan la mitad del PIC siendo los más informativos y de mayor utilidad para la caracterización genética (Balzarini et al., 2010), mientras que OPA02 fue de 0,24. La probabilidad que dos individuos compartan el mismo alelo al azar (PDICMA) ha permitido identificar materiales duplicados en caña de azúcar (Balzarini et al., 2010). OPA04, OPA12, OPA18 y OPF05 permiten discriminar duplicados dentro de las muestras analizadas, no reportando duplicados para el presente estudio (Tabla 2). El Número de alelos diferentes (Na) oscila entre 1,698 a 1,896 en los departamentos de Puno (1,698); Ucayali (1,896); Loreto (1,813) y San Martin; en Madre de Dios fue menor (1,271), similar comportamiento con el número de alelos efectivos (Ne) (1,449; 1,507; 1,496; 1,496 y 1,297, respectivamente).
Tabla 2 Evaluación básica de los primers de RAPDs en tornillo
| Primer | BP | PMF95 | PIC | AMP | PDICMA |
| OPA02 | 19 | 0,95 | 0,24 | 43,03 | 1,70E-08 |
| OPA04 | 20 | 0,90 | 0,30 | 53,57 | 1,50E-11 |
| OPA12 | 19 | 0,95 | 0,29 | 40,95 | 2,90E-11 |
| OPA18 | 20 | 095 | 0,31 | 52,47 | 2,00E-12 |
| OPF05 | 18 | 1 | 0,28 | 48,78 | 2,60E-10 |
| Total | 96 | 47,86 | 3,90E-51 |
Bandas polimórficas (BP), la proporción de loci polimórficos (PMF95), Índice de Contenido Polimórfico (PIC), Porcentaje de Amplificación (AMP), Probabilidad de dos individuos compartan el mismo alelo al azar (PDICMA).
El índice de Shannon (I) provee una información robusta del intercambio genético dentro de una población, en un sentido más amplio a los valores de dispersión que el Fst (Rossetto et al., 2008; Sherwin et al., 2006). I oscila entre 0,402 a 0,447 para los departamentos de Puno (0,402); Ucayali (0,447); Loreto (0,433) y San Martín (0,422), mientas que Madre de Dios presenta un menor valor (0,262). La heterocigosidad (He) se encuentran en el rango de 0,265 - 0,289 en los departamentos Ucayali (0,296); Loreto (0,289); San Martin (0,278) y Puno (0,265); mientras que Madre de Dios presenta menor valor en comparación a los otros departamentos (0,174). Se realizó un análisis de correlación de Pearson entre los índices de Shannon y Nei encontrándose una alta correlación (r2 = 1, p-value < 0,0001), la cual ha sido reportada en espe cies forestales como Cedrela odorata (Tijerino et al., 2016). De la misma forma el porcentaje de loci polimórfico alto (81,25% - 91,67%) para los departamentos de Puno, Ucayali Loreto y San Martin, mientras que en Madre de Dios el porcentaje de polimorfismo es reducido (Tabla 3).
Estructura genética
El análisis espacial a través de la prueba de mantel señala una correlación significativa entre los patrones genéticos y geográficos (rxy = 0,311; p-value < 0,001). El agrupamiento realizado a través del índice de Jaccard permite la identificación de tres grupos poblacionales (Loreto, Madre de Dios y Ucayali), dentro del grupo de Ucayali se observa una sub-agrupación la cual corresponde a los departamentos de San Martín, Ucayali. En el caso de Puno, se encuentra relacionado con Ucayali (Figura 2a), la cual fue corroborado mediante el Análisis de Coordenadas Principales (Figura 2b) identificándose cuatro grupos genéticos (Loreto, San Martin, Madre de Dios, Ucayali- Puno). El análisis de conglomerados (Figura 2c) permite evaluar el agrupamiento de los cinco departamentos, Loreto y Ucayali se encuentra relacionados, así mismo Puno está relacionada con Ucayali, mientras de Madre de Dios y San Martin se encuentran distanciadas a los dos primeros departamentos. El análisis de Correspondencia Canónica (Figura 2c) corrobora la cercanía de las poblaciones de Loreto y Ucayali, a la vez se visualiza relación entre Ucayali- Puno y Loreto- Madre de Dios y de San Martin. A la vez, las poblaciones del sur (Ucayali y Puno) se agrupan en un mismo cuadrante; mientras que las poblaciones del norte (Loreto y San Martin) se encuentra opuesto a las poblaciones del sur, mientras que la de Madre de Dios tiene una cercanía a la población de Loreto.
El árbol de recorrido mínimo (ARM) es un algoritmo de encadenamiento de objetos que permite una mejor interpretación de las ordenaciones en espacios de dimensión reducida, los puntos se conectan con segmentos de líneas rectas de manera que se encuentren unidos (Arroyo et al., 2005; Balzarini et al., 2010). Los dos componentes del ARM a nivel de departamento representa el 68 % de distribución poblacional, donde las poblaciones de Ucayali y Loreto son poblaciones diferenciadas y cercanamente emparentadas, mientras que la población de Puno tiene un origen en Ucayali, de la misma manera San Martin se encuentra más distanciado. A la vez que la población de Loreto interacciona con las de Madre de Dios. A nivel de localidad, las evaluaciones en la localidad de San Juan Bautista (Loreto) ha interaccionado con las localidades Ucayali (Masisea e Irazola), así también con las Piedras (Madre de Dios), siendo Masisea la localidad que ha interaccionado con las poblaciones de San Martin (Saposoa y Piscoyacu) y Puno (San Gabán) y Madre de Dios (Las Piedras) (Figura 3).
Tabla 3 Resumen de los índices de genética en las poblaciones de tornillo a nivel de departamento
| Departamento | N | Na | Ne | I | He | uHe | % Loci polimórfico |
| Puno | 11 | 1,698 | 1,449 | 0,402 | 0,265 | 0,278 | 81,25 |
| Ucayali | 21 | 1,896 | 1,507 | 0,447 | 0,296 | 0,304 | 91,67 |
| Loreto | 23 | 1,813 | 1,496 | 0,433 | 0,289 | 0,295 | 86,46 |
| San Martín | 19 | 1,813 | 1,469 | 0,422 | 0,278 | 0,285 | 88,54 |
| Madre de Dios | 17 | 1,271 | 1,297 | 0,262 | 0,174 | 0,179 | 53,13 |
Número individuos por localidad (N), Número de alelos diferentes (Na), Número de alelos efectivos (Ne), Índice de Shannon (I), Heterocigosidad esperada (He), Heterocigosidad esperada neutra (uHe).
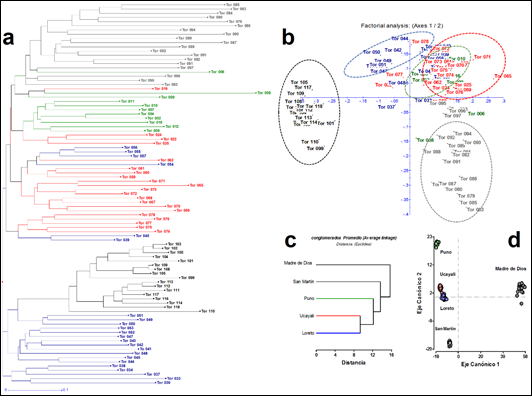
Figura 2 Estructura poblacional de las poblaciones de Tornillo a partir marcadores RAPDs. a) Agrupamiento mediante el índice de Jaccard a través del método de Neihgbor joining. b) Análisis de Coordenadas Principales (PCoA). c) Análisis de conglomerados de las poblaciones de Tornillo (distancia Euclidiana). d) Análisis de correspondencia de las poblaciones de Tornillo.
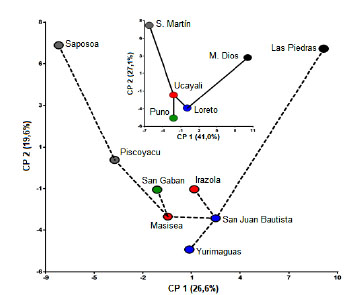
Figura 3 Interacción genética entre los departamentos y poblaciones de Tornillo por Árbol de Recorrido Mínimo
Tabla 5 Matriz de diferenciación poblacional pareada entre la distancia genética de Nei (diagonal abajo) vs. Variación genética poblacional (PhiPT) (diagonal arriba) en tornillo
| Departamento | Puno | Ucayali | Loreto | San Martin | Madre de Dios |
| Puno | 0,093 | 0,160 | 0,179 | 0,438 | |
| Ucayali | 0,059 | 0,082 | 0,171 | 0,397 | |
| Loreto | 0,083 | 0,048 | 0,226 | 0,368 | |
| San Martin | 0,106 | 0,080 | 0,112 | 0,419 | |
| Madre de Dios | 0,244 | 0,226 | 0,200 | 0,240 |
La variación genética en las poblaciones de Tornillo se presentan principalmente una alta variación de los individuos dentro de los departamentos (74%) y una baja se variación entre los departamentos (26%), el comportamiento de la alta variación dentro de departamentos es semejante a lo reportado en Cedrela odorata (Tijerino et al., 2016) los cuales presentan una alta significancia (PhiPT = 0,256; p-value < 0,0001), estos resultados permitiría a definir una estrategia para la conservación in situ y ex-situ como señala Andrianoelina et al. (2006).
La comparación pareada de la diferenciación poblacional fue calculada a partir de los índices de Nei y PhiP (Tabla 5). El apareamiento de PhiP se encontró en el rango de 0,082 - 0,438; la población de Madre de Dios tiene menor diferenciación con los otros departamentos, siendo Ucayali-Puno y Ucayali-Loreto los que presentan menor diferenciación de PhiP (0,093 y 0,082 respectivamente). Esta relación es similar con el índice de Nei (0,048 y 0,059). La última comparación es consistente geográficamente. En ambas comparaciones Puno es genéticamente similar a Ucayali, de la misma manera Loreto y Ucayali. Las poblaciones forestales con diferentes orígenes geográficos presentan altos niveles de variación (Hamrick et al., 1992), las cuales se encuentran influenciadas por el sistema de reproducción, el flujo de genes, la dispersión de semillas y la selección natural (Wang et al., 2011), estos conceptos contribuyen al desarrollo de estrategias para la identificación de unidades de conservación (Newton et al., 1999), las especies forestales con amplio rango geográfico, representa un alto nivel de diversidad genética en comparación a especies endémicas y especies autopolinizadoras (Casiva et al., 2002).
Conclusiones
Los 5 marcadores RAPDs seleccionados (OPA02, OPA04, OPA12, OPA18 y OPF05) ha generado 96 bandas polimórficas que ha permitido evaluar la diversidad genética de Tornillo, El PIC de los marcadores empleados presentan variaciones lo que permite el análisis de diversidad, los individuos colectados dentro de los departamentos presentan una variabilidad en la heterocigosidad (He), siendo Madre de Dios la región con menor valor, estos valores guardan una relación directa con el índice Shannon. Asimismo, las poblaciones estudiadas presentan una correlación entre las variables geográficas y genéticas. Loreto y Ucayali son las poblaciones estrechamente relacionada; los individuos de San juan Bautista (Loreto) presenta una relación directa con los Masisea e Irazola (Ucayali) y las Piedras (Madre de Dios); así mismo Masisea se relaciona con las poblaciones de Piscoyacu y Saposoa (San Martin).
El desarrollo de un programa de conservación es importante en el conocimiento de la distribución de la variación genética de una especie para el aprovechamiento racional a de la diversidad de grupos genéticos de la especie, por lo que es necesario la toma de decisiones basadas en patrones genéticos especialmente si la acción antropogénica tiene un papel predominante sobre los hábitats de las especies. En Tornillo, a través investigación en estructura y diversidad permite determinar el estado actual de la especie en el Perú, siendo este el primer reporte del estado poblacional que permitirá implementar una línea base del estado situacional de la especie como se viene desarrollando en especies nativas forestales de importancia económica en el Perú.