1. Introducción
Chenopodium quinoa Willd., comúnmente conocida como quinua, es un cultivo que presenta importancia alimenticia por su alto valor nutricional, ya que contiene ácidos grasos insaturados (linoleico y oleico) y alto contenido de aminoácidos (Peiretti, Gai, & Tassone, 2013), siendo considerada como una de las principales fuentes de proteínas para la alimentación humana. Es el único vegetal que contiene todos los aminoácidos esenciales y es rica en vitaminas, ácidos grasos, minerales y fibra (Avalos, 2018; FAO, 2018), atributos que pueden ser utilizados para combatir la seguridad alimentaria (Bazile et al., 2016). Actualmente la quinua ha tomado mayor importancia debido a que es una especie nativa con alto valor agronómico, nutritivo y de exportación (Rosales & Mercado, 2020), debido principalmente a la gran diversidad genética existente, que se expresa en los diferentes colores de panojas, de hojas, de grano, altura de planta, contenido de saponina, tipo de panoja y calidad de grano (Brenes, 2001). El Perú, presenta una amplia biodiversidad la que debe ser cuidada y aprovechada racionalmente, ya que actualmente se observa la pérdida de diversidad biológica aumenta día tras día (Chirinos-Arias, Jiménez, & Vilca-Machaca, 2015). Además de su importancia económica, social y cultural de la especie, se conserva y maneja con fines de investigación agronómica y obtención de nuevas variedades. Siendo de mucha importancia el conocimiento de la diversidad genética y el uso apropiado de la quinua, se hace necesaria la caracterización de su germoplasma. Las caracterizaciones y evaluaciones de las colecciones de quinua actualmente se realizan mediante metodologías complementarias basadas en marcadores morfológicos, bioquímicos y moleculares, con el fin de conocer la composición genética de este recurso. Las técnicas moleculares han sido relevantes para el estudio de genomas vegetales, ofreciendo niveles de confiabilidad más elevados si se les compara con las que sólo se limitan a estudiar características morfológicas, anatómicas o ecofisiológicas (Saldaña & Salazar, 2007).
Existen diversas técnicas de marcadores moleculares, las más empleadas en estudios de poblaciones son los AFLPs (Amplified Fragment Lenght Polymorphisms), RAPD (Random Amplified Polymorphic DNA), ISSR (Inter - simple sequence repeats) y microsatélites (SSR). Cada uno tiene sus ventajas y desventajas y su aplicación dependerá de la disponibilidad de recursos. Los marcadores AFLP son am pliamente empleados ya que permite una detección rápida de los polimorfismos del genoma, generan un gran nú mero de bandas, son altamente reproducibles y no se re quiere información previa del genoma para su aplicación (Vos et al., 1995).
Los marcadores AFLP se utilizaron para evaluar las relacio nes genéticas entre 12 especies de Trapa del río Yangtze Cuenca, reportando que con 264 bandas de AFLP todos los individuos estaban claramente divididos en grupos de acuerdo con sus posiciones taxonómicas y orígenes geo gráficos, ello demuestra la importancia de las pruebas moleculares para los estudios de filogenia (Xiang-Rong, Wu-Chao, Li, Wei, & Yuan-Yuan, 2021).
En la investigación sobre diversidad genética, estructura poblacional y evolución de F. cirrhosa y especies estrechamente relacionadas utilizando marcadores de AFLP, donde descubrieron que las especies ampliamente distribuidas mostraron una mayor diversidad que las especies endémicas, además las poblaciones de F. cirrhosa de las montañas de Hengduan estuvieron mejor protegidas, debido a su di versidad genética, en comparación con poblaciones marginales, por lo que los marcadores AFLP son una herramienta valiosa para la conservación y uso sostenible de plantas (Wu, Duan, Chen, & Zhang, 2020).
Existen estudios en los que se utiliza los marcadores AFLP como en la investigación sobre la diversidad genética y diferenciación de especies de plantas medicinales Amapola persa (Papaver bracteatumL.) usando marcadores AFLP, en el que se determinó que la variación genética entre las poblaciones deP. bracteatumfue mayor dentro de las poblaciones y demostró que el marcador AFLP es una herramienta eficiente y confiable para el análisis de la variación genética de la especie (Hadipour, Kazemitabar, Yaghini, & Dayani, 2020).
En la investigación sobre la caracterización fitoquímica y genética de cinco genotipos de quinua (Chenopodium quinoaWilld.) introducidos en Egipto, se utilizaron 4 marcadores RAPD y 7 ISSR para el análisis genético y en el análisis UPGMA de los marcadores polimórficos RAPD e ISSR, su conjunto de datos combinado y rasgos fitoquímicos se utilizaron para evaluar las relaciones genéticas entre genotipos concluyendo que los genotipos de quinua mostraron una variación razonable en los rasgos fitoquímicos estudiados y los que serán de base para mejorar la calidad de la semilla de quinua (Saad-Allah & Youssef, 2018).
La utilización de marcadores moleculares es fundamental para estudios genéticos y en la identificación de nuevas líneas polacas de Chenopodium quinoa (Willd.) mediante la confirmación de la estabilidad genética con SCoT y marcadores RAPD, el cual reveló una alta estabilidad genética de las líneas derivadas de 'Faro' y 'Titicaca', mientras que se detectó variaciones en plantas de quinua que representan cultivares originales presentando un patrón de bandas diferente al predominante que se visualizó en tres plantas de ‘Titicaca’ (distancias genéticas de 7,5% a 55,9%) y en una sola planta de 'Faro' (distancia genética 61,2% según indica la técnica SCoT) (Lema-Rumińska, Miler, & Gęsiński, 2018).
Se ha estudiado la diversidad genética y estructura poblacional de la quinua (Chenopodium quinoa Willd.) usando marcadores iPBS-retrotransposones de 17 genotipos de quinuas cultivadas en Turquía, en la que concluyeron que el estudio proporciona información útil para el manejo del germoplasma de quinua y contribuye a programas de mejoramiento genético. También presentaron el sistema de marcadores iPBS como una herramienta adecuada para la identificación y análisis de diversidad genética de genotipos de quinua (Hossein-Pour, Haliloglu, Ozkan, & Tan, 2019).
Se ha desarrollado la investigación sobre huellas dactilares de ADN y análisis de diversidad genética del germoplasma mundial de quinua, procedente de 8 países a través del sistema de marcadores iPBS-retrotransposón, determinando que mediante el análisis de varianza molecular (AMOVA) reveló que la mayoría de las variaciones (69%) en el germoplasma mundial de quinua son debido a las diferencias dentro de las poblaciones. Los hallazgos de este estudio se pueden utilizar para una comprensión más profunda de la relación genética y en la determinación de estrategias apropiadas de mejoramiento y conservación de la quinua (Barut, Nadeem, Karaköy, & Baloch, 2020).
En un estudio más amplio sobre la comprensión de la base química y el mecanismo genético del grano negro de la quinua, se emplearon análisis de metaboloma y transcriptoma para revelar las diferencias químicas y ge néticas entre cultivares de quinua blanca y negra, encon trándose un total de 157 metabolitos diferentes expresa dos en los cultivares de quinua blanca y negra. Este estu dio sugiere que la acumulación de proantocianinas pro duce el rasgo de grano negro en la quinua y la activación de los factores de transcripción desencadenan la vía de biosíntesis de proantocianidina (Wang et al., 2020).
Considerando la importancia de realizar estudios de variabilidad genética en quinua en la región de Ayacucho y evitar la potencial pérdida de su diversidad genética, la investigación se planteó como objetivo caracterizar a nivel molecular la quinua cultivada en esta región. La información generada permitirá fortalecer la conservación y utilización de los recursos genéticos del cultivo de quinua, cuya importancia va creciendo progresivamente no solo para los pobladores andinos sino para el mundo.
2. Materiales y métodos
Material Biológico
Se utilizaron 29 accesiones de quinua, del Banco de Ger moplasma de INIA, Ayacucho. Las muestras fueron ho jas de 3 semanas por accesión y se tomaron en la Esta ción Experimental Agraria Canaán - Ayacucho (INIA). Una vez colectadas las hojas, fueron empaquetadas en papel húmedo debidamente rotuladas, para luego ser transportadas al laboratorio de Biotecnología de la Universidad Nacional de San Cristóbal de Huamanga para realizar la extracción de ADN y posterior análisis de AFLPs en quinua.
Extracción de ADN. El procedimiento de extracción de ADN se realizó usando el método CTAB establecido por Doyle & Doyle (1990) modificado por el CIP (2017). La calidad y la cantidad de ADN fue verificada por el método de electroforesis en geles de agarosa al 1% (p/v), para la carga de las muestras al gel de agarosa se empleó 1μL de ADN genómico el cual fue diluido con 9 μL del colorante de carga SALB 1X. Como tampón de corrida se utilizó TBE 1X. Para el cálculo de la concentración de ADN de las muestras se comparó con la intensidad de la banda del estándar del ADN Lambda (λ) digerida con la endonucleasa de restricción Pst I.
Técnica de AFLP
Digestión del ADN genómico. El ADN extraído de las accesiones de quinua se llevó a las concentraciones de 50, 100, 200, 300 y 400 ng/µl para la estandarización de la técnica. Se preparó una mezcla de reacción para un volumen total de 20 μl conteniendo H2O libre de nucleasas (9,5 μl), MseI NEB 5U (0,5μl), EcoRI NEB 10 U (1 μl), buffer 10X (2,0 μl), BSA 1mg/ml (2,0 μl) y ADN (5 μl). Se verificó la digestión del ADN genómico, realizando electroforesis de 5 μl de muestra de ADN digerido en geles de agarosa 1%, en el cual se visualizó un barrido de ADN muy tenue en el transiluminador UV.
Ligación de adaptadores. Se prepararon mezclas de reacción de ligación con los siguientes componentes: ADN genómico digerido (21 μl), H2O libre de nucleasas (0,2 μl), adaptador EcoRI 10 μM (1 μl), adaptador MseI 100 μM (1 μl), Buffer de ligasa NEB (2,5 μl) y T4 ADN ligasa (0,3 μl). Una vez obtenido la mezcla de ligación se agregó a 21 μl de ADN genómico digerido. Luego esta mezcla de reacción de digestión/ligación (DL) se mezcló, centrifugó y se incubó a 16 ºC por toda la noche. Finalizada la incubación, se realizó una dilución de 1:3 de la mezcla digestión/ligación (DL) con solución tampón TE (10Mm, Tris-HCL (pH 8.0), 0,1 nM EDTA).
Preamplificación (+1/+1). Para la preamplificación (+1/+1) se utilizaron strips y se les adicionaron los siguientes componentes: 14,05 μl de H2O libre de nucleasas, 2,5 μl de Buffer 10X PCR, 1,25 μl de mezcla de dNTPs 5 mM, 1 μl de iniciador EcoRI+1, 1 μl de iniciador MseI+1, 0,2 μl de Taq polimerasa y 5 μl la dilución 1:3 de ADN digerido/ligado.
El perfil de temperatura utilizado para la PCR +1/+1 fue: un ciclo de 72 ºC por 2 minutos, un ciclo de 94 °C por 4 min, veintidós ciclos de 94 °C por 30 segundos, 56 ºC por 1 minuto, 72 °C por 1 minuto, y un ciclo de 72 ºC por 5 minutos, luego fue conservado a 4 ºC. Finalizada la preamplificación, se realizaron diluciones (1:4, 1:6, 1:10 y 1:20 para la estandarización de la técnica) del PCR +1/+1 con H2O libre de nucleasas.
Tabla 1 Combinaciones de iniciadores AFLPs utilizados en la selección de mejores combinaciones
| Combinación de iniciadores | EcoRI | MseI | ||
| E32M48* | E32 | AAC-3 | M48 | CAC-3 |
| E33M60* | E33 | AAG-3 | M60 | CTC-3 |
| E32M61 | E32 | AAC-3 | M61 | CTG-3 |
| E38M60 | E38 | ACT-3 | M60 | CTC-3 |
| E38M47* | E38 | ACT-3 | M47 | CAA-3 |
| E45M61* | E45 | ATG-3 | M61 | CTG-3 |
| E38M32 | E38 | ACT-3 | M32 | AAC-3 |
| E38M61* | E38 | ACT-3 | M61 | CTG-3 |
| E45M32 | E45 | ATG-3 | M32 | AAC-3 |
| E32M60 | E32 | AAC-3 | M60 | CTC-3 |
| E33M48* | E33 | AAG-3 | M48 | CAC-3 |
| E33M61* | E33 | AAG-3 | M61 | CTG-3 |
* Combinaciones de iniciadores seleccionados.
Amplificación Selectiva (+3/+3). Se adicionaron los siguientes componentes a la mezcla de reacción: 0,9 μl de H2O libre de nucleasas, 1,1 μl de Buffer 10X PCR, 0,6 μl de mezcla de dNTPs 5 mM, 2 μl de iniciador EcoRI +3, 0,3 μl de iniciador MseI+3, 0,1 μl de Taq polimerasa y 5 μl de ADN preamplificado. El programa utilizado para la amplificación selectiva fue el siguiente: un ciclo de 94 °C por 4 minutos, 94 ºC por 20 segundos, 65 ºC por 30 segundos y 72 ºC por 2 minutos. Diez ciclos de 94 ºC por 20 segundos, 65 ºC por 30 segundos y 72 ºC por 2 minutos, veinte ciclos de 94 ºC por 20 segundos, 56 ºC por 30 segundos, 72 ºC por 2 minutos, por último, un ciclo de 60 ºC por 30 minutos y a 4 ºC para su almacenamiento. Para el estudio se realizó un tamizado de 12 combinaciones de iniciadores AFLP en ocho accesiones de quinua escogidos al azar, fueron elegidas las siete mejores combinaciones (Tabla 1).
Análisis estadístico. Se determinó el índice de contenido polimórfico y se estimó los índices de variabilidad genética y proporción de loci polimórfico. A partir de las lecturas de las bandas que se obtuvieron con cada combinación de iniciadores se construyó una matriz de presencia y ausencia de bandas de ADN (1 y 0), con la que se realizó el análisis de similaridad calculando el coeficiente de Jaccard (Jaccard, 1908) y mediante la matriz de similaridad entre las accesiones, se realizó el análisis de agrupamiento por el método UPGMA (Unweighted Pair Group Method Using Arithmetic Average) con ello se generaron dendrogramas y coordenadas principales. Luego se determinó el coeficiente de correlación cofenético, utilizando la matriz del coeficiente de similitud, con su correspondiente matriz cofenético. El programa que se utilizó NTSYSpc 2.2.
3. Resultados y discusión
Extracción de ADN, calidad y concentración. En la extracción de ADN de quinua las concentraciones obtenidas se encontraron en un rango de 100 - 400 ng/μl. La calidad del ADN extraído mediante el método de Doyle & Doyle (1990) modificado, fue óptima para todas las muestras, obteniéndose un ADN genómico no degradado ya que se observaron bandas bien definidas sin la presencia de un barrido que indica degradación ADN.
En la Figura 1 se observa el gel donde se muestra las bandas de ADN genómico sin la presencia de ARN y las bandas no presentan degradación por parte de las ADNasas.
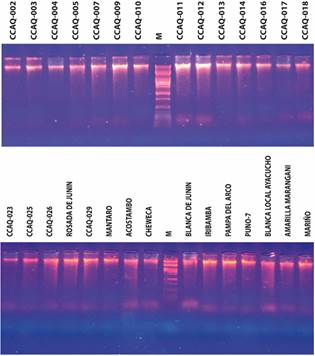
Figura 1 Gel de agarosa al 1% mostrando la calidad de ADN genómico de 29 accesiones de “quinua”. Marcador λ (M) digerido con Pst I. TBE 1X, 90V.
Estandarización de la técnica AFLP. La optimización de la técnica se inició con la modificación de la cantidad de ADN con que se inició la digestión enzimática ya que se contaba con diferentes concentraciones (100-400 ng/μl). Se realizaron varios ensayos (50, 100, 200, 300 y 400 ng/μl), determinándose que a concentraciones entre 100 y 200 ng/μl de ADN digerido fueron adecuadas para continuar de manera eficiente la técnica de AFLP. Además, en la investigación se ha encontrado mejores resultados con diluciones menores a las recomendadas por la técnica, siendo la dilución 1:4 los que mostraron perfiles de amplificados mucho más claros e intensos en comparación a los perfiles obtenidos cuando las diluciones fueron mayores. Este resultado es coherente con las modificaciones que se realizó a la técnica de AFLP en la investigación de Cóndor (2013). Al respecto Zhu, Gale, Quarrie, Jackson, & Bryan (1998) menciona que el ajuste en la dilución del producto de la preamplificación puede realizarse de 1 a 50 veces, dependiendo de la intensidad de las bandas, lo que permite obtener mejor resolución en los geles.

Figura 2 Patrón de bandas AFLPs obtenidas de siete combinaciones de iniciadores en “quinua”. Poliacrilamida 6% TBE 1X, 400v.
Selección de combinaciones de iniciadores AFLP y marcadores AFLP detectados. En el análisis de AFLP se eligieron las siete mejores combinaciones (E32M48, E33M60, E38M47, E45M61, E38M61, E33M48, E33M61), por presentar buena resolución de bandas, buen nivel de polimorfismo y buena reproducibilidad (Figura 2). En la Figura 3 se muestran bandas polimórficas y monomórficas producidas con los marcadores AFLP, estos perfiles de amplificación AFLP corresponden a las combinaciones E32M48 y E38M61. En la Tabla 2 se observa que las siete combinaciones de iniciadores AFLP generaron un total de 220 bandas de ADN. El número de bandas totales varió de 23 (con la combinación de los iniciadores E33M48) a 41 (con la combinación de los iniciadores E32M48), con un promedio de 31,4 bandas por combinación.
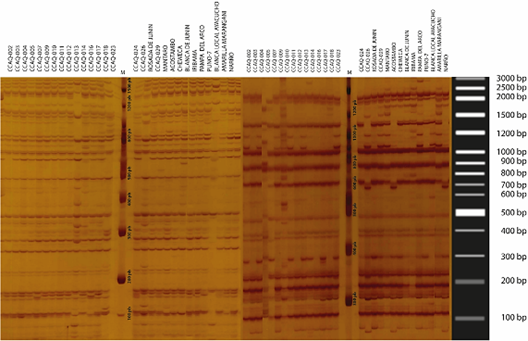
Figura 3 Perfil de amplificación AFLP de “quinua” (a) Combinación de iniciadores E32M48 y (b) Combinación de iniciadores E38M61. “M” representa el marcador de tamaño molecular. Poliacrilamida 6%, TBE1X, 400V.
Alrededor del 91,4% de las bandas (201 bandas) mos traron polimorfismo, el número de bandas polimórficas por combinación varió de 19 bandas (combinación E33M48) a 37 bandas de ADN (combinación E32M48). La detección de bandas polimórficas se realizó en un rango de 100 - 2000 pb (Figura 3). En la tabla 3 se observa que la combinación de iniciadores que detectó un mayor porcentaje de bandas polimórficas fue la combinación E32M48 con 18,41%, y la combinación de iniciadores que detectó menos porcentaje de bandas polimórficas fue la combinación E33M48 con 9,45%. Re sultados similares fueron reportados por Nolasco, Cruz, Santa Cruz y Gutiérrez (2013), quienes realizaron el tamizado de cinco combinaciones de iniciadores para el análisis de AFLP, observando que la combinación de iniciadores con mayor número de bandas polimórficas fue la E32M48, confirmando el obtenido con la misma combinación en esta investigación.
Tabla 2 Bandas monomórficos y polimórficos obtenidos con cada combinación de iniciadores en el análisis de AFLP en quinua
| Combinación de Iniciadores | Total de bandas | Bandas mono-mórficos | Bandas poli-mórficos |
| E45M61 | 29 | 3 | 26 |
| E38M61 | 31 | 1 | 30 |
| E33M48 | 23 | 4 | 19 |
| E33M61 | 31 | 1 | 30 |
| E33M60 | 34 | 4 | 30 |
| E38M47 | 31 | 2 | 29 |
| E32M48 | 41 | 4 | 37 |
| Total | 220 | 19 | 201 |
| Promedio | 31,4 | 2,7 | 28,7 |
| % | 100 | 8,6 | 91,4 |
Tabla 3 Porcentaje de bandas polimórficos obtenidos con cada combinación de iniciadores en el análisis de AFLPs de “quinua”
| Combinación de iniciadores | Bandas polimórficos | Bandas polimórficos (%) |
| E45M61 | 26 | 12,94 |
| E38M61 | 30 | 14,93 |
| E33M48 | 19 | 9,45 |
| E33M61 | 30 | 14,93 |
| E33M60 | 30 | 14,93 |
| E38M47 | 29 | 14,43 |
| E32M48 | 37 | 18,41 |
| Total | 201 | 100 |
Índice de iniciadores AFLP. En la Tabla 4 se muestran el promedio de PIC de cada combinación de iniciadores AFLP, observándose que el promedio de PIC va desde 0,13 con la combinación E32M48, hasta 0,23 con la combinación E33M61.
Tabla 4 Índice del contenido de información polimórfica (PIC) y promedio del índice de contenido polimórfico empleados en el análisis de marcadores AFLPs en Chenopodium quinoa Willd. “quinua”
| Combinación de Iniciadores AFLP | Índice del contenido de información polimórfica (PIC) | Promedio del Índice de Contenido Polimórfico |
| E45M61 | 4,36 | 0,17 |
| E38M61 | 5,00 | 0,17 |
| E33M48 | 3,2 | 0,17 |
| E33M61 | 6,99 | 0,23 |
| E33M60 | 4,17 | 0,14 |
| E38M47 | 5,49 | 0,19 |
| E32M48 | 4,78 | 0,13 |
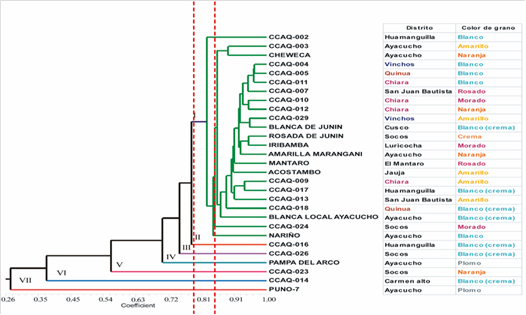
Figura 4 Dendrograma de Similaridad de Jaccard obtenido del análisis de 29 accesiones de Chenopodium quinoa Willd. “quinua”, provenientes del Banco de Germoplasma del INIA.
Al analizar todas las accesiones de quinua, podemos observar que la combinación de iniciadores E33M61 mostró ser el más discriminante porque su índice de iniciador fue 6,99 y su promedio de PIC fue 0,23, seguido por la combinación de iniciadores E38M47 con un índice de 5,49 y un promedio de PIC de 0,19. Indicando la utilidad de estos dos iniciadores como los marcadores más informativos para determinar polimorfismo y diversidad. Mientras que la combinación menos informativa fue la combinación E33M48 con un índice de iniciador de 3,2 y un promedio de PIC de 0,17. Al respecto Christensen et al. (2007), señaló que un buen marcador informativo con un alto índice de polimorfismo permite el estudio de la diversidad genética de las diferentes cultivares existentes.
Estimación de índices de variabilidad genética y proporción de loci polimórfico (Ps). Se determinó la variación genética promedio “Hi” (o índice de heterocigocidad), el índice de información de Shannon “I” y la proporción de loci polimórficos con un criterio del 95%. En este estudio se evaluaron 201 loci polimórficos que fueron obtenidos en el análisis de AFLP de 29 accesiones de quinua procedentes del Banco de Germoplasma del INIA; se encontró que la variación genética promedio fue de 0,306 ± 0,011, con un índice de información de Shannon de 0,463 ± 0,015. La proporción de loci polimórfico de las combinaciones elegidas fue de 91,120% (Tabla 5).
Recordando que el máximo valor que puede alcanzar la heterocigocidad en un estudio con marcadores dominantes es de 0,5 y el índice de Shannon 0,69 podemos indicar estos resultados mostraron un alto nivel de polimorfismo genético en las accesiones de quinua por las combinaciones de iniciadores AFLP evaluadas. Los valores de diversidad encontrados para las accesiones analizadas son altos y reafirman el alto grado de diversidad genética mantenido en el Banco de Germoplasma del INIA-Ayacucho. Los resultados concuerdan con Costa (2011), quienes caracterizaron molecularmente 51 poblaciones de Chenopodium quinoa Willd. “quinua” del Banco de Germoplasma de Argentina encontrando una alta diversidad genética (Hi = 0,355).
Tabla 5 Variación genética e índice de Shannon empleados en el análisis de marcadores AFLPs en Chenopodium quinoa Willd. “quinua”
| Combinación de iniciadores | Variación genética (He) | Índice de Shannon (I) | Proporción de loci polimórfico Ps (%) |
| E45M61 | 0,266 | 0,407 | 89,655 |
| E38M61 | 0,253 | 0,393 | 96,774 |
| E33M48 | 0,278 | 0,427 | 82,609 |
| E33M61 | 0,371 | 0,549 | 96,774 |
| E33M60 | 0,302 | 0,456 | 88,235 |
| E38M47 | 0,355 | 0,529 | 93,548 |
| E32M48 | 0,303 | 0,464 | 90,244 |
| Promedio | 0,306 | 0,463 | 91,120 |
Análisis de similaridad y coeficiente de correlación cofenético (r). El coeficiente de Correlación Cofenética (r) de la matriz de similaridad y la matriz cofenética del dendrograma de 29 accesiones de quinua, fue realizado mediante el coeficiente de Jaccard, siendo ésta 0,9935 el cual indicó que existía una buena estructura jerárquica entre la matriz de similaridad y su correspondiente dendrograma, ya que cuando el coeficiente de correlación cofenética es mayor a 0,9 éste puede ser interpretado como un buen ajuste del análisis de agrupamiento. Resultados similares fueron obtenidos por 14, en su investigación de análisis de diversidad genética en tomate utilizando AFLPs, reportó que encontró alta correlación de las matrices de similitud, con un índice de similitud de Jaccard de r = 0,997. En la matriz de similaridad se evidencian relaciones muy cercanas entre las accesiones CCAQ-004 y CAAQ-005, CCAQ-004 y CCAQ-011, CCAQ-005 y CCAQ-011 que mostraron índices de similaridad de Jaccard de 0,97 cada par; por otra parte, se encuentran bajos valores de similaridad (0,13) al comparar las accesiones CCAQ-014 y PUNO-7 indicando que son las accesiones que difieren más genéticamente, dichas relaciones se observan en el agrupamiento mostrado en el dendrograma (Figura 4), en ella se puede observar que las accesiones más diferenciado está compuesto por las accesiones CCAQ-014 y PUNO-7 y los más relacionados son CCAQ-004 y CAAQ-005, CCAQ-004 y CCAQ-011, CCAQ-005 y CCAQ-011.
Análisis de agrupamiento. Si bien es cierto en las colec ciones se muestra alta variabilidad, las accesiones del Grupo I se encuentran más relacionadas, esta congruen cia puede ser favorecida a que dentro de este grupo se encuentran variedades importantes comercialmente como son Blanca de Junín y Amarilla Marangani, lo que hace que éstas accesiones tengan características agronómicas semejantes incrementando su relación genética, favoreciendo así su diseminación y cultivo en distintos distritos, entre ellas zonas importantes productivamente como Chiara, lo cual se evidencia en el dendrograma (Figura 4).
También se puede apreciar en el dendrograma una alta diferenciación genética entre seis accesiones pertenecientes a los grupos II, III, IV, V y VI, el cual podría deberse a la poca comercialización de éstas, ya que, al no ser materiales importantes comercialmente, no ha sufrido ciclos de selección por parte de los agricultores y ha mantenido su estructura genética. Además, podrían ser accesiones con distinto origen histórico, identidad y quizá con ausencia de mejoramiento adaptadas a los sistemas agrícolas locales por un tiempo indefinido como lo menciona Camacho, Maxted, Scholten, & Ford-Lloyd (2005), siendo accesiones que presentan alelos diferentes incrementando así la diversidad en las muestras estudiadas. Se puede observar que algunas accesiones se agruparon por su origen geográfico, por ejemplo, todas las accesiones que fueron colectadas en Quinua se encuentran en el grupo I, del mismo modo todas las accesiones colectadas en Vinchos, Chiara y San Juan Bautista se encuentran en este grupo (Figura 4). También se puede observar agru pamiento de acuerdo con el color de grano, por ejemplo, las accesiones que presentan color de grano amarillo se encuentran en el grupo I. Además, dos accesiones (Pampa del Arco y Puno 7) pertenecientes a los grupos IV y VII respectivamente, presentan en común semillas color plomo y se presentan a mayor distancia que el grupo I (Figura 4).
Para apreciar visualmente las distancias genéticas entre accesiones de quinua, se realizó un análisis de Coordenadas Principales (PCO), el cual se muestra en la Figura 5 con un diagrama en dos dimensiones. El análisis de PCO, método de ordenamiento que centra a los genotipos en una nube de puntos preservando las relaciones de similitud establecidas entre ellos, permitió obtener un gráfico en dos dimensiones que reflejó un agrupamiento similar al que mostró el dendrograma del análisis realizado previamente. El análisis de PCO, a diferencia del análisis de agrupamiento, otorga más libertad en la expresión gráfica de las relaciones genéticas establecidas en la matriz de similitud. Las relaciones entre los genotipos están determinadas por la posición en que se disponen en un espacio reducido (Bonámico et al., 2004). La coincidencia observada en los resultados logrados con ambas metodologías de análisis multivariado valida el agrupamiento obtenido para los 29 genotipos analizados e indica, tal como sugiere Johnson (2000), que dicho agrupamiento es el que existe naturalmente entre estos genotipos. El Banco de Germoplasma del INIA-Ayacucho mantiene 140 accesiones de las Chenopodium quinoa Willd. “quinua” y actualmente realiza colectas con el fin de ampliar la colección.
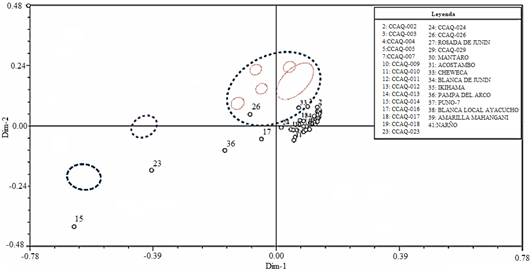
Figura 5 Diagrama en 2D de Coordenadas Principales de 29 accesiones de Chenopodium quinoa Willd. “quinua”.
Este tipo de estudios sobre los recursos fitogenéticos en general son importantes porque documentan la diversidad presente en la colección y que tan diferentes son unas accesiones de otras para evitar tener duplicados. Esta información es importante para la formación de colecciones núcleo que son un subgrupo de la colección original que alberga la mayor diversidad posible con el menor número de accesiones y de esta manera establecer prioridades sobre el gasto de recursos para mantener colecciones extensas.
4. Conclusiones
En la caracterización molecular de las accesiones de quinua del Banco de Germoplasma del INIA, no se detectó duplicados dentro de las accesiones evaluadas, siendo importante para el manejo eficiente de un Banco de Germoplasma al dar un alcance de la diversidad genética y en base al análisis molecular y a medida que se incremente el número de accesiones de la colección de Chenopodium quinoa Willd es necesario realizar estudios moleculares relacionados con estudios morfológicos de varios individuos por accesión con la finalidad de determinar mayor diversidad genética y es recomendable evaluar mayor número de combinaciones de iniciadores AFLP para incrementar el número de marcadores polimórficos y conseguir representatividad en los procesos de caracterización molecular.















