1. Introducción
Physalis peruviana es una solanácea nativa de los Andes Sudamericanos, conocida como “aguaymanto”, que en los últimos años se ha constituido en un cultivo económicamente importante para países como Colombia y Perú. Fue domesticado, cultivado y muy apreciado por los antiguos peruanos (Chasquibol & Yácono, 2015, Deza & Delgado, 2017) y es considerada como una fuente importante de compuestos nutricionales y principios bioactivos beneficiosos tanto para la salud humana como para la industria alimentaria (Obregón-La Rosa et al., 2021).
En general, no se reportan variedades definidas de la especie, pero usualmente es agrupada en ecotipos, teniendo en cuenta el color, forma y sabor del fruto, forma de la flor, hábito y altura de la planta, entre otros (Dostert et al., 2013). En Physalis peruviana se tiene determinado 3 ecotipos claramente diferenciados por sus caracteres morfológicos, los ecotipos Kenia, Sudáfrica y Colombia, los que llevan el nombre del lugar de procedencia, y son los más frecuentemente cultivados en el mundo; aunque existen referencias de más de 80 ecotipos descritos a nivel mundial (Dostert et al., 2013; Puente et al., 2011; Rodriguez et al., 2006).
En el Perú, Dostert et al. (2013) mencionan la existencia de 4 ecotipos provenientes de Cajamarca, referenciados en el Museo de Historia Natural, Perú. Cajamarca es el departamento con la mayor área cultivada de aguaymanto (Bonilla et al., 2019). Esto permite considerar a la región de Cajamarca como uno de los principales centros de diversidad genética de aguaymanto en nuestro país. Sin embargo, hay pocos estudios de identificación genética de la especie; entre estos, pueden mencionarse los estudios a nivel de proteínas seminales (Bonilla et al., 2019), o de recuento cromosómico (Escobar, 2016), así como la caracterización citogenética de 3 ecotipos de Cajamarca (Carbajal, 2018). Liberato et al. (2014) y Rodríguez (2006) reportan cariotipos con dotaciones cromosómicas de 2n = 48 y 2n = 24. Carbajal (2018) estudió poblaciones de Cajamarca, reportando una dotación cromosómica 2n = 48, adicionalmente evidenció presencia de aneuploidía somática en sus poblaciones analizadas observando células con 2n = 24, 46, 50, 52, 62, 80 y 82. Por otro lado, debido al nivel de ploidía esperado de 4X, no puede establecerse cariotipos a partir de grupos de homólogos de 4 cromosomas utilizando las técnicas de citogenética clásica, siendo muchas veces imposible la construcción de un cariotipo que caracterice a un ecotipo determinado (Rodríguez et al., 2006).
La técnica de Hibridización Fluorescente in Situ, FISH por sus siglas en inglés, constituye un método convencional muy utilizado para visualizar la distribución de elementos ADN dentro del genoma; permite realizar estudios relacionados con la estructura, mutación y evolución tanto de cromosomas individuales como de genomas completos (Jiang, 2019). Con aproximadamente 30 años de desarrollo, sus variantes técnicas y refinamientos la mantienen vigente como una herramienta muy útil en caracterización de especies o líneas de cultivo, entre otras, y continúa jugando un rol importante en investigaciones citogenéticas y genómicas (Garcia et al., 2017) La aplicación más común de FISH es el mapeo de sondas de ADNr en cromosomas, esto debido a que las secuencias son abundantes, repeti das y altamente conservadas (Setiawan et al., 2020; Jiang, 2019; Garcia et al., 2017).
Los genes de ADN ribosomal (ADNr) son los más utilizados como sondas en los estudios de citogenética molecular debido a que contribuyen con información sobre homología entre segmentos cromosómicos, así como por ser secuencias altamente conservadas, tal es el caso de los genes 5S y 45S los que se constituyen en excelentes marcadores moleculares para un amplio grupo de especies vegetales (Garcia et al., 2017; Fukushima et al., 2011; Muravenko et al., 2004; Jiang, 2019). La identificación en posición de estos genes en los cromosomas y el número de loci presentes, posibilita la caracterización de cromosomas y genomas, ayudando así a la caracterización a nivel citomolecular del germoplasma; esto a su vez, permite evaluar la variabilidad inter e intraespecífica, lo cual también servirá para un posterior análisis evolutivo a nivel cariotípico entre especies genéticamente relacionadas (Yagi et al., 2015).
En el caso de aguaymanto, no se reportan estudios de citogenética molecular, pero sí en otras especies pertenecientes a las Solanaceae (De Paula et al., 2015; Aguilera et al., 2016;Jiang, 2019). Teniendo en cuenta la carencia de estudios a nivel citomolecular en la especie, encontramos que resulta necesario llevar a cabo un análisis de variabilidad genética que permita identificar la presencia o ausencia de diferencias a nivel de citogenética molecular de aquellos ecotipos o poblaciones que ya han sido descritos en base a caracteres agronómicos y cromosómicos básicos.
El presente trabajo tuvo el objetivo de identificar el número de señales marcadas para el locus 5S ADNr en el conjunto de cromosomas de Physalis peruviana, aguaymanto, en las poblaciones de San Pablo y Celendín procedentes de la región de Cajamarca, Perú, utilizando la técnica de Hibridización Fluorescente In Situ, lo que podría servir como punto de partida para discernir si efectivamente se tratan de dos ecotipos diferentes como señalan sus productores refiriéndose como ecotipos Agroandino y Celendino respectivamente.
2. Materiales y métodos
Material Biológico
Se colectaron 10 frutos maduros por cada planta a partir de 10 plantas de aguaymanto, de poblaciones cultivadas en las Provincias de San Pablo (S°7° 5'43.9'', latitud; O°78°49'13.9'', longitud) y Celendín (S°6º52’35. 9’’, latitud; O°78º07'49'', longitud) del departamento de Cajamarca-Perú. Los frutos fueron colocados en envases de plástico transparente, con tapa y orificios en la misma que permi ten la circulación del aire, para su traslado al laboratorio de Genética de la Facultad de Ciencias Biológicas (UNMSM). Las muestras herborizadas de cada población fueron entregadas al Herbario del Museo de Historia Natural de la Universidad Nacional Mayor de San Marcos (MHN-UNMSM) para su identificación taxonómica (Códigos: 222-USM-2015, 223-USM-2015). Las semillas fueron retiradas de los frutos y almacenadas en tubos de polipropileno a 4 °C, para su posterior utilización (Carbajal, 2018).
Obtención de la sonda 5S rADN
Se realizó la extracción de ADN siguiendo el protocolo descrito por Doyle & Doyle (1990) con algunas modificaciones. Posteriormente se amplificó la región 5S ADNr a partir de los primers pr5S14 (5′-GGCGAGAGTAGTACTAGGATCCGTGAC-3′) y pr5S15 (5′- GCTTAACTTCGGAGTTCTGA TGGGA-3′) reportado por Volkov et al. (2001), siguiendo estas condiciones de PCR: Buffer de PCR 1X, MgCl 1.5 mM, dNTPs 0.2 µM, 0.2 µM de cada primer,1 U de Taq polimerasa y 30 ng de ADN genómico, en un volumen de reacción de 20 µl; llevado a un programa de 95 °C por 9 minutos seguidos de 40 ciclos de 95 °C por 30 segundos, 55 °C por 30 segundos y de 72 °C por 60 segundos, y una extensión final de 72 °C por 4 minutos. Después, el amplicón 5s rADN fue marcado con moléculas de biotina mediante la técnica molecular del Nick Translation, utilizándose el kit comercial DIG-Nick Translation Mix de ROCHE, siguiendo las instrucciones del fabricante.
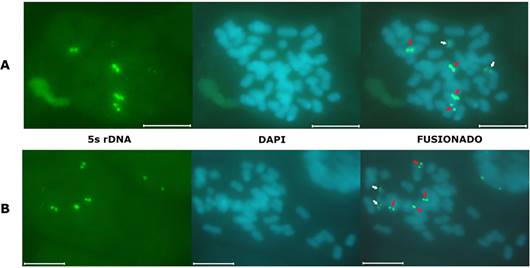
Figura 1 Metafase somática de P. peruviana, población Celendín (A) y población San Pablo (B) hibridadas con la sonda 5s ADNr, detectada con un conjugado de neutravidina - Oregon Green 488 (fluorescencia verde) y contrateñidas con DAPI. (las flechas rojas señalan las señales grandes y las flechas blancas señalan las más pequeñas, la barra blanca presenta una escala de 10 μm).
Preparación de cromosomas vegetales a partir de ápices radicales
Se realizó la germinación de las semillas siguiendo el protocolo establecido por Carbajal (2018) con algunas modificaciones: las semillas se sembraron sobre papel filtro humedecido con agua destilada en placas Petri a temperatura ambiente (22 - 28 ºC), manteniendo siempre la humedad del papel filtro. Las raíces de 1 a 2 cm de longitud fueron colectadas, entre las 10 y 12 de la mañana e inmediatamente sometidas a un pre-tratamiento con Colchicina al 0,025% por 1 h 20 minutos a temperatura ambiente (entre 22 - 25 ºC) y en oscuridad; seguido de una hipotonización en agua destilada por 60 minutos a 37 °C. Después, el tejido fue fijado en una solución etanol: ácido acético (3:1) durante 24 horas a 8 °C, hasta su posterior uso. Posteriormente para la preparación de placas metafásicas se tuvo en cuenta los protocolos reportados por Aliyeva-Schnorr et al. (2015) y Aguilera et al. (2016), siendo las raíces fijadas lavadas en agua destilada fría dos veces por 5 minutos cada vez, seguido de dos lavados en buffer citrato (0,01 M a pH 4,6) por 5 minutos cada vez. Luego, las raíces se transfirieron a una solución enzimática que contenía celulasa al 2% (de Aspergillus niger, Sigma) y pectinasa al 10% (de Aspergillus niger, Sigma), disueltas en 40% de glicerol en buffer citrato, durante 1 hora a 37 °C. Después, las raíces se enjuagaron dos veces por 10 minutos cada vez con buffer citrato y dos veces en etanol a 90° por cinco segundos, con agitación en cada enjuague. Seguidamente se colocó la punta de la raíz en una gota de ácido acético al 45% sobre una lámina, se cubrió con una laminilla y se realizó la técnica del “squash”. Las láminas elaboradas se observaron en un microscopio con contraste de fase, seleccionándose aquellas que presentaron metafases con cromosomas adecuadamente separados. Luego se levantó la laminilla con ayuda de hielo seco y un bisturí. Finalmente, las láminas se dejaron secar por 24 horas a temperatura ambiente y se almacenaron a 4 °C sumergidas en etanol absoluto hasta su uso.
Hibridización Fluorescente In Situ (FISH)
Para el FISH se siguió el protocolo reportado por Poggio et al. (2000), en cada uno de sus pasos.
Para poder evidenciar la hibridización de la sonda en los cromosomas se realizó la sobreposición de las fotos utilizando el programa GIMP 2.10 (www.gimp.org.es) que es un software de libre circulación.
3. Resultados y discusión
Identificación de la región 5S ADNr en los cromosomas mediante FISH
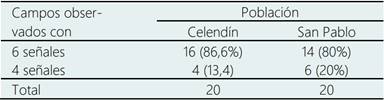
Los resultados de la localización del marcaje mediante FISH en las preparaciones cromosómicas de P. peruviana con la sonda ADNr utilizada, permitieron ubicar el locus 5S ADNr en el complemento cromosómico de las 2 poblaciones de aguaymanto analizadas. Se analizaron un total de 20 campos cromosómicos por población, detectándose 6 señales fluorescentes de color verde en el 86,6% de campos analizados para Celendín y de 80% para San Pablo, en tanto que se evidencia un porcentaje menor de 13,4% para Celendín y de 20% para San Pablo que muestran 4 señales. Las 6 señales se observan en 3 pares de cromosomas diferentes, para ambas poblaciones; 4 de ellas son fácilmente visibles por su tamaño e intensidad de brillo y 2 de ellas se ven más pequeñas y con un brillo de menor intensidad. (Figura 1, a y b; Tabla 1). En cuanto a la diferencia encontrada en tamaño e intensidad de brillo en los cromosomas hibridados, podríamos argumentar que un mayor tamaño e intensidad de la señal indicarían un mayor número de copias del gen 5S ADNr en esos loci, así como una menor intensidad y menor tamaño de la señal se debería a un menor número de copias en ellos. Esto ha sido observado en otras especies de la familia (Aguilera et al., 2016; Moscone et al., 2007; Neves et al., 2005), sin embargo, habría que ampliar las zonas de estudio en la especie para corroborarlo.
El locus 5S se mapea en posición intercalar, casi distal, en los brazos cromosómicos de ambas poblaciones estudiadas, aunque de momento no es posible precisar tamaño y morfología de los brazos. El locus 5S ADNr con sus secuencias repetidas en tándem suele ubicarse en cualquier parte del genoma, aunque usualmente lo hace cerca de las regiones teloméricas o centroméricas (Liu et al., 2010). Es así como en Capsicum (Solanaceae), el locus se encuentra en posición intercalar cerca al telómero del brazo corto del cromosoma que lo porta; por otro lado, en Passiflora se muestra en posición subterminal (Aguilera et al., 2016; De Melo et al., 2003) Estas evidencias encontradas en otras especies, tanto de la misma familia que aguaymanto o de otras familias, coinciden con nuestras observaciones.
De manera general, esta característica que muestran las señales de ambas poblaciones tanto en brillo como en tamaño sugiere un número de copias ribosómicas similar por locus para ambas poblaciones; esto también ha sido evidenciado en otras especies de la familia (Aguilera et al., 2016; Moscone et al., 2007; Neves et al., 2005).
Aguilera (2016) indica que en las Solanáceas suele encontrarse un locus 5S simple por genoma haploide para los taxa diploides y loci doble en taxa poliploides; sin embargo, en otros géneros, como Passiflora todos los diploides presentan 2 cromosomas marcados, los tetraploides presentan 4 cromosomas marcados y los hexaploides 6 cromosomas marcados, indicando así que el número de sitios guarda una clara relación con el nivel de ploidía (De Melo et al., 2003). En nuestro estudio, el número de señales identificadas en ambas poblaciones refuerza la idea de que estaríamos ante una especie poliploide (2n= 48; x=12) si bien es cierto que no podríamos precisar si se trata de una tetraploide o una hexaploide debido a que no se ha podido establecer aún un cariotipo con ordenamiento de homólogos de 4 o 6 cromosomas con las técnicas de citogenética clásica (Rodriguez et al., 2006; Carbajal, 2018) a causa del tamaño pequeño y la morfología similar de los cromosomas. Sin embargo, en nuestras observaciones habría que tener en cuenta que siendo el número básico x=12, para afirmar la presencia de hexaploidía deberíamos contar 72 cromosomas y no 48, que es el número que encontramos.
No hay reportes sobre estudios en identificación del locus 5S ADNr en aguaymanto por lo que este estudio sería el primero. En el estudio, no se observaron diferencias en cuanto a número, tamaño de las señales, e intensidad del brillo con la que se marca el locus, teniendo que es muy semejante en ambas poblaciones: 4 señales grandes y 2 más pequeñas en cada complemento; estos resultados refuerzan la idea de que las poblaciones de Celendín y San Pablo no corresponderían aún a ecotipos diferenciados, lo que va de acuerdo con lo mencionado por Bonilla et al. (2019) en el que se reporta que no existe diversidad genética entre las poblaciones de Celendín y San Pablo; los mismos autores señalan que es de conocimiento general que las poblaciones estructuradas presentan bajos niveles de diversidad genética y que este tipo de poblaciones es característico en poblaciones cultivadas, como es también en nuestro caso , dado que la colecta de las muestras trabajadas también se hicieron en campos cultivados con un programa de mejoramiento genético por selección masal (Bonilla et al., 2019).
4. Conclusiones
Se reporta por primera vez un estudio de identificación del locus 5S ADNr en los complementos cromosómicos de 2 poblaciones de aguaymanto procedentes del departamento de Cajamarca, encontrándose 6 zonas marcadas, 4 grandes y 2 pequeñas, en 3 pares de cromosomas para ambas poblaciones. Los resultados del presente estudio pueden ser utilizados para investigar el mapeamiento cromosómico de P. peruviana lo que, a su vez, contribuirá a la comprensión de la diversidad genética de esta especie en nuestro territorio y establecer relaciones de filogenia.