INTRODUCCIÓN
ANTECEDENTES
El COVID-19 es una pandemia según lo ha catalogado la OMS1, cuyas complicaciones sobre la salud aún no están claramente definidas, se desconoce de un tratamiento efectivo, siendo la única propuesta terapéutica el desarrollo de una vacuna útil en humanos2. Esta pandemia al 09 de abril del 2020 viene generando una mortalidad global de personas infectadas de 5,98%, en China 3.9%, España 10,1%, Francia 10,3%, Italia 12.7%, Reino Unido 12.1%, EE UU 3.6%, México 5.5%, Perú 2.6%, Chile 1%3. Por lo que, esta pandemia viene poniendo a prueba la capacidad de respuesta y de protección financiera de los sistemas de salud a nivel mundial, donde la mayoría de países vienen desarrollando variadas estrategias de contingencia, siendo la principal y algunos casos la única, la estrategia de medidas sociales de contención contra la transmisibilidad del virus SAR COV-21.
La certeza del diagnóstico varía según la muestra de estudio, el tipo de prueba y el momento de su aplicación4,5, así mismo, los parámetros de transmisibilidad varían en cada población; sin embargo, diferentes publicaciones científicas vienen dando a conocer estimaciones del comportamiento de la pandemia actual por el virus SARS COV-2 en poblaciones específicas, aplicando como base el modelamiento SIR y sus variaciones6,7,8. Si bien el modelamiento varía según complejidad en distintos escenarios de la población7,9,10, también existe la probabilidad de incrementar el sesgo y falta de precisión con cada parámetro adicional incluido al modelamiento6,7; en la pandemia actual se desconocen parámetros propios, y no se considera adecuado extrapolar valores de un estudio a otras poblaciones7,11,12.
El objetivo del estudio consistió en analizar la evolución de la pandemia Covid-19 estimando el efecto de las medidas de contención realizadas en una población como la nuestra.
MATERIAL Y METODOS
Estudio observacional aplicando el modelamiento matemático epidemiológico SIR. El estudio se realizó considerando 31237385 personas, según último censo realizado por el Instituto Nacional de Estadística (INEI), siendo el año 2017. Posterior al censo realizado, no fueron consideradas las defunciones ni los nacimientos ocurridos hasta el presente estudio.
Variables: casos susceptibles identificados como persona en riesgo de adquirir la infección; casos infectados persona con resultado positivo según pruebas moleculares de laboratorio para la infección del virus SARS COV-2; casos recuperados identificados como todo aquel que no puede transmitir la infección del virus SARS COV-2 (pacientes que sobrevivieron y pacientes fallecidos); tiempo de ocurrencia del pico máximo de infecciones definido como el periodo de días en el cual la población peruana llegara al mayor número de casos infectados; efecto de las medidas de contención medido según tasa efectiva de propagación de la infección (controlada al ser menor a 1) y según porcentaje de infecciones evitadas hasta el día pico de infección; medidas de contención identificadas como cierre de fronteras, aislamiento social obligatorio, inmovilización social obligatoria de 8 pm a 5 am y suspensión de viajes nacionales e internacionales.
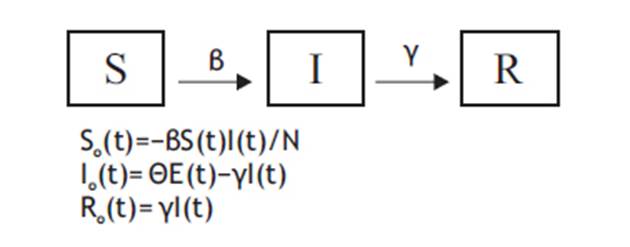
Los datos fueron obtenidos mediante un seguimiento diario de los reportes del Ministerio de Salud en su página oficial (https://covid19.minsa.gob.pe/sala_situacional.asp). Para el modelamiento se utilizó el modelo Susceptible-Infectado-Recuperado (SIR)9. El modelo comprende un sistema de ecuaciones diferenciales resueltas con el método Euleriano, las cuales corresponden a:
Donde: N: población total (S+I+R), S: casos susceptibles, I: casos infectados, R: casos recuperados, β: tasa de transmisión de la infección, γ: 1/periodo de infección.
Se aplicó el modelamiento matemático SIR sin medidas de contención (Modelo 1,2,3,4,5,6) y según propagación de la infección con medidas de contención (Modelos 7,8,9,10,11,12). El modelamiento se realizó en función del número oficial reportado de infecciones (Modelos 1, 3, 5, 7, 9, 11) y el doble del número de casos oficialmente reportados (Modelos 2, 4, 6, 8, 10,12). Según el reporte diario de casos infectados se calibro el índice de propagación promedio, estimando la tasa de propagación de la infección sin medidas de contención (Ro1) y con medidas de contención (Ro2) practicadas en población peruana. La tasa de transmisión sin medidas de contención (β1) se obtuvo mediante: β1=(Ro1)(γ)/S. La tasa de transmisión con medidas de contención (β2) se obtuvo mediante β2=(Ro2)(γ)/S. La tasa de recuperación se obtuvo para 11, 14 y 21 días de infección. Los cálculos estimados para no uso de medidas de contención fueron realizados con los datos reportados entre el 10 de marzo y el 18 de marzo 2020; mientras que los cálculos estimados con medidas de contención fueron realizados con los datos reportados entre el 24 de marzo y el 01 de abril 2020 (resultados bajo medidas de contención). El error de estimación en cada modelo con medidas de contención fue obtenido contrastando los resultados del modelamiento con la data oficialmente reportada hasta el 08 de abril del 2020.
Se aplicó la ecuación de transmisibilidad epidémica con medidas de contención7, para estimar respuesta de acciones gubernamentales: β(t) = βo(1-α)(1-D(t)/N)k ; donde βo es la tasa de infección sin medidas de contención, α es el resultado de las acciones gubernamentales, D(t) es la sensación publica de riesgo como consecuencia de los casos críticos y muertos conocidos, K mide la intensidad de reacción de los individuos; estudios indican un valor de K: 1117.3 y un valor D(t) del 5%.
Se realizó el análisis de datos siguiendo el modelo matemático SIR con ecuaciones diferenciales ordinarias definidas para simular el comportamiento epidemiológico del COVID-19 y ejecutadas en el lenguaje de programación RStudio. A partir de los casos infectados reportados diariamente y los casos prevalentes del día anterior se calibro el índice de propagación diario de la infección para la etapa sin medidas de contención y con medidas de contención. A partir del índice de propagación calibrado se estimó de forma proporcional la tasa de propagación de infección en función a los periodos de infección de 11, 14 y 21 días; así mismo, se estimó sus respectivos IC 95% mediante 5 mil replicas Bootstrap sin reemplazamiento. Los modelamientos realizados en la etapa de medidas de contención fueron comparados mediante el cálculo del error cuadrático medio para identificar el modelo con menor error de estimación. Se midió porcentualmente los casos infectados estimados de los modelamientos matemáticos para calcular la reducción de casos infectados según modelamiento matemático SIR.
RESULTADOS
Se desarrolló 6 modelos SIR sin considerar medidas de contención y 6 modelos considerando las medidas de contención practicadas en población peruana. Se obtuvo tasas de recuperación de 0.091, 0.071 y 0.048 para duración de la infección de 11, 14 y 21 días respectivamente. El modelo 12 presento una tendencia de casos infectados estimados con el menor error de estimación; dicho modelo presento una mayor tasa de propagación de la infección. El análisis de la transmisibilidad de la infección estimó un resultado de las acciones gubernamentales del 61% en la aplicación de las medidas de contención gubernamental. Para la calibración de la tasa de propagación se consideró el índice de propagación promedio de la infección, siendo de 0.376 sin contención, y de 0.146 para medidas con contención; a partir de los cuales se obtuvo el Número reproductivo básico respectivamente, según duración de la infección (Tabla 1).
Tabla 1 Variables y parámetros en el modelamiento SIR de la infección del SARS CoV-2 en población peruana.
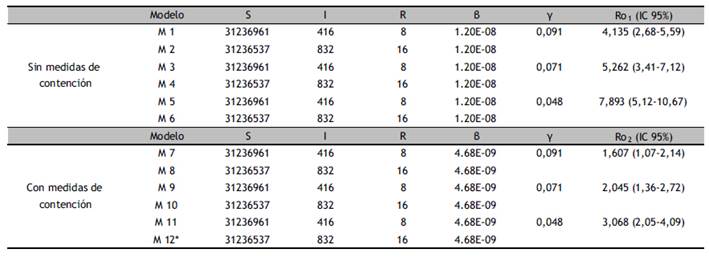
*Modelamiento con menor error de estimación (RMSE=527.27)
S: susceptibles; I: infectados; R: recuperados; γ: 1/periodo de infección; β: tasa de transmisión de la infección; Ro: Numero Reproductivo Básico.
Se observó que el tiempo para alcanzar el pico máximo de infecciones en el M 12 (usando medidas de contención) se desplaza entre 100 y 150 días; en comparación del M 6 (sin medidas de contención), que presenta un tiempo de pico máximo de infecciones entre 50 y 75 días. De igual forma, se observa la tendencia en la disminución de casos infectados usando medidas de contención. (Figura 1)
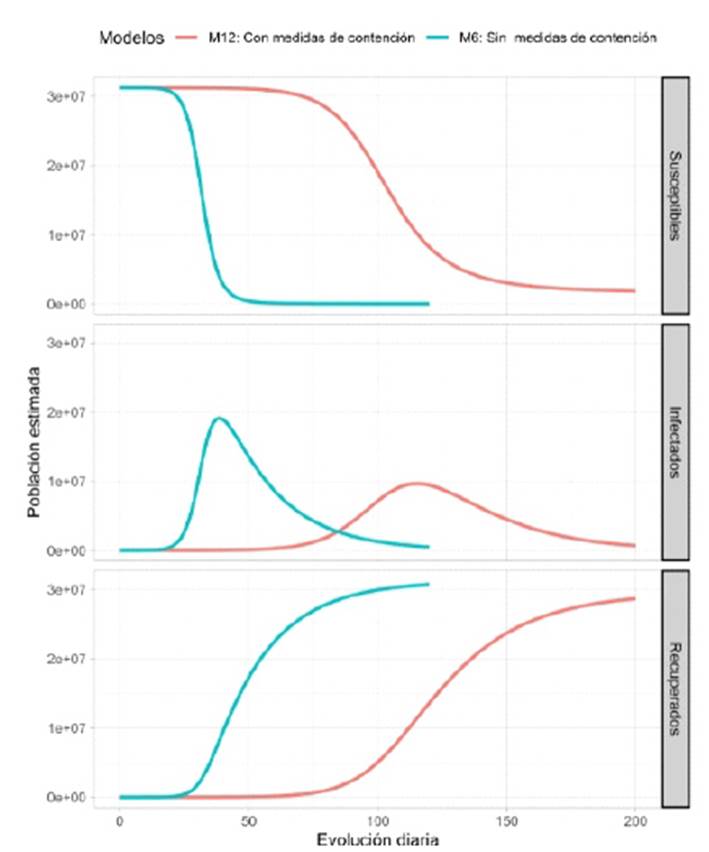
Figura 1 Comportamiento epidemiológico de la enfermedad de COVID-19 con y sin medidas de contención en población peruana, usando modelamiento SIR.
Se estimó la tendencia de la tasa de propagación de la infección en población peruana, según estimación del índice de propagación por periodo de infección y medidas de contención; observándose una menor propagación usando medidas de contención; sin embargo, ésta fue errática y superior a 1. (Figura 2)
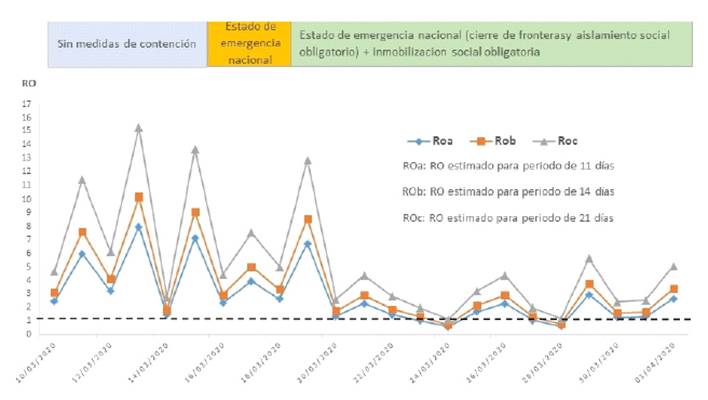
Figura 2 Estimación de la tendencia en la tasa de propagación de la enfermedad de COVID-19 en población peruana.
Se observó que bajo medidas de contención los modelos con menor tasa de propagación permiten evitar entre el 30% y el 33% de casos infectados en la población peruana hasta alcanzar el pico máximo de personas infectadas. (Tabla 2)
Tabla 2 Comparación de casos infectados estimados al pico de infección según variación de tasa de propagación ante igualdad de variables en los modelos SIR.
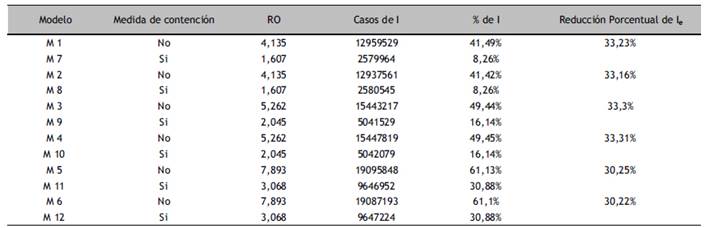
Ro: Numero Reproductivo Básico; I: infectados al alcanzar el pico de infección máxima; Ie: infectados evitados. Porcentaje calculado en base a la población estimada por INEI.
DISCUSION
En la literatura científica publicada se viene contrastando el comportamiento de la actual pandemia bajo medidas de contención practicadas en determinadas poblaciones. Entre modelos propuestos está el modelo epidemiológico SEIR en la epidemia de Covid-19 cuyo parámetro más complejo de estimar es β, que indica el número de personas infectadas asintomáticas que pueden estar infectando a otros6; este es parámetro limitante en una población como la nuestra, donde el tamizaje es selectivo y no existir estudios publicados de los parámetros bajo este modelamiento. Por otro lado, es conocido que los parámetros de modelamientos no son adecuados extrapolarlos a una población distinta7,11,12. A pesar de las limitaciones en cada variante del modelamiento epidemiológico, los resultados de modelos logísticos y SIR, realizados a medida que avanza la epidemia, son de interés para evaluar el efecto de las medidas de contención en el control de la propagación de la epidemia en una población especifica9.
En el presente estudio se realizaron 12 modelamientos, según uso de medidas de contención, transmisibilidad y periodos de infección de 11, 14 y 21 días. Estudios han reportado que el virus SARS COV-2 tiene un periodo promedio de 6 días de incubación, en el cual infecta silenciosamente13,14; y posteriormente continúa infectando alrededor de 7 a 14 días de iniciado los síntomas15. Al considerar el doble de casos infectados reportados inicialmente (Modelo 12), se ajusta con el menor error de estimación en comparación a los otros modelos del estudio; esto puede deberse a la liberación temprana del virus en un paciente infectado incluso antes de presentar síntomas, pudiendo ser esto responsable del 50% de casos nuevos, así mismo de la persistencia de eliminación del virus en las personas luego de la curación clínica14.
Se observó que sin medidas de contención (Modelo 6) se incrementa el número de casos infectados y el pico máximo de infecciones varía entre 50 a 75 días; a diferencia del modelamiento con medidas de contención (Modelo 12) que disminuye la tendencia de casos infectados estimados desplazando el pico máximo de infecciones entre 100 a 150 días. En las estimaciones del comportamiento de una epidemia comprenden aspectos cualitativos y cuantitativos6,7; sin embargo, en el contexto actual resulta impreciso considerar números exactos para casos infectados10,11,16. Por lo cual, su mayor rendimiento es conocer la evolución cualitativa, efectos porcentuales y calibrando tasas de propagación de la infección. Se estimó que el resultado de las acciones gubernamentales mediante medidas de contención representa el 61.28% en nuestra población; según la literatura se puede considerar como medidas severas de contención cuando se alcanza el 70%7.
Se obtuvieron tasas de propagación de infección que varía entre 4.13 a 7.89 sin medidas de contención y entre 1.61 a 3.06 con medidas de contención; siendo superiores a la de otros estudios10,11. Es conocido que el número reproductivo básico es un cálculo epidemiológico imperfecto y que puede variar en el tiempo incluso en una misma población12,17. En el presente periodo de estudio, las medidas actuales disminuyen la tendencia de la propagación de la enfermedad; sin embargo, este indicador se mantuvo superior a 1 en el periodo de estudio y con una tendencia errática, como para ser consideradas medidas efectivas de control (Ro <1)10,12,18. Sin embargo, con las medidas actuales se proyecta que podrían evitarse entre 30% y 33% de casos infectados en la población, hasta alcanzar el pico máximo de infecciones; de igual modo estudios han demostrado que las medidas de contención disminuyen el número de infecciones y propagación de la enfermedad19,20.
La evolución de COVID-19 en población peruana varía según medidas de contención, haciendo de estas medidas necesarias para la disminución de casos infectados a costa de aplazar el pico máximo de infección. Sin embargo, es probable que se requieran de estrategias adicionales, para que las actuales medidas, sean consideradas suficientes en el control efectivo de la propagación de esta enfermedad. Se hace necesaria una monitorización continua de estas medidas identificando puntos críticos en su práctica, según geolocalizaciones; lo que permitiría intervenciones correctivas específicas para el control de la evolución de la actual pandemia en nuestra población