Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Ginecología y Obstetricia
On-line version ISSN 2304-5132
Rev. peru. ginecol. obstet. vol.60 no.1 Lima 2014
SIMPOSIO AVANCES EN INFERTILIDAD
Origen de las aneuploidías en blastocistos humanos, analizados por la técnica de polimorfismo de nucleótido único con parental support
Origin of human blastocysts aneuploidies analyzed by single nucleotide polymorphism microarray with parental support
Eduardo Gazzo Benavides1, Gigliana Catanzaro Foppiano1, Ernesto Escudero Velando1,2, Federico Valdez León2, Luis Noriega Hoces1, Soledad Sepúlveda1
1 Grupo PRANOR, Sede Monterrico, Lima, Perú
2 Genomics Perú, Lima, Perú
RESUMEN
Introducción: El diagnóstico genético preimplantacional (PGD) por medio de las técnicas de aCGH y polimorfismo de nucleótido único (SNPs - single nucleotide polymorphism) se ha convertido en una herramienta útil en los ciclos de reproducción asistida, superando al PGD por hibridación fluorescente in situ (FISH). Estas técnicas nos permiten conocer las aneuploidías en cada uno de los cromosomas. Además, la SNPs con Parental Support (Natera, Inc) posibilita conocer el origen de la aneuploidía en los blastocistos, si por el espermatozoide o por el ovo-cito. Objetivos: Determinar la tasa de aneuplodías únicas en embriones humanos obtenidos por reproducción asistida, utilizando la técnica de polimorfismo de nucleótido único. Diseño: Estudio retrospectivo. Institución: Grupo PRANOR, Sede Monterrico, y Genomics Perú, Lima, Perú. Material biológico: Embriones humanos. Intervenciones: Análisis de los registros de 429 embriones estudiados con PGD por SNPs, embriones obtenidos de 105 ciclos de reproducción asistida, entre 2011 y 2013. Principales medidas de resultados: Aneuploidía de embriones, relación con edad materna y origen paterno o materno. Resultados: El 48,8% de embriones resultó normal, con tasa de aneuploidía de 51,2%. La proporción de embriones sanos varió de acuerdo a la edad de la madre, disminuyendo cuando la edad aumentaba. En todos los grupos etarios estudiados más de 66% de las aneuploidías fue de origen materno, incluyendo el grupo de ovodonación (OD). Conclusiones: El diagnóstico preimplantacional mediante SNPs tendría gran valor pronóstico y sería una herramienta útil para conocer el origen de las aneuploidías en los embriones de las pacientes que se someten a procedimientos de reproducción asistida.
Palabras clave: Técnica de polimorfismo de nucleótido único - SNPs, aneuploidías, origen materno, diagnóstico genético preimplantacional.
ABSTRACT
Background: Preimplantation genetic screening (PGS) using comparative genomics hybridization (aCGH) and single nucleotide polymorphism (SNP) technology has become a useful tool in assisted reproduction by improving clinical outcomes. Both techniques identify aneuploidies and chromosomal rearrangement. However, SNPs with Parental Support (Natera Inc.) give extra information of the aneuploidy origin either from the sperm or the oocyte. Objectives: To determine single aneuplodies rate in human embryos obtained by assisted reproduction using single nucleotide polymorphism microarray. Design: Retrospective study. Setting: Grupo PRANOR, Sede Monterrico, and Genomics Peru, Lima, Peru. Biologic material: Human embryos. Interventions: From 2011 to 2013, 105 patients underwent IVF/ ICSI with SNPs and Parental Support. In total, 429 embryos were analyzed and records were reviewed. Main outcome measures: Embryos aneuploidy, relation with maternal age and paternal or maternal origin. Results: From the 429 embryos 208 (48.8%) were chromosomally normal. The proportion of normal embryos decreased with increasing maternal age. In all age groups more than 66% of aneuploidies had maternal origin, including the ovodonation group (OD). Conclusions: PGS by SNPs with Parental Support resulted in good prognostic value and would be useful in determining the origin of aneuploidy in embryos of patients who underwent assisted reproduction technology.
Keywords: Single nucleotide polymorphism - SNP, aneuploidies, maternal origin, preimplantation genetic diagnosis.
INTRODUCCIÓN
De acuerdo a los análisis de diagnóstico genético preimplantacional por FISH, realizado en el tercer día de desarrollo, la mayoría de los embriones tiene aneuploidías. Y cuando se analiza los embriones en el día 5 de cultivo mediante la técnica de array CGH, aproximadamente el 50 % de estos presentan aneuploidías(1). Actualmente, el análisis mediante diagnóstico genético preimplantancional (PGD) para 24 cromosomas por medio de SNPs (single nucleotide polymorphisms (SNPs) microarrays con la tecnología informática Parental Support (Natera Inc) permite evaluar los 24 cromosomas del embrión(1,2).
Por medio de técnicas de biología molecular y algoritmos computarizados es posible no solo detectar la presencia o ausencia de los 24 cromosomas en el embrión analizado, sino que también puede detectar modificaciones cromosómicas que podrían generar embarazos con fenotipos anormales, así como conocer el origen de las aneuploidías, es decir, si la trisomía o monosomía resultante fue heredada del ovocito o el espermatozoide (1-3).
Esta técnica se realiza por primera vez en el Perú y se ha demostrado que tiene una confiabilidad superior al 99%(2-4). Nosotros consideramos que tiene un valor diagnóstico importante, tanto para el médico tratante como para la pareja que se está sometiendo a procedimientos de reproducción asistida, dado que puede ayudar a tomar decisiones, como utilizar semen de donante u óvulos de donante en futuros procedimientos y ayudar a descubrir en algunos casos el motivo de la infertilidad en la pareja.
El objetivo del presente trabajo fue describir los resultados de un programa de diagnóstico preimplantacional con SNP. Se analiza las tasas de embarazo y aborto, así como el aporte materno y paterno a las aneuploidías presentes en los embriones, en ciclos de FIV/ICSI.
MÉTODOS
En el presente estudio retrospectivo se analizó los datos colectados en el programa de SNPs microarray del Laboratorio PRANOR, sede Monterrico, entre agosto 2012 y julio 2013. Se incluyó un total de 105 ciclos de fecundación in vitro, en los que 429 blastocistos fueron analizados.
El cultivo embrionario in vitro fue desarrollado siguiendo el protocolo modificado de Sepúlveda y col.(5). Brevemente, los embriones fueron cultivados en un medio de cultivo único (Global Total, Life Global) hasta el estadio de blastocisto. En el día 4 de su desarrollo embrionario se realizó un canal en la zona pelúcida de todos los embriones (assisted hatching). Para dicho procedimiento se empleó un microscopio marca Olympus modelo XI 71 y micromanipuladores Narishige, además de un láser (Zilos Tk, Hamilton Thorne Biosciences), con series de 5 pulsos de 140 microsegundos cada uno, al 100% de potencia.
Se realizó la biopsia de trofoectodermo en el día 5 o 6 del desarrollo embrionario, dependiendo del día de blastulación de los embriones. Para este procedimiento se usó agujas de vidrio modelo Holding MPH-MED 30 de la marca Origio Inc. (Charlottesville, VA USA) para sujetar el embrión y aguja de vidrio modelo Biopsy MBB-FP-L-30 de la marca Origio Inc. (Charlottesville, VA USA) para extraer las células de trofoectodermo y despegarlas del blastocisto, ayudado con el láser (Zilos Tk, Hamilton Thorne Biosciences) por una serie de pulsos en el área de constricción. Dichos pulsos se realizaron a una intensidad de 400 microsegundos cada uno, al 100% de potencia.
Tras la biopsia de trofoectodermo, los blastocistos fueron vitrificados siguiendo el protocolo de Kitazato® (Kitazato Corporation, Biopharma), hasta esperar los resultados del análisis genético. Todas las transferencias se llevaron a cabo siempre que el análisis genético tuviera resultados normales, al menos para uno de los embriones analizados. En todos los casos donde la transferencia embrionaria fue realizada, los embriones fueron descongelados siguiendo el protocolo de la misma casa comercial.
Las células del trofoectodermo obtenidas por la biopsia fueron colocadas en un tubo estéril dentro de 5 µl de búffer para polymerase chain reaction (PCR), tras una serie de lavados en una solución hipotónica. Las muestras de trofoectodermo fueron colocadas en hielo seco y preparadas para su envío al Laboratorio Natera (Natera, USA), para su procesamiento con SNPs array con Parental Support.
Una vez recibidas la muestras en el laboratorio, estas fueron descongeladas y lavadas tres veces en wash buffer (5,6 mg/mL KCL, 6 mg/mL bovine serum albumin). Para la lisis/amplificación se utilizó dos protocolos: I) amplificación genómica total de Rubicon con búffer proteinasa K de Sigma; y, II) una variación de la amplificación desplazada múltiple con búffer PKB. Para el protocolo II, las muestras fueron colocadas en PKB (Artururs PicoPure Lysis Buffer, 50 mM ditiotreitol) e incubadas a 56*C por 1 hora; luego fueron inactivadas por calor a 95°C por 10 minutos. Las múltiples reacciones de amplificación desplazadas fueron incubadas a 30°C por 2,5 horas y luego a 95°C por 5 minutos.
Para las muestras de sangre y el hisopado bucal de ambos padres, se utilizó los kits DNeasy para sangre y para tejidos de Qiagen.
Ambos tipos de muestra fueron genotipificados utilizando el Illumina Infinium II genome-wide genotyping microarray (HapMap CNV370Quad o CytoSNP-12). Para el tejido se utilizó el protocolo estándar Infinium II y para las muestras de una sola célula se usó una modificación del protocolo estándar Infinium II.
Para el análisis de los resultados se utilizó un novedoso algoritmo, el cual usa mediciones de los SNPs de los padres y de los embriones para determinar el número de copias de los cromosomas y, a su vez, discernir el origen de la aneuploidía (2-4). El método es muy importante en análisis de muestras de ADN muy pequeñas, en las que las mediciones directas son muy susceptibles al error. No depende solamente de la intensidad de los resultados como otras pruebas y funciona excepcionalmente incluso en la presencia de tasas altas de allele dropout (4).
RESULTADOS
Resultados por embrión
Se estudió 105 ciclos de reproducción asistida, lo que resultó en 429 embriones analizados, de los cuales se obtuvo 208 embriones con resultado normal (48,5%) y 222 embriones anormales (51,7%). Cuando se analizó la tasa de embriones euploides con respecto a la edad de la madre, se encontró que la tasa cayó cuando la edad de la madre aumentaba. En mujeres de 35 años o menores, el 68% de los embriones analizados fue normal y en mujeres mayores de 35 años, la tasa de embriones normales disminuyó a 33% (figura 1).
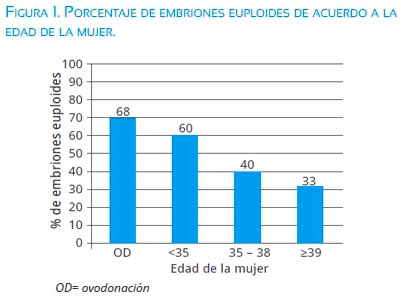
Cuando las anormalidades fueron separadas según su origen materno o paterno y se analizó los resultados según la edad materna, observamos que siempre el mayor aporte a los embriones aneuploides provenía del ovocito (figura 2).
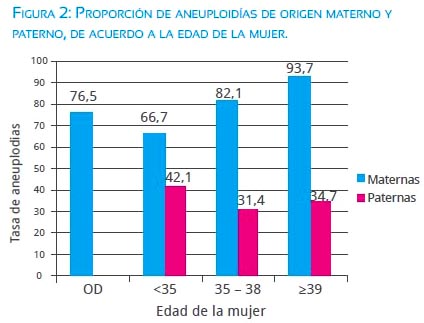
En el grupo mujeres entre 30 y 34 años (28 casos – 125 embriones analizados), el 66,7% de las aneuploidías fue de origen materno. En el grupo de mujeres entre 35 y 38 años (33 casos – 133 embriones analizados) el 82,1% de las aneuploidías fue de origen materno, y en el grupo de mujeres mayores de 38 años (30 casos – 105 embriones analizados), el 93,7% de las aneuploidías fue de origen materno.
En el grupo de las donantes (OD) con un promedio de edad de 24 años (14 casos – 66 embriones analizados), el 76,5% de las aneuploidías fue de origen ovocitario (figura 2).
Resultados por ciclos
De 105 ciclos de reproducción asistida con análisis por SNPs, 18 no tuvieron embriones sanos para transferir, con una tasa de cancelación de 17,1% (tabla 1).
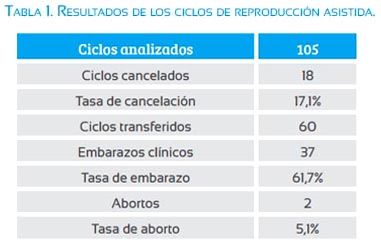
De 60 ciclos desvitrificados y transferidos se obtuvo 37 embarazos clínicos, dando una tasa de embarazo de 61,7%. Hasta la fecha, solo 2 ciclos han abortado espontáneamente (5,1%) (tabla 1).
DISCUSIÓN
En los procedimientos de reproducción asistida se ha demostrado que los embriones humanos en estadios temprano sufren de anomalías cromosómicas(6,7) y que más de la mitad de los embriones generados durante un ciclo de FIV contienen células aneuploides(8,9).
Es conocido que las anomalías cromosómicas conducen siempre a resultados negativos, como la falla en la implantación, abortos espontáneos y hasta nacimientos de niños con varios tipos de trisomías(10). Es por eso que, el poder comprender la mecánica de las aneuploidías junto con su origen parental debería ayudarnos a mejorar las tasas de éxito en los procedimientos de reproducción asistida.
La técnica de SNPs microarray se realiza por primera vez en el Perú y consideramos que es una técnica útil para el paciente que se somete a algún procedimiento de reproducción asistida, Poder tener la mayor cantidad de información sobre los gametos y embriones siempre ha sido uno de los fines de todas las investigaciones en el área reproductiva y mientras más información tengamos mejores decisiones tomaremos para conseguir el embarazo de la paciente. Uno de los aportes importantes de esta técnica es saber de quién es heredada la aneuploidía, si del ovocito o del espermatozoide. Esta información le permite al médico tratante plantear diferentes estrategias para los siguientes procedimientos.
Los resultados encontrados en este estudio sugieren que el responsable de la gran mayoría de aneuploidías es el ovocito, sin importar la edad materna.
En conclusión, el diagnóstico preimplantacional mediante SNPs tendría gran valor pronóstico y sería una herramienta útil para conocer el origen de las aneuploidías en los embriones de las pacientes que se someten a procedimientos de reproducción asistida. Los resultados muestran que las aneuploidías embrionarias provienen en su mayoría del ovocito y que estas aumentan de acuerdo a la edad, sugiriendo errores de disyunción durante la meiosis del ovocito (3).
REFERENCIAS BIBLIOGRÁFICAS
1. Vanneste E, Voet T, Le Caignec C, Ampe M, Konings P, Melotte C. Chromosome instability is common in human cleavage-stage embryos. Nat Med. 2009;15:577–83. [ Links ]
2. Rabinowitz M, Banjevic M, Demko Z, Johnson D. System and method for cleaning noisy genetic data from target individuals using genetic data from genetically related individuals. US patent application 2007. [ Links ]
3. Rabinowitz M, Singer J, Banjevicb M, Johnson D, Kijacic D, Petrov D. System and method for cleaning noisy genetic data and determining chromosome copy number. World Intellectual Property Organization published application WO/2008/115497. September 25, 2008. [ Links ]
4. Johnson DS, Gemelos G, Baner J, Ryan A, Cinnioglu C, Banjevic M, et al. Pre-clinical validation of a microarray method for full molecular karyotyping of blastomeres in a 24-h protocol. Hum Reprod. 2010;(25):1066–75. [ Links ]
5. Sepúlveda S, Garcia J, Arriaga E, Diaz J, Noriega-Portella L, Noriega-Hoces L. In vitro development and pregnancy outcomes for human embryos cultured in either a single medium or in a sequential media system. Fertil Steril. 2009;91(5):1765-70. [ Links ]
6. Rabinowitz M, Ryan A, Gemelos G, Hill M, Baner J, Cinnioglu C, Banjevic M, Potter D, Petrov DA, Demko Z. Origins and rates of aneuploidy in human blastomeres. Fertil Steril. 2012;97(2):395-401. doi: 10.1016/j. fertnstert.2011.11.034. [ Links ]
7. Wells D, Delhanty JD. Comprehensive chromosomal analysis of human preimplantationembryos using whole genome amplification and single cellcomparative genomic hybridization. Mol Hum Reprod. 2000;6:1055–62. [ Links ]
8. US Department of Health and Human Services Public Health Service, Centers for Disease Control and Prevention. Assisted reproductive technology and success rates: National summary and fertility clinic reports. Atlanta, GA:CDC; 2006. [ Links ]
9. Munne S, Chen S, Colls P, Garrisi J, Zheng X, Cekleniak N. Maternal age, morphology, development, and chromosome abnormalities in over6000 cleavage-stage embryos. Reprod Biomed Online. 2007;14:628–34. [ Links ]
10. Munne S, Magli C, Bahce M, Fung J, Legator M, Morrison L. Preimplantation diagnosis of the aneuploidies most commonly found in spontaneous abortions and live births: XY, 13, 14, 15, 16, 18, 21, 22. Prenat Diagn.1998;18:1459–66. [ Links ]
Conflictos de interés: No existen
Correspondencia:
M. Sc. Eduardo Gazzo Benavides
Celular: 981 186 648
eduardogazzo@yahoo.com














