Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO
Share
Revista Peruana de Ginecología y Obstetricia
On-line version ISSN 2304-5132
Rev. peru. ginecol. obstet. vol.65 no.2 Lima Apr./Jun. 2019
http://dx.doi.org/10.31403/rpgo.v65i2173
SIMPOSIO AVANCES EN BIOLOGÍA EN REPRODUCCIÓN ASISTIDA
El uso de la tecnología Time-Lapse y algoritmo predictivo KIDScore 5 ayuda a mejorar la selección de embriones euploides para la transferencia
Use of both the Time-Lapse technology and the KIDScore 5 predictive algorithm helps to improve selection of euploid embryos for transference
Eduardo Gazzo1,a, Fernando Peña1,a, Federico Valdez2,b, Arturo Chung1,a, Claudio Bonomini1,c, Mario Ascenzo B1,c, Marcelo Velit1,c, Ernesto Escudero1,c
1. Clínica de Fertilidad INMATER, Lima, Perú.,
2. GENOMICS PERÚ, Lima, Perú
a. Maestro en ciencias
b. Tecnólogo en citogenética (ascp)
c. Médico cirujano
ABSTRACT
Objectives: To determine if the use of the KIDScore 5 algorithm (known implantation data) can help select between euploid embryos in order to improve pregnancy and implantation rates in patients undergoing assisted reproductive procedures. Methods: Retrospective cohort study in a fertility clinic, from October 2016 to December 2018, of 1 049 embryos from 328 patients. All the embryos were cultured in the Time-Lapse, Embryoscope® incubator (Vitrolife®, Canada) for 5-6 days. Of these, 896 embryos (85.4%) were biopsied and analyzed by NGS, and assessed with the predictive KIDScore 5 algorithm (Vitrolife®, Canada). The 153 remaining embryos (14.6%) were assessed with the predictive KIDScore 5 algorithm only. 256 single euploid embryos were transferred in couples undergoing IVF treatments at the Inmater clinic’s laboratory of assisted reproduction in Lima – Peru. Results: The implantation rate was significantly higher (p = 0.004) in euploid embryos transferred when selected by the KIDScore 5 algorithm (Group 2) versus those selected using only genetic study by NGS technology (Group 1) (71.2% vs. 48.8%). The rate of implantation of the euploid embryos transferred with KIDScore value = 6 versus those transferred with KIDScore = 1 was statistically different (73.5% vs. 50.8%; p = 0.030). When assessing the relationship between the rate of euploid embryos versus the result of the KIDScore 5 value, we found highly significant differences in the rates of euploid embryos with values 6 and 5 versus those with KIDScore 0 and 1 (60.5% vs. 45.7%; p = 0.0004). Conclusions: The embryo selection with the KIDScore 5 algorithm offers advantage on implantation and pregnancy rates only when euploid embryos are transferred. Its use as an additional criterion to embryo selection should be considered when accompanied by genetic study of the embryos to be transferred. Euploid embryos with a higher value in the KIDScore 5 algorithm scale have better rates of implantation and euploidy than embryos with the minimum value of this algorithm.
Key words: Time-Lapse monitoring, Kidscore 5, Preimplantation genetic diagnosis; Single embryo transfer. .
RESUMEN
Objetivos. Evaluar si el uso del algoritmo KIDScore 5 (known implantation data) puede ayudar a seleccionar entre los embriones euploides, para mejorar las tasas de embarazo e implantación en pacientes sometidas a procedimientos de reproducción asistida. Métodos. Estudio de cohorte retrospectivo en una clínica de fertilidad, desde octubre 2016 a diciembre 2018. Se estudió 1 049 embriones provenientes de 328 pacientes. Todos los embriones fueron cultivados en la incubadora Time-Lapse, Embryoscope® (Vitrolife®, Canadá) durante 5 a 6 días. De estos, 896 embriones (85,4%) fueron biopsiados y analizados mediante NGS y recibieron una valoración otorgada por el algoritmo predictivo KIDScore 5 (Vitrolife®, Canadá). Los 153 embriones restantes (14,6%) únicamente recibieron la valoración mediante el algoritmo predictivo KIDScore 5. Se realizó 256 transferencias únicas de embriones euploides en parejas sometidas a tratamientos de FIV en el laboratorio de reproducción asistida de la Clínica Inmater, Lima – Perú. Resultados. La tasa de implantación de los embriones euploides transferidos con valores de KIDScore = 6 versus los transferidos con valores de KIDScore = 1 tuvo diferencia estadísticamente significativa (73,5% vs. 50,8%; p=0,030). Al evaluar la relación entre la tasa de euploidia embrionaria versus el resultado del valor de KIDScore 5, se obtuvo diferencias altamente significativas en las tasas de euploidia en los embriones con resultados de KIDScore 6 y 5 versus los de KIDScore 0 y 1 (60,5% vs. 45,7%; p=0,0004). Conclusiones. La selección embrionaria con ayuda del algoritmo KIDScore 5 ofrece ventaja en las tasas de implantación y embarazo únicamente cuando se transfieren embriones euploides. Su uso como criterio adicional a la selección embrionaria debiera ser considerado siempre que se acompañe estudio genético a los embriones a transferir. Los embriones euploides con valor más alto en la escala del algoritmo KIDScore 5, tienen mejores tasas de implantación y euploidía que los embriones con el valor mínimo de dicho algoritmo.
Palabras clave. Monitoreo con Time-Lapse, KIDScore 5, Diagnóstico genético preimplantacional; Transferencia de un solo embrión.
Introducción
Uno de los desafíos más importantes para los laboratorios de reproducción asistida es conseguir el embarazo con la transferencia de un solo embrión. Por esta razón, la mayoría de embriólogos se amparan en la valoración morfológica y, cuando es sugerido, el tamizaje genético preimplantacional, como herramienta indispensable para identificar cuál es el embrión que posee la mayor capacidad de implantación y llegar a un embarazo a término con un bebe sano en casa. Sin embargo, desde la incorporación a los laboratorios de las incubadoras que usan la tecnología time-lapse como el Embryoscope® y los algoritmos que derivan de los análisis morfocinéticos proporcionados por estas, los embriólogos disponen de una herramienta más para la predicción del embrión con mayor oportunidad de implantar.
La tecnología time-lapse permite el monitoreo ininterrumpido de los embriones sin necesidad de retirarlos de las incubadoras, manteniendo estables y controladas las condiciones del cultivo in vitro. Por medio de esta tecnología es posible mapear todos los eventos morfológicos del embrión, con el punto exacto de tiempo de ocurrencia(1). De esta forma se inició la búsqueda de los parámetros morfocinéticos capaces de predecir la formación a blastocistos, aneuploidías y potencial de implantación(2-4).
El algoritmo KIDScore 5 (known implantation data) es producto del análisis de las características morfocinéticas relacionadas con la implantación, que se basa en un amplio conjunto de datos de embriones procedentes de más de 24 clínicas(5). El alcance biológico de este algoritmo es significativo, ya que permite identificar embriones con desarrollo rápido o lento, patrones de división irregular o embriones que no alcanzan una etapa de desarrollo deseable para el día 3 o 5 de cultivo(5). De esta manera, el uso de este algoritmo permite excluir embriones morfológicamente normales, pero que muestran patrones de división aberrantes.
La mayoría de algoritmos de predicción hasta ahora se han desarrollado sobre una base de datos relativamente pequeños, lo que hace cuestionable su aplicación clínica en la rutina diaria de los laboratorios de reproducción asistida(5). Existen además estudios en donde se ha comparado los resultados de varios algoritmos publicados con la opinión de los embriólogos, a ver si existía concordancia en el embrión escogido para la transferencia(6), y otros en donde recomiendan que cada laboratorio debería determinar sus propios criterios de selección embrionaria basándose en sus propios datos y creando su propio modelo de algoritmo predictivo(7).
Es por ello que, este estudio tuvo como propósito comparar los resultados de las tasas de embarazo e implantación de los embriones transferidos usando el algoritmo KIDScore 5 como herramienta adicional a la valoración morfológica y al tamizaje genético.
Métodos
Estimulación ovárica
Para la estimulación ovárica se utilizó una dosis de 150 a 300 UI/día de FSH y/o HMG, durante los primeros 2 a 5 días del ciclo, de acuerdo con la edad, índice de masa corporal y respuesta a las estimulaciones previas. Cada dosis fue ajustada según la evaluación por ultrasonido, realizada a intervalos de 2 a 3 días. Por otro lado, recibieron antagonistas de la GnRH y la estimulación continuó hasta que los folículos principales alcanzaron 18 mm de diámetro medio. Para desencadenar la ovulación, se administró gonadotropina coriónica humana hCG o una agonista de la GnRH. Luego de 36 horas, se realizó la punción ovárica.
Aspiración de ovocitos y fertilización
Todos los procedimientos de aspiración folicular fueron realizados después de 36 horas de aplicada la inyección de hCG (gonadotrofina coriónica humana), vía ecografía transvaginal y bajo anestesia general; se utilizó aguja 17 (Ops Classic avec Robinet, Laboratoire CCD).
Los ovocitos recuperados fueron lavados en medio Global ® (LifeGlobal, Canadá) y cultivados en Global total for fertilization (LifeGlobal, Canadá), a una concentración de 5,6% de CO2, 5,0% de O2 y 37°C. Todos los casos fueron ingresados en incubadoras trigas K-Systems® invi cell G210, por un periodo de tres horas previa decumulación de los ovocitos. La remoción de las células de la granulosa fue realizada de manera mecánica con pipeta de vidrio. Se utilizó hialuronidasa y Global ® total with HEPES (LifeGlobal, Canadá) para enjuagar los ovocitos. Los ovocitos en estadio de metafase II fueron incubados 40 minutos antes de ser microinyectados.
Cultivo embrionario
Todos los ovocitos fueron evaluados alrededor de la hora 18 a 20 postinseminación y los que mostraban signos de fecundación fueron trasladados a una placa Embryoslide® (Vitrolife, Denmark) previamente equilibrada la noche anterior. Las placas se ingresaron al incubador Embryoscope® (Vitrolife, Denmark) y se mantuvieron los 5 a 6 días que duró el cultivo embrionario. Las placas Embryoslide® contienen 12 pocillos, cada uno con 20 uL de GTL (Vitrolife, Canadá) cubiertos con 1,8 mL de aceite mineral OVOIL (Vitrolife, Canadá) para prevenir la evaporación. Los embriones no se evaluaron ni retiraron de la incubadora hasta el día 4 de cultivo, momento en el cual se hizo la eclosión asistida de los embriones que pasarían a tamizaje genético preimplantacional. Los embriones que alcanzaron el estadio de blastocito expandido o hatching, fueron biopsiados.
Anotaciones en Time-Lapse y revisión de los vídeos
La adquisición de imágenes estuvo programada cada 15 minutos, con una resolución de 1 000 x 1 000 pixeles. Las fotografías para cada embrión fueron registradas en 7 planos focales, con distancias entre ellas de 15 um, lo que garantizaba una adecuada evaluación de la morfología embrionaria en el momento de la revisión del vídeo.
Inmediatamente después de retirar los embriones del EmbryoScope®, se revisó los vídeos por dos expertos en el manejo del software EmbryoViewer ® (EG y FP), llegando a un acuerdo imparcial cuando existió discrepancias entre sus anotaciones. Se consideró como hora de inicio de blastulación cuando el embrión mostró signos de formación del blastocele y adelgazamiento de la zona pelúcida. La valoración morfológica de la masa celular interna y trofoectodermo se realizó según los criterios de Gardner(8). Luego, con ayuda del software EmbryoViewer ® se obtuvo el valor del algoritmo KIDScore 5 para cada uno de los embriones evaluados.
Biopsia de los blastocistos
Los blastocistos fueron biopsiados inmediatamente después de ser retirados del EmbryoScope®, usando un microscopio invertido Olympus IX73, el láser Likos (Hamilton Thorne) y los micromanipuladores TransferMan 4r (Eppendorf). Las microcintas con las que se realizaron las biopsias embrionarias fueron Holding MPH-MED-30 (Origio) y Biopsy MBB-FP-M-30 (Origio).
Para realizar la biopsia se graduó el láser en modo Validation (100% power – pulsos de 430 microsegundos), y se usó un máximo de 3 disparos del láser para separar las células del trofoectodermo. Posteriormente a la biopsia se procedió a realizar el tubing según el protocolo descrito y recomendado por el laboratorio de genética Genomics Perú.
Estudio genético de los blastocistos por NGS
Un total de 1 049 embriones fueron estudiados en el Embryoscope®, de los cuales 896 fueron biopsiados para el estudio genético. Posteriormente a la biopsia los embriones fueron vitrificados y se mantuvieron a la espera del resultado.
El PGS fue realizado en el laboratorio de genética asociado Genomics Perú, utilizando un equipo Miseq® (Illumina® Inc). La amplificación del genoma completo se realizó por el método Sureplex siguiendo las indicaciones del fabricante y la preparación de las librerías se hizo usando kits de Illumina ® Veriseq. El análisis de datos de citogenética molecular se realizó por medio del software BlueFuse ilumina®.
Preparación endometrial y transferencia embrionaria
Todas las pacientes fueron tratadas con terapia hormonal de reemplazo el ciclo anterior a la transferencia. A partir del día 15 se adicionó a la terapia 3,75 mg de acetato de leuprolida vía intramuscular. Al segundo día del ciclo de la transferencia, se comenzó a administrar 6 mg diarios de valerato de estradiol vía oral. Para el soporte lúteo, se administró 800 mg/día de progesterona micronizada vía vaginal.
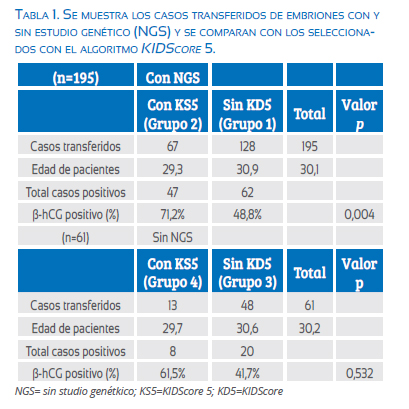
Una vez obtenidos los resultados del estudio genético, se formó dos grupos: grupo 1: 128 embriones transferidos prescindiendo de su valor KIDScore 5; grupo 2: 67 embriones transferidos previamente seleccionados por su valor KIDScore 5; para formar parte del grupo 2, las pacientes debieron tener al menos dos embriones normales y que difirieran en su valor del KIDScore (del 0 al 6), para así poder escoger el embrión con un mejor valor de KIDScore para la transferencia.
Análisis estadísticos
Para el análisis de proporciones, no paramétricas, se usó la prueba chi-cuadrado. Se consideró estadísticamente significativo cuando p<0,05. Para el análisis e interpretación de datos se usó el Statistical Package for the Social Sciences 24.0 (SPSS Inc.).
Resultados
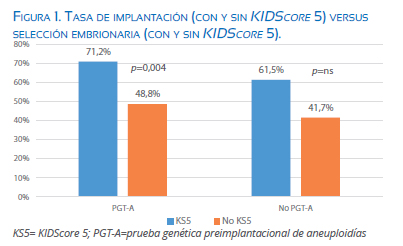
Durante el estudio, un total de 256 blastocistos únicos fueron transferidos (promedio de edad de las pacientes fue 30,6 años). Todos los blastocistos transferidos tuvieron una valoración del algoritmo KIDScore 5 (256/256), y fueron divididos en cuatro grupos: el grupo 1 estuvo conformado por 128 blastocistos euploides transferidos prescindiendo de la selección embrionaria por KIDScore 5; se reportaron 62 casos con β-hCG positiva, (tasa de implantación 48,8%); el grupo 2, por 67 blastocistos euploides transferidos con la ayuda del KIDScore 5 como método de selección adicional al NGS, y se reportaron 47 casos con β-hCG positiva (tasa de implantación 71,2%). Tabla 1. La comparación entre estos dos grupos obtuvo significancia estadística en favor de los embriones transferidos utilizando el KIDScore 5 (p=0,004).
El grupo 3 estuvo conformado por 48 blastocistos transferidos sin estudio genético y prescindiendo de su valor KIDScore 5, de los cuales se reportaron 20 casos con β-hCG positiva (tasa de implantación 41,7%); y el grupo 4 tuvo 13 blastocistos transferidos sin estudio genético y previamente seleccionados por su valor KIDScore 5; se obtuvieron 8 casos con β-hCG positiva (tasa de implantación 61,5%). La comparación entre estos dos grupos (grupo 3 y 4) no obtuvo significancia estadística (p= 0,532). Figura 1.
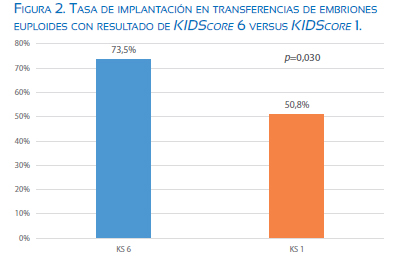
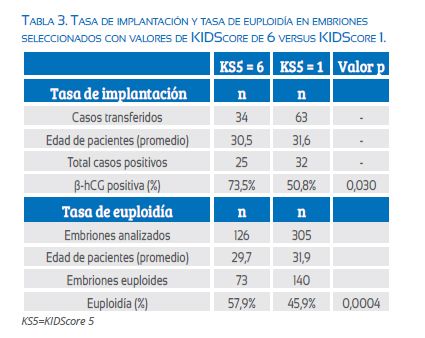
Cuando analizamos únicamente las tasas de implantación de los embriones euploides transferidos con valores de KIDScore = 6 (38 embriones) versus los que obtuvieron valores de KIDScore = 1 (69 embriones), encontramos que fue significativamente mayor (p=0,030) a favor de los embriones transferidos con el valor de KIDScore = 6 (73,5% vs. 50,8%), respectivamente. Figura 2.
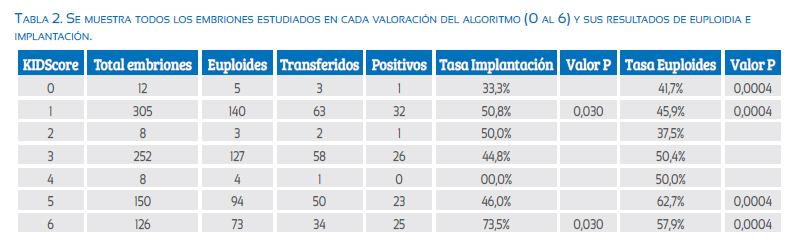
Por cada valor de KIDScore, se determinó el número de embriones que se asignaron a ese puntaje, así como los resultados de euploidia y de β-hCG positiva, y el valor p. Se puede notar que fueron muy pocos los embriones que tuvieron valores de KIDScore iguales a 0, 2 y 4. Por lo tanto, el análisis para estos tres valores queda sin efecto por ser muy pobre la casuística. Cuando evaluamos los puntajes 1, 3, 5 y 6, podemos apreciar una tendencia al aumento tanto para la tasa de implantación como para la tasa de euploidia en los embriones estudiados. Tabla 2. Ambas tasas de implantación y de euploidia mejoraron significativamente conforme mejoraba el valor otorgado por el algoritmo en estudio. Tabla 3.
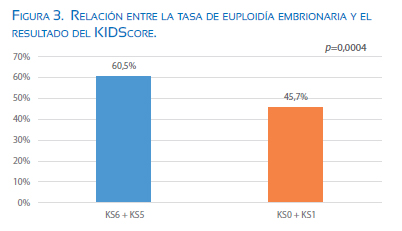
Los resultados fueron similares al seguir comparando los embriones con valores extremos de KIDScore. En este caso, agrupamos los embriones de los dos valores extremos del KIDScore, es decir, embriones con puntajes 0 y 1 versus los embriones con puntajes 5 y 6. Cuando estudiamos la tasa de euploidia que presentaron dichos grupos de embriones, encontramos que de los 276 embriones analizados con puntajes 5 y 6, 167 de ellos resultaron ser euploides (tasa de euploidía = 60,5%), mientras que de los 317 embriones analizados con puntajes 0 y 1, 145 resultaron ser euploides (tasa de euploidía = 45,7%); esta diferencia alcanzó alta significancia estadística (p = 0,0004). Figura 3.
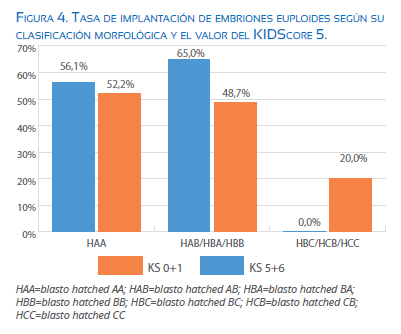
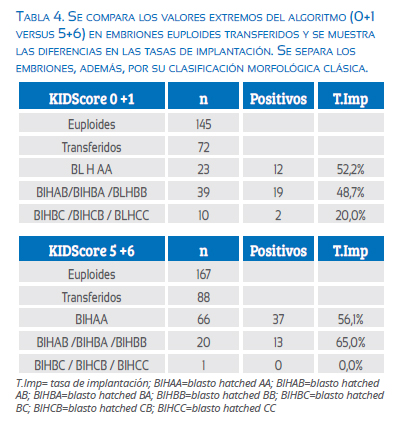
Cuando evaluamos la relación entre las tasas de implantación de los embriones euploides transferidos con su clasificación morfológica y valoración mediante el algoritmo KIDScore, no se pudo apreciar una clara evidencia de que la valoración mediante el algoritmo es más eficiente que la clasificación morfológica tradicional. Sin embargo, se aprecia una ligera tendencia hacia una mejora en las tasas de implantaciones en los embriones que fueron clasificados con el algoritmo en estudio. Figura 4 y tabla 4.
Discusión
Uno de los objetivos de este trabajo fue evaluar la utilidad de la tecnología time-lapse en el laboratorio de reproducción asistida. Para este fin, utilizamos la incubadora Embryoscope® y le insertamos el algoritmo KIDScore 5 (known implantation data) implementado especialmente para desarrollo embrionario hasta día 5 y/o 6 (Vitrolife®). Pero, sin duda, el objetivo fundamental de nuestro trabajo era establecer si esta tecnología puede ser considerada como una herramienta adicional a la valoración morfológica y el tamizaje genético preimplantacional para mejorar las tasas de embarazo e implantación en pacientes sometidas a procedimientos de reproducción asistida.
Basile, en 2014, observó que existían ciertos parámetros morfocinéticos que se encontraban y se correlacionaban con aneuploidías embrionarias, proponiendo que los embriones euploides y aneuploides se comportan de manera diferente(9). Sin embargo, hubo investigadores que obtuvieron resultados contradictorios a los del grupo de Basile. Tal es el caso de Rienzi, en 2014, quien no encontró algún tipo de correlación entre los parámetros morfocinéticos y el estatus cromosómico de los embriones que analizaron(10). Aunque muchos investigadores refieren que el monitoreo con time-lapse ha demostrado su eficiencia mejorando las tasas de embarazo e implantación, lo único que está claro hasta el momento es que el monitoreo mediante tecnología time-lapse no debe reemplazar al tamizaje genético preimplantacional (PGS)(11).
Pudimos comprobar que la selección embrionaria con ayuda del Embryoscope® y el algoritmo KIDScore 5 ofrece una clara ventaja sobre las tasas de embarazo e implantación únicamente cuando se transfieren embriones euploides seleccionados por el algoritmo en mención (71,2% vs. 48,8%). Recomendamos su uso como un criterio adicional para la selección embrionaria y debería considerarse siempre que vaya acompañado de un estudio genético a los embriones a transferir. Cuando transferimos embriones sin conocer el estatus genético, observamos clara tendencia hacia una mejor tasa de implantación en los embriones escogidos con ayuda del KIDScore (61,5% vs. 41,7%), pero la casuística no fue contundente; por ese motivo creemos que la estadística no fue significativa.
De la misma manera, se encontró que los embriones euploides con mejores valoraciones de parte del algoritmo KIDScore, tienen mejor probabilidad de implantación (KS6 = 73,5% vs. KS1 = 50,8%). Estos análisis tuvieron significancia estadística válida (p=0,030), y podríamos inferir que los tiempos de clivaje de los embriones -independientemente de su morfologíapueden estar estrechamente relacionados a mejores probabilidades de implantación. Esto se evidencia cuando observamos dos embriones euploides y morfológicamente óptimos, pero con opuestas valoraciones según el algoritmo predictivo, y únicamente embaraza el embrión con valoración más alta. Sin embargo, hace falta más evidencia al respecto para demostrar dichas hipótesis.
Cuando comparamos la eficiencia de la clasificación morfológica tradicional y la comparamos con la clasificación del algoritmo KIDScore, no pudimos observar claras diferencias a favor de ninguno de los dos métodos de selección. Creemos que la casuística para cada uno de los grupos se vio fuertemente debilitada. Sin embargo, hay tendencia hacia una mejora en los embriones que tuvieron altas valoraciones en su KIDScore. También pareciera claro que la clasificación morfológica tradicional sigue siendo una herramienta vigente y útil para clasificar embriones cuando no se dispone de otra herramienta.
Los algoritmos predictivos están comenzando a tener una singular importancia para escoger los mejores embriones a transferir, especialmente si tenemos especial interés en transferir un solo embrión, esté o no analizado genéticamente.
Definitivamente, estos resultados son preliminares y nuestro objetivo a un futuro inmediato es ampliar nuestra casuística y poder esclarecer de manera más contundente las ventajas que nos proporcionaría la tecnología time-lapse dentro de los laboratorios de reproducción asistida.
Financiamiento: por los autores
Conflictos de interés: ninguno
Citar como: Gazzo E, Peña F, Valdez F, Chung A, Bonomini C, Ascenzo M, Velit M, Escudero E. El uso de la tecnología TIME-LAPSE y algoritmo predictivo KIDScore 5 ayudan a mejorar la selección de embriones euploides para la transferencia. Rev Peru Ginecol Obstet. 2019;65(2):189-195. DOI: https://doi.org/10.31403/rpgo.v65i2173
Referencias Bibliográficas
1. Ciray HN, Campbell A, Agerholm IE, Aguilar J, Chamayou S, Esbert M, et al. Proposed guidelines on the nomenclature and annotation of dynamic human embryo monitoring by a time-lapse user group. Hum Reprod. 2014;29(12):2650–60. DOI:10.1093/humrep/deu278 [ Links ]
2. Meseguer M, Herrero J, Tejera A, Hilligsøe KM, Ramsing NB, Remohí J. The use of morphokinetics as a predictor of embryo implantation. Hum Reprod. 2011;26(10):2658–71. DOI:10 .1093/humrep/der256 [ Links ]
3. Motato Y, de los Santos MJ, Escriba MJ, Ruiz BA, Remohí J, Meseguer M. Morphokinetic analysis and embryonic prediction for blastocyst formation through an integrated time-lapse system. Fertil Steril. 2016 Feb;105(2):376-84.e9. doi: 10.1016/j.fertnstert.2015.11.001. [ Links ]
4. Campbell A. Retrospective analysis of outcomes after IVF using an aneuploidy risk model derived from Time-Lapse imaging without PGS. Reprod BioMed Online. 2013;27(2):140–6. DOI:10.1016/j.rbmo.2013.04.013 [ Links ]
5. Petersen BM, Boel M, Montag M, Gardner DK. Development of a generally applicable morphokinetic algorithm capable of predicting the implantation potential of embryos transferred on day 3. Hum Reprod. 2016;31(10):2231–44. DOI:10 .1093/humrep/dew188 [ Links ]
6. Storr A, Venetis C, Cooke S, Kilani S, Ledger W. Time-lapse algorithms and morphological selection of day-5 embryos for transfer: a preclinical validation study. Fertil Steril. 2018;109(2):276–83.e3. DOI:10.1016/j.fertnstert.2017.10.036 [ Links ]
7. Yalçınkaya E, Ergin EG, Çalışkan E, Öztel Z, Özay A, Özörnek H. Reproducibility of a Time-Lapse embryo selection model based on morphokinetic data in a sequential culture media setting. J Turkish Ger Gynecol Assoc. 2014;15(3):156–60. DOI: 10.5152/jtgga.2014.13068
8. Gardner DK, Schoolcraft WB. In vitro culture of human blastocyst. In: Janson R, Mortimer D, editors. Towards Reproductive Certainty: Infertility and Genetics Beyond. Carnforth: Parthenon Press; 1999:378–88. [ Links ]
9. Basile N, Nogales MDC, Bronet F, Florensa M, Riqueiros M, Rodrigo L, et al. Increasing the probability of selecting chromosomally normal embryos by time-lapse morphokinetics analysis. Fertil Steril. 2014;101(3):699– 704.e1. DOI:10.1016/j.fertnstert.2013.12.005 [ Links ]
10. Rienzi L, Capalbo A, Stoppa M, Romano S, Maggiulli R, Albricci L, et al. No evidence of association between blastocyst aneuploidy and morphokinetic assessment in a selected population of poor-prognosis patients: A longitudinal cohort study. Reprod Biomed Online. 2015;30(1):57–66. DOI:10.1016/j.rbmo.2014.09.012 [ Links ]
11. Basile N, Vime P, Florensa M, Aparicio Ruiz B, García Velasco JA, Remohí J, et al. The use of morphokinetics as a predictor of implantation: A multicentric study to define and validate an algorithm for embryo selection. Hum Reprod. 2015;30(2):276–83. DOI:10.1093/humrep/deu331. [ Links ]
Correspondencia:
Eduardo Gazzo
eduardogazzo@yahoo.com
Recibido: 7 enero 2019
Aceptado: 17 enero 2019
Publicado online: 8 mayo 2019