INTRODUCCIÓN
La actual infección mundial por COVID-19, causada por el nuevo coronavirus (SARS-CoV-2), en tan solo cuatro meses desde los primeros casos reportados en la ciudad Wuhan, el epicentro en China, pudo lograr infectar a millones de personas en todo el mundo1. Esta nueva pandemia se caracteriza por presentar una rápida capacidad de transmisión humano a humano y mortalidad variada, debido al síndrome de dificultad respiratoria aguda (SDRA), insuficiencia multiorgánica y otras complicaciones graves2.
Gracias a su rápida capacidad de transmisión, las tasas de infección y mortalidad (aun cundo su letalidad es relativamente baja) han llegado a superar a la de cualquier otro virus respiratorio en el presente siglo. Diversos medicamentos aún se encuentran en diferentes fases clínicas, por lo que no se cuenta con un manejo especifico3. A pesar de que la mayoría de los pacientes tienen un pronóstico favorable, aquellos con más edad y/o con condiciones crónicas subyacentes como sobrepeso, obesidad, diabetes, hipertensión, enfermedades coronarias, etc. suelen tener una mala evolución y un peor pronóstico4.
La coinfección entre otros microorganismos y el SARS-CoV-2, como virus, bacterias y hongos, podría ser un factor muy importante en COVID-19, el cual podría aumentar las dificultades en el diagnóstico, manejo, pronóstico, e incluso aumentar los síntomas y la mortalidad5. Algunos estudios sugieren que la coinfección por enterovirus (EV), rinovirus (RV), metaneumovirus (MPV), y virus sincitial respiratorio (VSR) podrían ser comunes5. Así mismo, se ha demostrado que la coinfección por bacterias como S. pneumoniae y S. aureus juegan un rol importante en la mortalidad y complicaciones en casos de otros patógenos virales, por lo que se recomienda, ante pacientes de alto riesgo, el uso de antibioterapia empírica ante una mala evolución clínica6.
Los estudios en relación al rol de estas coinfecciones en pacientes con COVID-19, siguen siendo escasas en el Perú y en general en países latinoamericanos. Pese a la escasa evidencia al respecto. La administración de antibióticos ya sea como automedicación o en el contexto de la administración hospitalaria parece ser la regla. El objetivo de la presente revisión es describir la evidencia científica publicada respecto a coinfección y su relevancia en pacientes con COVID-19.
MÉTODOS
Formulación de pregunta PECO
Las preguntas clínicas abordada para la presente revisión fueron ¿Cuál es la frecuencia de coinfección con patógenos respiratorios en pacientes con COVID-19? y ¿La coinfección por otro patógeno respiratorio es un factor de riesgo en personas con diagnóstico de COVID-19?
(P)oblación: Paciente con diagnóstico de COVID-19
(E)xposición: Haber presentado coinfección por otro patógeno respiratorio
(C)ontrol: No haber presentado coinfección por otro patógeno respiratorio
(O)outcome/desenlace: Mortalidad, ingreso a la Unidad de Cuidados Intensivos (UCI) o cualquier otro desenlace clínicamente relevante.
Criterios de elegibilidad
Fueron incluidos los estudios que cumplían con:
Ser de tipo observacionales o ensayos clínicos, que presenten datos acerca de la prevalencia de coinfección y la asociación con sus desenlaces posibles.
Estudios en pacientes con diagnóstico confirmado de COVID-19 por pruebas serológicas o moleculares.
Fueron excluidos los estudios que cumplían con:
Artículos que no presenten resultados inéditos, como editoriales, artículos de opinión, artículos de revisión o revisiones sistemáticas.
Estudios in vitro o en modelos animales.
Estudios basados en simulación estadística.
Pre prints o publicaciones que no hayan pasado un proceso de revisión por pares. Las revisiones sistemáticas relevantes fueron comentadas en el apartado de discusión.
Estrategia de búsqueda
Se llevo a cabo una búsqueda bibliográfica de estudios publicados en idioma español o inglés usando los buscadores de PubMed, The Cochrane Library y Google Scholar. Sólo se buscaron estudios publicados en el transcurso del año 2020 y 2021 hasta la fecha del 24 de enero. Los términos usados para la estrategia de búsqueda se muestran a continuación: (“coinfection” OR “coinfected” OR “coinfecting” OR "coinfections" OR "coinfects") AND (“2019 nCoV” OR 2019nCoV OR “2019 novel coronavirus” OR “COVID 19” OR “COVID-19” OR “COVID19” OR “new coronavirus” OR “novel coronavirus” OR “SARS CoV-2” OR (Wuhan AND coronavirus) OR “SARS-CoV” OR “2019-nCoV” OR “SARS-CoV-2” OR “respiratory virus”) Esta estrategia de búsqueda fue adaptada para realizar la búsqueda en cada buscador previamente mencionado.
Selección de evidencia y extracción de datos
La selección de los estudios se realizó a través de una primera etapa consistente en la identificación de los estudios potencialmente relevantes para responder la pregunta de investigación planteada a partir de los títulos y resúmenes de los resultados arrojados por los buscadores al aplicar la estrategia de búsqueda, este procedimiento fue realizado por un único investigador. Posteriormente, se procedió a una segunda etapa de lectura a texto completo de cada antecedente que haya sido identificado como potencialmente relevante, en este punto se valoraron los criterios de selección y, de cumplirse, se seleccionó el artículo para formar parte de la revisión, este proceso fue realizado por un único investigador.
RESULTADOS
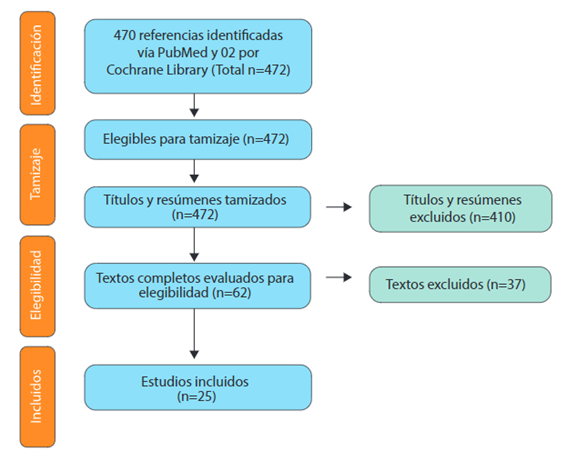
Se encontró un total de 470 artículos en la búsqueda bibliográfica. Tras la selección y eliminación de articulo duplicados, y la lectura de estos, fueron preseleccionadas 62 estudios para lectura a texto completo. Se resume este procedimiento en laFigura 1, finalmente, se incluyeron 25 estudios que cumplieron los criterios de inclusión definidos para la realización de esta investigación. (Ver Anexo 1).
Características de los estudios:
Los artículos que elegimos fueron desarrollados en diversos continentes como son América (Estados Unidos), Europa (España, Rusia, Suiza, Países Bajos, Italia, Reino Unido y Bélgica) y Asia (China, Japón, Singapur). Gran parte de los estudios fueron longitudinales, siendo 9 cohortes de tipo retrospectiva7-15, 1 cohorte prospectiva16; mientras que de los estudios transversales, 8 estudios fueron analíticos17-24y 4 estudios fueron descriptivos transversales25-28. Se incluyeron 3 cartas al científicas29-31. La mayoría de estos estudios fueron realizados en un ambiente hospitalario (incluyendo tanto a pacientes ambulatorios como hospitalizados); sin embargo, algunos también incluyeron pacientes ingresados a UCI16,21,29,30. El aumento del riesgo de mortalidad y severidad de la enfermedad también fueron identificados en algunos estudios8-11,26. El número de pacientes tuvo una amplia variabilidad (desde 24 hasta 8488).
Coinfección bacteriana
El riesgo de coinfección bacteriana fluctuó entre 0%12,19,23y 91,8%20de acuerdo a las definiciones empleadas, el tipo de pacientes incluidos y los métodos diagnósticos empleados. Los estudios realizados en base a técnicas de biológica molecular reportaron frecuencias entre 0%12,19,23y 91,8%20, mientras que los resultados basados en cultivos convencionales mostraron una frecuencia entre 1,2%32y 46%21. Las bacterias más frecuentemente encontradas en general fueron S. aureus y P. aeruginosa, aunque la enrome variabilidad impide obtener una medida resumen.
Lv et al. encontró que las bacterias más frecuentemente encontradas fueron Acinetobacter baumannii y E. coli con 12,5% ambos10; Wu Q. et al. encontró una prevalencia de coinfección de Mycoplasma pneumoniae del 14,9%, en un estudio realizado en 74 pacientes pediátricos17; por otro lado, He F. et al. encontró una prevalencia de P. aeruginosa de 29,4% y de Bordetella pertussis de 10,3%28. En Estados Unidos, Lehmann CJ. et al. encontró una prevalencia de coinfección bacteriana del 2,2%; en el que la bacteria más frecuentemente encontrada fue S. aureus con 0,6%25; en este mismo país, Hazra A. et al.23y Kim D et al.19no encontraron coinfección bacteriana en pacientes hospitalizados; en Estados Unidos también, Nori P. et al. encontró una prevalencia de coinfección bacteriana de 59,8% en 152 pacientes de la Unidad de Cuidados Intensivos (UCI); donde las bacterias más frecuentes fueron S. aureus con 44% y Pseudomona aureginosa con 16%21. En Reino Unido, en un estudio hecho en 836 pacientes, se encontró una prevalencia de coinfección por Pseudomona spp. de 1,44%, S. aureus de 0,72%, Klebsiella spp. 0,7%, Enterobacter spp. 0,6% y Serratia spp. de 0,24%9. Así también, en un estudio de 989 pacientes hospitalizados por al menos 48 horas en España, se encontró una prevalencia de coinfección bacteriana del 3,1%, siendo principalmente causada por Streptococcus pneumoniae con 1,21% y S. aureus con 0,61%8. Un estudio basado en una definición desarrollada por un equipo multidisciplinario en 1016 pacientes en Estados Unidos de Norteamérica, encontró una tasa de coinfecciones más bien baja, incluyendo 1,3% de casos de neumonía bacteriana confirmada y 1,1% de casos de neumonía bacteriana probable. Aunque el 69% de pacientes recibieron terapia antibiótica al ingreso, esta fue detenida en la mayoría de pacientes dentro las 48 horas de ingreso en ausencia de demostración de patógenos32. Otro aspecto evaluado importante fue la sobreinfección, la cual consiste en la infección bacteriana sobre agregada al paciente posterior al diagnóstico de COVID-19. En Rusia, en un estudio en 1204 pacientes hospitalizados se encontró una prevalencia de sobreinfección bacteriana en pacientes de 35,96%11; por otro lado, en Países bajos, en 48 pacientes en UCI, se encontró una sobreinfección por S. aureus del 10,4% y por Pseudomona aureginosa del 2,1%29. En China, dos estudios encontraron prevalencias de sobre infección del 10% y 15%7,24; mientras que, en pacientes con enfermedad severa y crítica, se encontró una proporción de sobre infección respiratoria del 55,3%, donde el 76,9% de los patógenos encontrados fueron bacterias30, siendo los más frecuentes K. pneumoniae con 28,95%; Enterococcus faecium con 23,68%; Acinetobacter baumannii con 21,1%. En España, la sobreinfección en pacientes hospitalizados fue del 4,7%, de estos pacientes, las principales bacterias asociadas a la ventilación mecánica fue S. aureus (0,4%), P. aeruginosa (0,3%), Stenotrophomonas maltophilia (0,2%) y K. pneumoniae (0,1%); mientras que las bacterias más comunes en neumonías intrahospitalarias fueron: S. aureus (0,1%), P. aeruginosa (0,1%), Stenotrophomonas maltophilia (0,1%) y K. pneumoniae (0,1%)8.
Tabla 1. Prevalencia de coinfección bacteriana en pacientes con COVID-19, revisión de estudios
| Autor(es) | Lugar del estudio | Tamaño de muestra | Método diagnóstico | Prevalencia de coinfección bacteriana |
|---|---|---|---|---|
| He F. et al28 | China | 194 | PCR | Coinfección: Streptococcus pneumoniae: 7,2% Bordetella pertussis: 10,3% Streptococcus pyogenes: 1,5% Staphylococcus aureus: 0,15% Neisseria meningitidis: 3,6% Haemophilus influenza: 8,8% Pseudomona aureginosa: 29,4% Global bacteriana: 50% |
| Garcia-Vidal, C et al8 | España | 989 | Cultivo de muestras respiratorias | Coinfección: Streptococcus pneumoniae: 1,1% Staphylococcus aureus: 0,61% Haemophilus influenzae: 0,2% Moraxella catarrhalis: 0,1% General: 2,5% Sobreinfección (asociada a ventilación mecánica): S. aureus: 0,4% P. aeruginosa: 0,3% Stenotrophomonas maltophilia: 0,2% K. pneumoniae: 0,1% Sobreinfección (neumonía intrahospitalaria): S. aureus: 0,1% P. aeruginosa: 0,1% Stenotrophomonas maltophilia: 0.1% K. pneumoniae: 0.1% |
| Hughes, S et al9 | Reino Unido | 836 | Cultivo de muestras respiratorias | Coinfección: Staphylococcus aureus: 0,72% Pseudomonas spp: 1,44% Enterobacter spp: 0,6% Klebsiella spp: 0,7% Serratia spp:0,24% |
| Wu, Q et al17 | China | 74 pacientes pediátricos | Prueba de ácido nucleico | Coinfección: Mycoplasma pneumoniae: 14,86% |
| Hazra, A et al23 | Estados Unidos | 459 | Panel respiratorio BioFire FilmArray 2 | Mycoplasma pneumoniae: 0% Chlamydophila pneumoniae: 0% Bordetella pertussis: 0% |
| Zhang, H et al15 | China | 38 | Cultivo o secuenciación metagenómica confirmada por RT-PCR | Infección secundaria: K. pneumoniae: 28,95% Enterococcus faecium: 23,68% Acinetobacter baumannii: 21,1% |
| Lehmann, CJ et al25 | Estados Unidos | 321 | Cultivo 21% | Coinfección: Cualquiera: 2,2% Staphylococcus aureus: 0,6% Proteus mirabilis: 0,3% |
| Panel de patógenos respiratorios | Coinfección: Bordetella parapertussis: 0,3% | |||
| Kim, D et al19 | Estados Unidos | 116 | Panel de patógenos respiratorios | Coinfección: Chlamydia pneumoniae: 0% Mycoplasma pneumoniae: 0% |
| Lv, Z et al10 | China | 354 | Hemocultivo | Coinfección: Acinetobacter baumannii: 12,5 % E. coli: 12,5 % Staphylococcus haemolyticus: 5% P. aeruginosa: 5% Mycoplasma pneumoniae: 2,5% Stenotrophomonas maltophilia: 2,5% Enterococcus faecium: 2,5% |
| Cultivo de lavado bronquio alveolar | ||||
| Zhu, X et al20 | China | 257 | RT-PCR | Coinfección: 91,80% |
| Sharov, Konstantin S11 | Rusia | 1204 | Cultivo y subsecuente prueba bioquímica o serológica | Sobreinfección: 35,96% |
| Zhou, F et al7 | China | 191 | Cultivo | Infección secundaria: 15% |
| Huang, C et al24 | China | 41 | Cultivo | Infección secundaria: 10% |
| Hirotsu, Y et al12 | Japón | 40 | Panel respiratorio FilmArray | 0% |
| Verroken A et al16 | Bélgica | 32 (UCI) | Panel respiratorio FilmArray | Coninfección: 40,60% |
| Nieuwenhuis MB et al29 | Países bajos | 48 (UCI) | PCR | Superinfección: Staphylococcus aureus: 10,4% P. aureginosa: 2,1% |
| Intra J et al30 | Italia | 61 (UCI) | Sistema de Espectrometría de Masas de Ionización-Tiempo de Vuelo con Desorción Láser Asistida por Matriz | Colonización pulmonar: P. aureginosa: 9,8% Staphylococcus aureus: 3,3% K. pneumoniae: 1,6% Escherichia coli: 1,6% Klebsiella oxytoca: 1,6% Enterobacter cloacae: 1,6% Staphylococcus epidermidis: 1,6% |
| Nori P et al21 | Estados Unidos | 152 (99 de ellos en UCI) | Cultivos de muestras respiratorias | Coinfección: Staphylococcus aureus: 44% P. aureginosa: 16% Klebsiella spp: 10% Enterobacter spp: 8% E. coli: 4% General: 46% |
Tabla 2. Prevalencia de coinfección viral en pacientes con COVID-19, revisión de estudios
| Autor(es) | Lugar del estudio | Tamaño de muestra | Método diagnóstico | Prevalencia de coinfección viral |
| Garcia-Vidal, C et al8 | España | 989 | PCR | Coinfección: Influenza A: 0,4% Influenza B: 0,2% Virus sincitial respiratorio: 0,1% |
| Wu, Q et al17 | China | 74 | Prueba de ácido nucleico | Infección secundaria: Virus sincitial respiratorio: 2,7% Virus de Ebstein Barr: 2,7% Citomegalovirus: 2,7% Influenza A: 1,35% Influeza B: 1,35% |
| Hazra, A et al23 | Estados Unidos | 459 | Panel respiratorio BioFire FilmArray 2 | Coinfección: Adenovirus: 0,4% Coronavirus NL63: 0,2% Human metapneumovirus: 0,4% Influenza A: 0,7% Parainfluenza 2: 0,2% Resto 0% |
| Zhang, H et al15 | China | 38 | mNGS positivo confirmado por RT-PCR | Coinfección: 15,79% |
| Lehmann, CJ et al25 | Estados Unidos | 321 | Panel respiratorio FilmArray | Coinfección: Influenza A: 0,9% Rinovirus/enterovirus: 0,6% |
| Kim, D et al19 | Estados Unidos | 116 | Panel de patógenos respiratorios | Coinfección: Influenza A: 0,9% Influenza B: 0% Virus sincitial respiratorio: 5,2% Parainfluenza 1: 0,9% Parainfluenza 2: 0% Parainfluenza 3: 0,9% Parainfluenza 4: 0,9% Metapneumovirus: 1,7% Rinovirus/enterovirus: 6,9% Adenovirus: 0% Otros coronavirus: 4,3% |
| Lv, Z et al10 | China | 354 | Kit de detección de ácidos nucleicos patógenos | Coinfección: Boca virus: 1,26% (de los evaluados) |
| Zhu, X et al20 | China | 257 | RT-PCR | Coinfección: 31,50% |
| Sharov, KS11 | Russia | 1204 | Cultivo y subsecuente prueba bioquímica o serológica | Coinfección: 26,08% |
| Hirotsu, Y et al12 | Japón | 40 | Panel respiratorio FilmArray | Coinfección: Rinovirus/enterovirus: 27,5% Metapneumovirus: 17,5% Coronavirus 229E: 10% Coronavirus OC43: 7,5% Adenovirus: 5% Virus sincitial respiratoria: 5% Coronavirus NL63: 2,5% Adenovirus: 2,5% |
| Ma, S et al13 | China | 93 | Inmunofluorescencia indirecta de IgM | Coinfección: Virus de la influeza A: 47,3% Virus de la influenza B: 2,2% |
| Yue, H et al18 | China | 307 | Serología IgM | Coinfección: Influenza A: 49,8% Influenza B: 7,5% |
| Nowak, MD et al14 | Estados Unidos | 1204 | Panel Respiratorio PCR2, Flu/RSV PCR | Coinfección: Influenza A: 0,08% Influenza B: 0% Virus sincitial respiratorio: 0,31% Coronavirus NL63: 0,63% Coronavirus HKU1: 0,45% Coronavirus 229E: 0,36% Coronavirus OC43: 0,09% Rinovirus/enterovirus: 0,73% Metapneumovirus: 0,36% Adenovirus: 0,18% Parainfluenza: 0% |
| Leuzinger, K et al27 | Suiza | 930 | Panel viral múltiple respiratorio | Coinfección: Coronavirus humano: 0,54% Rinovirus: 0,54% Virus parainfluenza: 0,32% Influenza A: 0,22% Adenovirus: 0,11% Virus sincitial respiratorio: 0,11% |
| Si, Y et al22 | China | 24 | Panel viral múltiple respiratorio | Coinfección: 4,20% |
| Lin, D et al31 | China | 92 | Kit de detección rápida multiplex 2.0 | Coinfección: 3,20% |
| Wee, LE et al26 | Singapur | 431 | Panel de patógenos virales respiratorios | Coinfección: 1,39% General 0,69% Rhinovirus 0,46% Parainfluenza 0,23% otro Coronavirus coronavirus |
Tabla 3. Prevalencia de coinfección fúngica en pacientes con COVID-19, revisión de estudios
| Autor(es) | Lugar del estudio | Tamaño de muestra | Método diagnóstico | Prevalencia de coinfección fúngica |
| Hughes, S et al9 | Reino Unido | 836 | GeneXpert | Infección secundaria: Candida spp: 2,87% Aspergillus spp: 0,36% |
| Zhang, H et al15 | China | 38 | RT-PCR | Coinfección: 5,26% |
| Lv, Z et al10 | China | 354 | Cultivo de lavado bronquio alveolar | Coinfección: Candida albicans: 7,% Candida tropicalis: 2,5% Candida parapsilosis: 2,5% Candida lusitaniae: 2,5% |
| Zhu, X et al20 | China | 257 | RT-PCR | Coinfección: 23,30% |
| Intra J et al 30 | Italia | 61 (UCI) | Cultivo de lavado bronqueo alveolar | Coinfección: Candida albicans: 23% Candida glabrata: 6,6% Aspergillus fumigatus: 1,6% |
Tabla 4. Principales desenlaces ante la presencia de coinfección o sobreinfección en pacientes con COVID-19, revisión de estudios
| Autor(es) | Lugar del estudio | Tamaño de muestra | Desenlaces |
|---|---|---|---|
| Garcia-Vidal, C et al8 | España | 989 | La coinfección por un patógeno bacteriano, viral o fúngica aumento el ingreso a UCI en casi el doble (25,8% vs 11,9%; p=0,02), pero no hubo relación estadísticamente significativa con la mortalidad (16,1% vs 9,4%; p=0,21). La sobreinfección por un patógeno bacteriano, viral o fúngica aumentó el ingreso a UCI (67,4% vs 11,9%; p<0,001) y la mortalidad (18,6% vs 9,4%; p=0,047). |
| Hughes, S et al9 | Reino Unido | 836 | Tener coinfección detectada por cultivo de sangre se asoció con un RR de mortalidad de 1,51 (p=0,3543), mientras que tener coinfección detectada por cultivo de esputo tuvo un RR de 0,90 (p=0,8462). |
| Zhang, H et al15 | China | 38 | Los pacientes con infección secundaria tuvieron: una menor tasa de alta hospitalaria en un seguimiento de 60 días (log rank p<0,001), mayor necesidad de ventilación mecánica (86,36% vs 25%; p<0,0001) y mayor mortalidad (36,4% vs ~7%). |
| Lv, Z et al10 | China | 354 | La coinfección con otros patógenos respiratorios (bacteriana o fúngica), así como los linfocitos y el dímero D se asociaron con la gravedad de COVID-19 (R=0,375 p<0,001). |
| Sharov, Konstantin S11 | Rusia | 1204 | La mortalidad en pacientes sin coinfección fue de 1,3%, en pacientes con coinfección viral 4,2%, en pacientes con sobreinfección bacteriana 18,2% y en pacientes con coinfección viral y bacteriana 15,1%. |
| Ma, S et al13 | China | 93 | La mortalidad entre pacientes coinfectados por influenza fue de 47,82% y de no coinfectados fue de 46,81%, sin asociación estadísticamente significativa. |
| Wee, LE et al26 | Singapur | 431 | La mortalidad en coinfectados fue 0% (0/6), mientras que en no coinfectados fue 0,23% (1/425), sin haber asociación estadísticamente significativa. |
Coinfección viral
El riesgo de confección viral fluctuó entre 0,08%14y 49,8%18, siendo los virus más frecuentes Influenza A, Influenza B, Virus sincitial respiratorio y Rinovirus/enterovirus. Con respecto a las coinfecciones virales; en un estudio en Rusia, la prevalencia de coinfección viral fue de 26,1%11. Los estudios en China encuentras prevalencias de coinfección viral que van del 3,2% al 31,5%20,22,31; el virus más frecuentemente encontrado fue el de la influenza A, con 47,3% y 49,8% en dos muestras diferentes de pacientes hospitalizados, mientas que el virus de la influenza B se encontró en 2,2% y 7,5%; en las respectivas muestras13,18. En niños en cambio, los virus más frecuentemente encontrados fueron el Virus sincitial respiratorio (2,7%), virus de Ebstein Barr (2,7%) y citomegalovirus (2,7%); por encima de los virus de la Influenza A y B, con 1,35% cada uno. Por otro lado, los estudios realizados en Estados Unidos encuentran prevalencias de coinfección por el virus de la influenza A menores a las reportadas en China, como 0,08%14, 0,7%23o 0,9%19,25; mientras que el virus de la influenza B estuvo ausente en los estudios revisados14,19,23; otros virus como el rinovirus/enterovirus se encontró coinfectando en 0,6%25, 6,9%19y 0,73%14; el virus sincitial respiratorio en 5,2%19y 0,31%14; mientras que otros coronavirus se encontraron coinfectando en 4,3%19, o menos14, a pacientes con COVID-19. Resultados similares se encontraron en España, donde la coinfección viral fue de 0,4% para Influenza A, 0,2% para Influenza B y 0,1% para el virus sincitial respiratorio8. Por su lado, en un estudio con 930 pacientes en Suiza, se encontró coinfección por coronavirus humano de 0,54%, rinovirus de 0,54%, virus parainfluenza de 0,32%, influenza A de 0,22%, adenovirus de 0,11% y virus sincitial respiratorio de 0,11%27. Por su parte, en un estudio de 40 pacientes en Japón, la coinfección viral para rinovirus/enterovirus fue del 27,5%, metapneumovirus 17,5%, coronavirus 229E 10%, coronavirus OC43 7,5%, adenovirus 5%, virus sincitial respiratoria 5%, coronavirus NL63 2,5% y adenovirus 2,5%12. La sobreinfección por virus fue reportada en menos estudios, siendo la frecuencia de 15,79% en un estudio realizado en 38 pacientes con enfermedad grave o severa15.
Coinfección fúngica
La coinfección fúngica se reportó con una frecuencia entre 0,36%(9)hasta 23,3%20, siendo Candida spp y Aspergillus los hongos reportados con mayor frecuencia. La coinfección fúngica fue reportada en menor frecuencia, los estudios en China señalan que ésta se mantuvo entre 5,26%15y 23,3%20; Lv et al. encontró que, del cultivo obtenido a partir de lavado bronquio alveolar a 354 pacientes, la prevalencia de coinfección de Candida albicans fue de 7,5%, de Candida tropicalis fue 2,5%, de Candida parapsilosis fue 2,5% y de Candida lusitaniae fue 2,5%10. En Italia, en un estudio de 61 pacientes internados en UCI, se encontró que la coinfección por Candida albicans fue de 23%, por Candida glabrata fue de 6,6% y por Aspergillus fumigatus fue de 1,6%30. Mientras que, con respecto a la sobre infección, en Reino Unido, usando el método diagnóstico GGG en 836 pacientes, la prevalencia para Candida spp fue de 2,87% y para Aspergillus spp fue de 0,36%9.
Riesgos asociados a la coinfección
García Vidal et al. indico que la coinfección aumento el ingreso a UCI en casi el doble (p=0,02), pero no hubo relación estadísticamente significativa con la mortalidad (p=0,21). Por otro lado, la sobreinfección aumento significativamente el ingreso a UCI (p<0,001) y la mortalidad (p=0,047)8. De la misma manera Hughes et al. menciona que aquellos pacientes que tenían patógenos identificados en sangre tenían un mayor riesgo relativo de muerte, pero no estadísticamente significativo, en comparación con el valor inicial de los pacientes ingresados (P: 0,3543)9. Por otro lado, Zhang, H et al.15indico hubo una menor tasa de alta hospitalaria en pacientes con infección secundaria en un seguimiento de 60 días (log rank p <0,001), mayor necesidad de ventilación mecánica (86,36% vs 25%; p<0,0001) y mayor mortalidad (36,4% frente a alrededor del 7%). Asimismo Lv Z et al.10indica que ante la coinfección con patógenos bacterianos o fúngicos se obtuvieron valores de linfocitos y dímero D asociados a la gravedad de COVID-19 (R=0,375 p<0,001). Respecto a la mortalidad Sharov KS11señala que en pacientes sin coinfección fue de 1,3%, en pacientes con coinfección viral 4,2%, en pacientes con sobreinfección bacteriana 18,2% y en pacientes con coinfección viral y bacteriana 15,1%. En contraste con estudios de Ma, S et al.13y Wee LE et al.26donde indican que no hubo una asociación estadísticamente significativa en relación a mortalidad con paciente coinfectados o no coinfectados.
Coinfección en pacientes críticos
Cuatro estudios evaluaron la coinfección bacteriana en pacientes críticos16,21,29,30, siendo el microorganismo común entre los cuatro estudios el Staphyloccoco Aureus, además se identificó a la Pseudomona auroginosa y Klebsiella neumoniae tanto como causa de coinfección, superinfección y colonización bacteriana. Se identificó un estudio en relación a la coinfección por hongos en pacientes críticos, este estudio realizado por Intra J et al, menciona que se aislaron hongos patógenos como Candida albicans (23%), Candida glabrata (6,6%) y Aspergillus fumigatus (1,6%)30.De acuerdo con Lv Z et al, los pacientes críticos presentaron la mayor mortalidad, conteo de linfocitos más bajo, niveles de dímero D más altos, y la tasa de coinfección con bacterias/hongos más alta10.
DISCUSIÓN
La presencia de coinfección fue extremadamente variable de acuerdo con el tipo de paciente, método diagnóstico empleado y país de estudio. Los resultados de las revisiones sistemática y metaanálisis previos muestran resultados heterogéneos. Los estudios realizados en China encontraron prevalencias altas de coinfección bacteriana, desde 50% en un estudio de 194 pacientes28, hasta 91,8 % en un estudio en 257 pacientes hospitalizados20.
Lansbury et al.1en un metaanálisis con una población conjunta de 3834 pacientes encontró una prevalencia de coinfección bacteriana del 7%; asimismo, otro metaanálisis con una población conjunta de 3338 encontró que la coinfección bacteriana fue del 3,5%33Con respecto a las coinfecciones virales, las revisiones sistemáticas encuentran prevalencias de 3%1y 8,45%34; siendo 1,49% la frecuencia de coinfección por Influenza A y 0,42% por Influenza B35.
La ausencia de criterios estandarizados para definir la presencia de coinfecciones no permite estimar la problemática de la coinfección a nivel mundial. De hecho, la enrome heterogeneidad de los resultados no permite realizar medidas de síntesis (metaanálisis). Asimismo , la frecuencia de coinfección depende en gran medida del país en donde se realiza el estudio. Los resultados no son extrapolables entre los diferentes países. No se identificaron estudios en Latinoamérica, por lo que ese necesario contar con más información a nivel regional.
La mayoría de los estudios no diferencian colonización de coinfección. Esto es particularmente importante para los estudios de biología molecular que muestran reportes de coinfección con prevalencias mucho mayores que los estudios basados en cultivos bacterianos. Por otro lado, los cultivos bacterianos convencionales son en general poco sensibles para la detección de patógenos respiratorios36,37.
Muchos de los estudios más recientes y en particular aquellos que han desarrollado definiciones estandarizadas de coinfección (diferenciándola explícitamente de la colonización) muestran que las coinfecciones parecen ser relativamente escasas. Sin embargo, dado que la variabilidad entre países hace que los resultados mostrados no sean extrapolables al Perú, es importante generar evidencia local para apoyar la toma de decisiones. Esto es particularmente importante en contextos como el peruano, donde el uso irracional de antibióticos frente a COVID tiene niveles alarmantes38,39.
En muchos estudios la presencia de coinfección se asoció a la presencia de desenlaces desfavorables. Esto podría ser relevante, en particular en pacientes con cuadros de evolución más agresivos. Sin embargo, la evidencia al respecto aun no es concluyente. Es necesario el desarrollo de estudios que permitan evaluar esta posible asociación. Si la coinfección juega un rol importante en mortalidad o complicaciones, debería considerarse la mejora en los métodos basados en cultivos convencionales y la implementación de diagnóstico molecular en la mayoría de hospitales que atienden a los pacientes más críticos.
Dentro de las limitaciones de la presente revisión, se encuentra que en muchos estudios no se hacía una diferencia clara acerca del reporte de coinfección y colonización, por lo que éstas proporciones podrían haber sido sobreestimadas. Por otro lado, siendo que el riesgo de coinfección depende mucho de localización geográfica y las características de la población, la ausencia de estudios en nuestra región constituye también una limitación, ya que no se pudo describir una realidad similar a la que se puede encontrar en el Perú, en la que el uso de antibióticos de manera empírica no es precisamente infrecuente.
CONCLUSIÓN
La revisión incluye 25 artículos procedentes de diversos continentes (América, Asia y Europa). Todos los estudios contaron con pacientes con diagnostico confirmado de COVID-19 sumado a alguna otra prueba que identifico alguna coinfección. Acotamos que los estudios cuentan con poblaciones muy heterogéneas en cantidad y perfil clínico y epidemiológico.
Se identificaron 18 artículos que mostraron coinfección bacteriana, el riesgo de coinfección bacteriana llegó hasta 91,8% con métodos moleculares y 46% con cultivo convencional. Las bacterias más frecuentemente encontradas fueron S. aureus y P. aeruginosa.
En relación con la coinfección viral, se identificaron 17 artículos, donde el riesgo de confección viral llegó hasta 49,8%, siendo los agentes más frecuentes Influenza A, Influenza B, Virus sincitial respiratorio y rinovirus/enterovirus.
Cinco artículos reportaron coinfección fúngica, uno de ellos en pacientes críticos; la cual llegó hasta 23,3%, siendo los agentes fúngicos más frecuentes Cándida spp y Aspergillius spp.
Los hallazgos mostraron una mayor frecuencia a padecer enfermedad grave y de mortalidad ante la presencia de coinfección, sin embargo, la heterogeneidad de los resultados implica la necesidad de estudios más específicos.











 text in
text in