Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO  uBio
uBio
Compartir
Revista de Investigaciones Altoandinas
versión On-line ISSN 2313-2957
Rev. investig. Altoandin. vol.20 no.4 Puno oct./dic. 2018
http://dx.doi.org/10.18271/ria.2018.420
ARTÍCULO ORIGINAL
Identificación molecular de bacterias ácido lácticas con propiedades probióticas aisladas del intestino posterior de tilapia del Nilo (Oreochromis niloticus)
Molecular identification of lactic acid bacteria with probiotic properties isolated from the posterior intestine of Nile tilapia (Orechromis niloticus)
Arnaldo E. Castañeda1,2*, Jorge L. Aguilar 2,3, Adrian E. Zatan2,3, Odalis E. Toledo1,2, Manuel A. Feria1,2 & Deysy Castillo2,3
1 Empresa de investigación y capacitación en biotecnología molecular, Incabiotec – Tumbes, Perú
2 Empresa en investigación y capacitación en biotecnología piscícola, Pezbiotec – Tumbes, Perú
3 Facultad de Ingeniería Pesquera y Ciencias del Mar, Universidad Nacional de Tumbes – Tumbes, Perú
* Autor para correspondencia, e-mail: arnaldoc_ve@hotmail.com
RESUMEN
Las bacterias probióticas en la acuicultura representan una alternativa de control y prevención sobre agentes patógenos, además, reducen el uso de antibióticos. Sin embargo, resulta necesario la búsqueda de otros grupos de bacterias con similares o mejores propiedades, obtenidas incluso de la misma fuente. El propósito del estudio fue identificar de forma molecular, bacterias acido-lácticas con propiedades probióticas aisladas del intestino posterior de tilapia del Nilo (Oreochromis niloticus). Desde noviembre del 2017 hasta marzo del 2018 se desarrolló el estudio evaluándose las actividades, proteolítica, amilolítica, de sensibilidad antibiótica y antagónica frente a los siguientes patógenos: Aeromonas hydrophila, Aeromonas veronii, Pseudomonas aeruginosa, Pseudomonas putida y Plesiomonas shigelloides. Se identificó mediante el secuenciamiento del gen ARNr 16S que, Enterococcus faecium (IP5-2a) y Pediococcus pentosaceus (IP5-2b) inhibieron el crecimiento de agentes patógenos, además, presentaron sensibilidad frente a los antibióticos cloranfenicol, amoxicilina/ácido clavulánico, gentamicina, eritromicina y tetraciclina, respectivamente. Los resultados muestran que E. faecium y P. pentosaceus poseen las mejores actividades antagónicas, propiedad de mayor relevancia para determinar una bacteria potencialmente probiótica. Por lo tanto, se concluye que las bacterias identificadas en el presente estudio son buenos candidatos para ser considerados probióticos, sin embargo, se recomienda confirmar sus propiedades mediante estudios in vivo.
PALABRAS CLAVES: bacterias acido-lácticas, probióticos, identificación molecular, intestino, tilapia.
ABSTRACT
Probiotic bacteria in aquaculture represent a control alternative and prevention of pathogens, in addition, reduce the use of antibiotics. However, it is necessary to search for other groups of bacteria with similar or better properties, obtained even from the same source. The aim of the study was to identify molecularly lactic acid bacteria with probiotic properties isolated from the posterior intestine of Nile tilapia (Oreochromis niloticus). The study was carried out from November 2017 to March 2018 evaluating the antagonistic, proteolytic, amylolytic, and antibiotic sensitivity activities, and antagonistic to the following pathogens: Aeromonas hydrophila, Aeromonas veronii, Pseudomonas aeruginosa, Pseudomonas putida and Plesiomonas shigelloides. It was identified by sequencing the 16S rRNA gene that Enterococcus faecium (IP5-2a) and Pediococcus pentosaceus (IP5-2b) inhibited the growth of pathogens, in addition, were sensitive to the antibiotics chloramphenicol, amoxicillin/clavulanic acid, gentamicin, erythromycin and tetracycline, respectively. The results show that E. faecium and P. pentosaceus possess the best antagonistic activities, property of greater relevance to determine a potentially probiotic bacterium. Therefore, it is concluded that the bacteria identified in the present study are good candidates to be considered probiotic, however, it is necessary to confirm their in vivo properties.
KEYWORDS: lactic acid bacteria, probiotics, molecular identification, intestine, tilapia.
INTRODUCCIÓN
Después de la carpa (Cyprinus carpio), la tilapia (Oreochromis sp.) se ha convertido a nivel mundial, en la especie de mayor producción acuícola por su acelerado crecimiento, tasa comercial y estabilidad de sus precios (Wang & Lu, 2016; FAO, 2018). Sin embargo, existe mayor susceptibilidad a enfermedades y comportamiento animal debido a, la carga ambiental que se generan (Argota et al., 2018) incluyendo a las propias producciones de intensificación acuícolas (Peso-Echarri et al., 2012). Bajo las condiciones de estrés, la proliferación de agentes patógenos como los virus, protozoos, hongos y bacterias se favorece. En el caso de las bacterias, representan el grupo microbiano fundamental sobre la aparición de enfermedades infecciosas y entre las especies se hallan Aeromonas hydrophila, Streptococcus agalactiae, Streptococcus iniae, Vibrio sp., Edwarsiella tarda, Flavobacterium columnare y Pseudomonas sp. entre otras (Asencios et al., 2016; Hiucab-Pech et al., 2016, 2017).
Dentro de las aplicaciones para la reducción de enfermedades infecciosas está el uso o administración oral de antibióticos (Rodgers & Furones, 2009) pero el principal efecto perjudicial o limitante son las bacterias oportunistas que aparecen ante la modificación sobre la microbiota intestinal (Romero et al., 2012; Modi et al., 2014) siendo más acuciante por la baja eficiencia metabólica relacionada con los antibióticos donde se excretan en alto porcentaje mediante las heces y la orina (Burridge et al., 2010; Cabello et al., 2013). De este modo, gran alerta existe por la presencia de antibióticos (contaminación) en el medio acuático como la resistencia bacteriana (Bidhan et al., 2014; Yang et al., 2014). Como consecuencia preventiva para protección y mantenimiento de la sanidad acuícola (Martínez et al., 2012; Zorriehzahra et al., 2016), algunas alternativas como los probióticos (Lazado & Caipang, 2014; Mohapatra et al., 2014) son considerados y entre ellos, las bacterias acido-lácticas extraídas del intestino de los peces siguen siendo referencias (Hai, 2015; Reda et al., 2017). Entre las diversas respuestas observadas mediante el uso de las bacterias como probióticos está la producción de enzimas proteasas y lipasas (Zorriehzahra et al., 2016), modulación del sistema inmune (Pirarat et al., 2011; Lazado et al., 2011) y la producción de péptidos antimicrobianos (Sihag & Sharma, 2012; Bidhan et al., 2014; Akhter et al., 2015). A pesar de lo planteado, continúa siendo un reto científico para la acuicultura, la búsqueda de bacterias con estas propiedades.
El propósito del estudio fue identificar de forma molecular, bacterias acido-lácticas con propiedades probióticas aisladas del intestino posterior de tilapia del nilo (Oreochromis niloticus).
MATERIALES Y MÉTODOS
El presente estudio se desarrolló en el laboratorio de investigación y capacitación en biotecnología molecular Incabiotec ubicado en el Departamento de Tumbes – Perú, durante los meses de noviembre 2017 a marzo 2018. Para su elaboración se seleccionaron dos individuos de Oreochromis niloticus aparentemente sanos con un peso corporal de 60±5g del Centro Colectivo Educativo Experimental de Biología y Biotecnología Acuática y Acuícola de Puerto Pizarro (CEBAP) ubicado en Puerto Pizarro, Tumbes.
Los peces fueron eutanasiados cumpliendo con los estándares de ética de manejo de animales, se realizó una anestesia previa con eugenol a concentración de 286.55 mg/l (Vidal et al., 2008). La piel se desinfectó con etanol al 70% y se diseccionó la cavidad peritoneal con bisturí estéril. Posteriormente se realizó un frotis intestinal de la región posterior utilizando hisopos estériles y se colocaron en caldo Man Rogosa Sharpe (MRS) a 28ºC en microaerofilia durante 24 h. Se realizaron seis diluciones en (desde 10-1 hasta 10-6) y se subcultivaron en placas Petri con agar MRS a 28°C durante 24 a 32 horas. Las colonias bacterianas se subcultivaron en placas con medio MRS hasta lograr su purificación mediante la visualización de un solo tipo de morfología bacteriana por tinción de Gram y se les asignó una codificación de acuerdo a su localización como IP (intestino posterior), los códigos fueron los siguientes: P3-2a, IP3-2b, IP3-d, IP4-1a, IP4-2a, IP5-2a, e IP5-2b. Las bacterias se almacenaron a -20°C con una solución de glicerol a 18 % del volumen.
Se evaluó la actividad hemolítica de los aislados utilizando placas Petri con agar Sangre (BAPs), siguiendo el protocolo de do Vale Pereira et al. (2017). Los aislados se sembraron BAPs e incubaron a 28ºC por 24 h. Se evaluó el potencial probiótico de los aislados mediante los signos de hemólisis beta, hemólisis alfa, y hemólisis gamma.
Se determinó la actividad antagónica frente a cinco patógenos de peces previamente aislados: Aeromonas hydrophila, Aeromonas veronii, Pseudomonas aeruginosa, Pseudomonas putida y Plesiomonas shigelloides, utilizando el método de difusión en agar adaptado de Balcázar et al. (2008). Las bacterias patógenas se subcultivaron en Caldo Tripticasa de Soya (TSB) y se sembraron 100 μl de cada bacteria patógena en placas Petri con medio agar Nutritivo. Posteriormente, se hicieron pocillos en la superficie del agar con ayuda de una pipeta Pasteur estéril. Se agregaron 30 μl (5 x 108 unidades formadoras de colonia, UFC) de los aislados potencialmente probióticos en los respectivos pocillos. Finalmente, se incubaron a 28 ºC durante 24 h. Se calculó el diámetro de las zonas de inhibición en milímetros (mm) considerándose positivas a las cepas que mostraron una zona clara de inhibición.
La prueba de sensibilidad a los antibióticos se realizó en agar Mueller Hilton (MHA) utilizando el método de difusión en disco de Kirby-Bauer (Bauer et al., 1966). Se utilizaron cinco antibióticos (discos de sensibilidad): cloranfenicol 30μg, amoxicilina/ácido clavulánico 30 μg, gentamicina 2μg, eritromicina 15μg, tetraciclina 30μg y trimetoprima/sulfametoxazol 25μg. Luego se sembraron 100 μl de 108 cfu/ml en placas Petri con agar MHA, se colocaron los discos de sensibilidad en el medio de cultivo y las placas se incubaron a 28ºC durante 24 horas. Se midió la zona de inhibición en milímetros y se calificó como sensible (>20 mm) o resistente (< 20mm) a los antibióticos.
Los ensayos se realizaron para evaluar la capacidad de producción de enzimas proteolíticas y amilolíticas. Se evaluó la actividad proteolítica sobre agar Skim Milk (10%) utilizándola como fuente de proteína. Se sembraron 30 μl de cada bacteria potencialmente probiótica en sus respectivos pocillos y se incubaron a 28ºC durante 24 horas. Los aislados con actividades proteolíticas mostraron zonas transparentes alrededor.
En cuanto a la actividad amilolítica, se sembraron 30 μl de cada bacteria en agar Starch (2.5%) y se incubaron a 28ºC durante 24 horas. Posteriormente se cubrió el crecimiento bacteriano con Lugol al 1% y se observó la producción de amilasa como una zona transparente alrededor (Reda et al., 2017).
Se extrajo el ADN total de las bacterias analizadas utilizando el método CTAB y se procedió a realizar la amplificación del gen ARNr 16S utilizando los cebadores universales F518 (Forward 5'- CCAGCAGCCGCGGTAATACG-3') y 16S R800 (Reverse 5'-TACCAGGGTATCTAATCC-3'). Para la Reacción en Cadena de la Polimerasa (PCR) se utilizaron las siguientes condiciones: un ciclo a 94 ºC durante 5 min, seguido de 35 ciclos a 94ºC durante 30 s, 58ºC durante 45 s, 7ºC durante 1 min y 30 s, y un ciclo final de 72ºC durante 6 min. Se verificaron los productos de PCR en un gel al 1.5% de agarosa teñido con bromuro de etidio. Los productos amplificados se enviaron a Macrogen (USA) para su respectivo secuenciamiento. Las secuencias obtenidas se analizaron con las secuencias homólogas más cercanas del GenBank utilizando el programa BLAST.
RESULTADOS
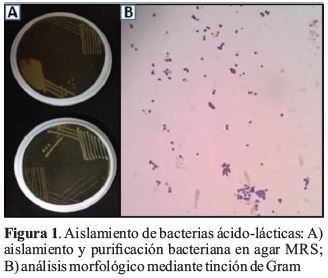
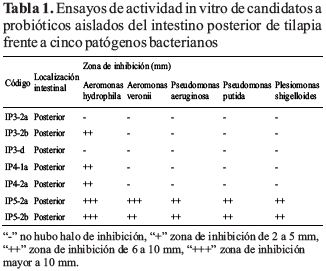
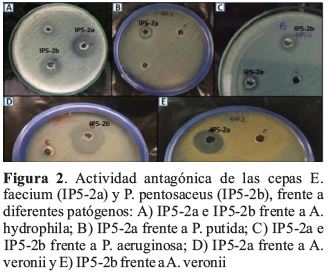
Se aislaron siete bacterias correspondientes a la región posterior (Figura 1). Se consideraron bacterias con actividad antagónica a aquellas que mostraron actividad frente a A. hydrophila, A. veronii, P. aeruginosa, P. putida o P. shigelloides. Cinco de las siete bacterias aisladas incluyendo Enterococcus faecium (IP5-2a) y Pediococcus pentosaceus (IP5-2b) inhibieron el crecimiento de al menos una cepa patógena. Cinco aislados inhibieron el crecimiento de A. hydrophila; además, E. faecium (IP5-2a) y P. pentosaceus (IP5-2b) inhibieron el crecimiento de A. veronii, P. aeruginosa, P. putida y P. shigelloides (Figura 2) con halos de inhibición mayores a 10 mm frente a A. hydrophila y A. veronii, mostrando las mejores actividades antagónicas (Tabla 1).
El ensayo reveló que los aislados IP3-2a e IP3-d mostraron actividad hemolítica debido a la formación de zonas claras alrededor de sus colonias (β-hemólisis) sugiriendo su potencial patogenicidad, por lo que fueron descartados.
Las bacterias E. faecium (IP5-2a) y P. pentosaceus (IP5-2b) mostraron fuerte actividad proteolítica evidenciadas en la formación de zonas claras alrededor de los pocillos. En cuanto a la actividad amilolítica, los mismos aislados fueron capaces de hidrolizar el agar almidón.
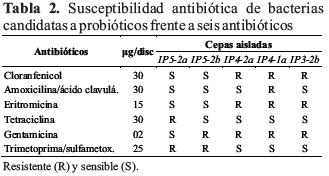
Los resultados del ensayo de susceptibilidad antibiótica se describieron en la Tabla 2.
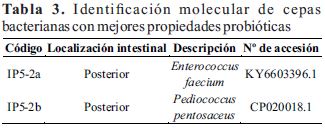
El secuenciamiento del gen ARNr 16S de los aislados bacterianos que exhibieron las mejores propiedades probióticas mostró que IP5-2a e IP5-2b poseen alta similitud de secuencias con Enterococcus faecium (99%) y Pediococcus pentosaceus (100%) (Tabla 3).
DISCUSIÓN
Los animales acuáticos están continuamente expuestos a una amplia variedad de microorganismos que pueden llevarlos a la presentación de enfermedades. Sin embargo, varias alternativas han sido consideradas para contrarrestar dichas enfermedades, siendo una de ellas los antibióticos (Hai, 2015). En la presente investigación se aislaron bacterias acido-lácticas (BAL) del intestino de tilapia y se evaluaron sus actividades antagónicas, carencia de actividad hemolítica, resistencia a los antibióticos, y actividades proteolíticas y amilolíticas.
La presente investigación mostró que dos bacterias identificadas como Enterococcus faecium y Pediococcus pentosaceus fueron capaces de inhibir el crecimiento tanto de P. aeruginosa, P. putida y A. hydrophila. Estudios realizados por Reda et al. (2017) mostraron que ocho de diez bacterias aisladas del intestino de tilapia tuvieron actividad antagónica frente a A. hydrophila. Otras investigaciones similares demuestran la capacidad de aislados bacterianos de inhibir el crecimiento tanto de A. hydrophila y P. aeruginosa (Aly et al., 2008; Vijayabaskar & Somasundaram, 2008; Chemlal-Kherraz et al., 2012; del'Duca et al., 2013; Kaktcham et al., 2017). Además, estudios en otras especies como Arapaima gigas, Oncorhynchus mykiss, Mystus vittatus y Common carp revelaron actividad antagónica común de aislados bacterianos frente a A. hydrophila, una bacteria Gram negativa (Castillo et al., 2017; do Vale Pereira et al., 2017; Kaktcham et al., 2017; Nandi et al., 2017); sin embargo, un solitario estudio en salmónidos reportó que las BAL tienen mayor actividad antagónica frente a bacterias patógenas Gram positivas que a Gram negativas (Ringo, 2008). Por otro lado, algunas BAL son capaces de producir sustancias con efectos bactericidas o bacteriostáticos como bacteriocinas, peróxido de hidrógeno, proteasas, indol, entre otras, las cuales inhiben el crecimiento de bacterias Gram negativas (De B et al., 2014; Zorriehzahra et al., 2016), lo cual podría explicar la capacidad antagónica de los aislados en el presente estudio, además, de acuerdo con Banerjee y Ray (2017) la actividad antagónica es la propiedad más importante de una bacteria potencialmente probiótica.
Otra característica importante que se debe tener en cuenta para determinar bacterias probióticas es la ausencia de actividad hemolítica, propiedad presente en algunas bacterias patógenas potenciales. Dicha actividad está determinada por la producción de hemolisina, un factor de virulencia que produce lisis de los eritrocitos. Las cepas hemolíticas son causantes frecuentes de anemia y edema, motivo por el cual es preferible no seleccionarlas (Nandi et al., 2016). En este estudio, como en el referido por do Vale Pereira et al. (2017), se descartaron las bacterias productoras de hemolisina debido a su potencial patogenicidad.
Aunque la resistencia a los antibióticos es una característica no deseada debido a la presencia de genes de resistencia transmisibles, existe sin embargo la resistencia intrínseca, la cual no se transmite y además podría ayudar en restaurar la microbiota benéfica luego de un tratamiento con antibióticos (Reda et al., 2017). Los hallazgos en este estudio fueron similares a los expuestos por Vlková et al. (2006) en donde se evidencia que las BAL aisladas mostraron resistencia a los antibióticos de la familia de aminoglucósidos y a antibióticos de espectro frente a Gran negativas. De manera similar, existe otro estudio en donde se concluye que la mayoría de bacterias aisladas e identificadas como Enterococcus faecium fueron resistentes a los aminoglucósidos y otros antibióticos utilizados contra bacterias Gram negativas (Termerman et al., 2003). Por lo tanto, la resistencia a estos antibióticos es de carácter intrínseco, por mutaciones, es decir, no hay transferencia de estos genes de resistencia a otras bacterias de la microbiota (Ammor et al., 2007).
La evaluación de actividades proteolíticas como amilolíticas es importante sobre todo en el campo de la nutrición. Ray et al. (2012) mencionan que la microbiota de los peces cumple una función relevante en la digestión debido principalmente a la producción de enzimas. El presente estudio, como en el referido por Nandi et al. (2016), se lograron confirmar las habilidades para la producción de proteasas y amilasas extracelulares, sugiriendo su capacidad de producción enzimática.
Las bacterias que exhibieron las mejores propiedades probióticas fueron identificadas como Enterococcus faecium (IP5-2a) y Pediococcus pentosaceus (IP5-2b). De manera similar, Lara-Flores & Olvera-Novoa (2013) lograron aislar e identificar E. faecium a partir del intestino de tilapia, demostrando en estudios in vivo que su adición en dietas mejoró la ganancia de peso y crecimiento de tilapias. Tanto E. faecium como P. pentosaceus fueron aislados de diferentes especies de agua dulce como Oncorhynchus mykiss, Arapaima gigas y Zacco koreanus (Bajpai et al., 2016; Abedian et al., 2017; do Vale Pereira et al., 2017;). Existen reportes que muestran diferentes BAL con propiedades probióticas aisladas a partir del intestino de tilapia como: Lactococcus sp. y Lactobacillus sp. (Aly et al., 2008; Chemlal-Kherraz et al., 2012; Kaktcham et al., 2017). De acuerdo con Petersen & Dalsgaard (2003), la especie E. faecium fue la más predominante aislada del intestino de peces en granjas integradas, considerándose como parte de la microbiota normal, además, un estudio basado en análisis metagenómico de la microbiota intestinal de peces mostró que el filum Firmicutes, el cual incluye tanto a E. faecium como a P. pentosaceus, es uno de los más dominantes, representando cerca del 90% de las comunidades (Tarnecki et al., 2017). Se ha demostrado también el efecto probiótico in vivo de P. pentosaceus sobre la composición corporal y la microbiota intestinal en Acipenser baerii (Moslehi et al., 2016), además, Martinez et al. (2017) demostraron que P. pentosaceus aislado a partir de O. mykiss fue capaz de absorber y degradar aflatoxinas causantes de enfermedades en la acuicultura. Sin embargo, hasta el desarrollo del escrito no se encontró reportes sobre P. pentosaceus con propiedades probióticas demostradas in vitro aisladas a partir del intestino de tilapia Nilotica.
CONCLUSIÓN
Se lograron aislar siete bacterias nativas del tracto gastrointestinal posterior de Oreochromis niloticus, las cuales fueron sometidas a pruebas para evaluar sus propiedades probióticas como actividad antagónica, actividad hemolítica, actividades proteolíticas y amilolíticas, y sensibilidad a los antibióticos. Las bacterias que presentaron mejores características fueron identificadas molecularmente como Enterococcus faecium y Pediococcus pentosaceus. Sin embargo, aunque el presente estudio mostró dos buenos candidatos a probióticos, se recomienda una confirmación mediante estudios in vivo para evaluar su capacidad de colonización gastrointestinal y el mejoramiento de la salud e inmunidad.
AGRADECIMIENTOS
El presente trabajo de investigación fue financiado por Cienciactiva-Concytec, y desarrollado a través del proyecto "Círculo de investigación en biotecnología molecular para el desarrollo y la sostenibilidad de los sectores acuícolas del Perú", código: 132-2015.
Los autores agradecen a la empresa Acuicultura de Huaura S.A.C por brindar el material biológico.
REFERENCIAS BIBLIOGRÁFICAS
Abedian Amiri, A., Azari Takami, G., Afsharnasab, M., & Razavilar, V. (2017). The comparative effects of dietary supplementation with Pediococcus acidilactici and Enterococcus faecium on feed utilization, various health-related characteristics and yersiniosis in rainbow trout (Oncorhynchus mykiss Walbaum, 1792). Iranian Journal of Fisheries Sciences, 16(2), 753-773. http://jifro.ir/article-1-2747-en.html. [ Links ]
Akhter, N., Wu, B., Memon, A. M., & Mohsin, M. (2015). Probiotics and prebiotics associated with aquaculture: a review. Fish & shellfish immunology, 45(2), 733-741. doi: 10.1016/j.fsi.2015.05.038. [ Links ]
Aly, S. M., Mohamed, M. F., & John, G. (2008). Effect of probiotics on the survival, growth and challenge infection in Tilapia nilotica (Oreochromis niloticus). Aquaculture research, 39(6), 647-656. doi: 10.1111/j.1365- 2109.2008.01932.x. [ Links ]
Ammor, M. S., Flórez, A. B., & Mayo, B. (2007). Antibiotic resistance in non-enterococcal lactic acid bacteria and bifidobacteria. Food Microbiology, 24(6), 559-570. doi: 10.1016/j.fm.2006.11.001. [ Links ]
Argota, P.G. & Iannacone, O.J. (2017). Comportamiento de refugio y actividad de la acetilcolinesterasa cerebral en Gambusia punctata (Poey, 1854) (Poeciliidae) por plomo biodisponible. Revista The Biologist; 17(1), 141-153. https://www.researchgate.net/publication/323675051 [ Links ]
Asencios, Y. O., Sánchez, F. B., Mendizábal, H. B., Pusari, K. H., Alfonso, H. O., Sayán, A. M., & Chaupe, N. S. (2016). First report of Streptococcus agalactiae isolated from Oreochromis niloticus in Piura, Peru: Molecular identification and histopathological lesions. Aquaculture Reports, 4, 74-79. doi: 10.1016/j.aqrep.2016.06.002. [ Links ]
Bajpai, V. K., Jeong-Ho, H., Rather, I. A., Majumder, R., Nam, G. J., ChanSeo, P., & Yong-Ha, P. (2016). Characterization of lactic acid bacterium Pediococcus pentosaceus 4I1 from fresh water fish Zacco koreanus and its antibacterial mode of action. PeerJ PrePrints. doi: 10.7287/peerj.preprints.2121v1.
Balcázar, J. L., Vendrell, D., de Blas, I., Ruiz-Zarzuela, I., Muzquiz, J. L., & Girones, O. (2008). Characterization of probiotic properties of lactic acid bacteria isolated from intestinal microbiota of fish. Aquaculture, 278(1-4), 188-191. doi: 10.1016/j.aquaculture.2008.03.014. [ Links ]
Banerjee, G., & Ray, A. K. (2017). The advancement of probiotics research and its application in fish farming industries. Research in veterinary science, 115, 66-77. doi: 10.1016/j.rvsc.2017.01.016. [ Links ]
Bauer, A. W., Kirby, W. M., Sherris, J. C., & Turck, M. (1966). Antibiotic susceptibility testing by a standardized single disk method. American journal of clinical pathology, 45(4), 493. https://www.ncbi.nlm.nih.gov/pubmed/5325707. [ Links ]
Bidhan D. C., Meena, D. K., Behera, B. K., Das, P., Mohapatra, P. D., & Sharma, A. P. (2014). Probiotics in fish and shellfish culture: immunomodulatory and ecophysiological responses. Fish physiology and biochemistry, 40(3), 921- 971. doi: 0.1007/s10695-013-9897-0. [ Links ]
Burridge, L., Weis, J. S., Cabello, F., Pizarro, J., & Bostick, K. (2010). Chemical use in salmon aquaculture: a review of current practices and possible environmental effects. Aquaculture, 306(1-4), 7-23. doi: 10.1016/j.aquaculture.2010.05.020. [ Links ]
Cabello, F. C., Godfrey, H. P., Tomova, A., Ivanova, L., Dölz, H., Millanao, A., & Buschmann, A. H. (2013). Antimicrobial use in aquaculture re‐examined: its relevance to antimicrobial resistance and to animal and human health. Environmental microbiology, 15(7), 1917-1942. doi: 10.1111/1462-2920.12134. [ Links ]
Castillo, D. 2017. Caracterización molecular de la microbiota intestinal de alevines de paiche Arapaima gigas y selección de cepas potencialmente probióticas. Tesis de maestría, Universidad Nacional de Tumbes, Tumbes. Perú. 38 pp. [ Links ]
Chemlal-Kherraz, D., Sahnouni, F., Matallah-Boutiba, A., & Boutiba, Z. (2012). The probiotic potential of lactobacilli isolated from Nile tilapia (Oreochromis niloticus)'s intestine. African Journal of Biotechnology, 11(68), 13220-13227. doi: 10.5897/AJB12.748. [ Links ]
Del'Duca, A., Cesar, D. E., Diniz, C. G., & Abreu, P. C. (2013). Evaluation of the presence and efficiency of potential probiotic bacteria in the gut of tilapia (Oreochromis niloticus) using the fluorescent in situ hybridization technique. Aquaculture, 388, 115-121. doi: 10.1016/j.aquaculture.2013.01.019. [ Links ]
do Vale Pereira, G., da Cunha, D. G., Pedreira Mourino, J. L., Rodiles, A., Jaramillo‐Torres, A., & Merrifield, D. L. (2017). Characterization of microbiota in Arapaima gigas intestine and isolation of potential probiotic bacteria. Journal of applied microbiology, 123(5), 1298-1311. doi: 10.1111/jam.13572. [ Links ]
FAO (2018). El estado mundial de la pesca y la acuicultura. http://www.fao.org/3/I9540EN/i9540en.pdf. [ Links ]
Hai, N. V. (2015). The use of probiotics in aquaculture. Journal of applied microbiology, 119(4), 917-935. doi: 10.1111/jam.12886. [ Links ]
Huicab-Pech, Z. G., Castañeda-Chávez, M. R., & Lango-Reynoso, F. (2017). Pathogenic Bacteria in Oreochromis Niloticus Var. Stirling Tilapia Culture. Fish Aqua J, 8:197. doi: 10.4172/2150-3508.1000197.
Huicab-Pech, Z. G., Landeros-Sánchez, C., Castañeda-Chávez, M. R., Lango-Reynoso, F., López-Collado, C. J., & Platas Rosado, D. E. (2016). Current State of Bacteria Pathogenicity and their Relationship with Host and Environment in Tilapia Oreochromis niloticus. J Aquac Res Development, 7(5). doi: 10.4172/2155-9546.1000428.
Kaktcham, P. M., Temgoua, J. B., Zambou, F. N., Diaz-Ruiz, G., Wacher, C., & de Lourdes Pérez-Chabela, M. (2018). In Vitro Evaluation of the Probiotic and Safety Properties of Bacteriocinogenic and Non-Bacteriocinogenic Lactic Acid Bacteria from the Intestines of Nile Tilapia and Common Carp for Their Use as Probiotics in Aquaculture. Probiotics and antimicrobial proteins, 10(1), 98-109. doi: 10.1007/s12602-017-9312-8. [ Links ]
Lara-Flores, M., & Olvera-Novoa, M. A. (2013). The use of lactic acid bacteria isolated from intestinal tract of Nile tilapia (Oreochromis niloticus), as growth promoters in fish fed low protein diets. Latin American Journal of Aquatic Research, 41(3). doi: 103856/vol41-issue3-fulltext-12.
Lazado, C. C., & Caipang, C. M. A. (2014). Mucosal immunity and probiotics in fish. Fish & shellfish immunology, 39(1), 78-89. doi: 10.1016/j.fsi.2014.04.015. [ Links ]
Lazado, C. C., Caipang, C. M. A., Brinchmann, M. F., & Kiron, V. (2011). In vitro adherence of two candidate probiotics from Atlantic cod and their interference with the adhesion of two pathogenic bacteria. Veterinary microbiology, 148(2-4), 252-259. doi: 10.1016/j.vetmic.2010.08.024. [ Links ]
Martínez Cruz, P., Ibáñez, A. L., Monroy Hermosillo, O. A., & Ramírez Saad, H. C. (2012). Use of probiotics in aquaculture. ISRN microbiology, 2012. doi: 10.5402/2012/916845.
Martinez, M. P., Gonzalez Pereyra, M. L., Pena, G. A., Poloni, V., Fernandez Juri, G., & Cavaglieri, L. R. (2017). Pediococcus acidolactici and Pediococcus pentosaceus isolated from a rainbow trout ecosystem have probiotic and ABF1 adsorbing/degrading abilities in vitro. Food Additives & Contaminants: Part A, 34(12), 2118-2130. doi: 10.1080/19440049.2017.1371854 [ Links ]
Modi, S. R., Collins, J. J., & Relman, D. A. (2014). Antibiotics and the gut microbiota. The Journal of clinical investigation, 124(10), 4212-4218. doi: 10.1172/JCI72333. [ Links ]
Mohapatra, S., Chakraborty, T., Prusty, A. K., PaniPrasad, K., & Mohanta, K. N. (2014). Beneficial effects of dietary probiotics mixture on hemato-immunology and cell apoptosis of Labeo rohita fingerlings reared at higher water temperatures. PloS one, 9(6), e100929. doi: 10.1371/journal.pone.0100929. [ Links ]
Moslehi, F., Sattari, M., & Masouleh, A. S. (2016). Effects of Pediococcus pentosaceus as a probiotic on intestinal microbiota and body composition of Siberian sturgeon, Acipenser baerii Brandt, 1869. International Journal of Aquatic Biology, 4(1), 11-16. [ Links ]
Nandi, A., Dan, S. K., Banerjee, G., Ghosh, P., Ghosh, K., Ringø, E., & Ray, A. K. (2017). Probiotic potential of autochthonous bacteria isolated from the gastrointestinal tract of four freshwater teleosts. Probiotics and antimicrobial proteins, 9(1), 12-21. doi: 10.1007/s12602-016-9228-8. [ Links ]
Peso-Echarri, P., Frontela-Saseta, C., González-Bermúdez, C. A., Ros-Berruezo, G. F., & Martínez-Graciá, C. (2012). Polisacáridos de algas como ingredientes funcionales en acuicultura marina: alginato, carragenato y ulvano. Revista de biología marina y oceanografía, 47(3), 373-381. doi: 10.4067/S0718-19572012000300001. [ Links ]
Petersen, A., & Dalsgaard, A. (2003). Species composition and antimicrobial resistance genes of Enterococcus spp., isolated from integrated and traditional fish farms in Thailand. Environmental Microbiology, 5(5), 395-402. doi: https://www.ncbi.nlm.nih.gov/pubmed/12713465. [ Links ]
Pirarat, N., Pinpimai, K., Endo, M., Katagiri, T., Ponpornpisit, A., Chansue, N., & Maita, M. (2011). Modulation of intestinal morphology and immunity in nile tilapia (Oreochromis niloticus) by Lactobacillus rhamnosus GG. Research in veterinary science, 91(3), e92-e97. doi: 10.1016/j.rvsc.2011.02.014. [ Links ]
Ray, A. K., Ghosh, K., & Ringø, E. (2012). Enzyme‐producing bacteria isolated from fish gut: a review. Aquaculture Nutrition, 18(5), 465-492. doi: 10.1111/j.1365-2095.2012.00943.x [ Links ]
Reda, R. M., Selim, K. M., El-Sayed, H. M., & El-Hady, M. A. (2017). In Vitro Selection and Identification of Potential Probiotics Isolated from the Gastrointestinal Tract of Nile Tilapia, Oreochromis niloticus. Probiotics and antimicrobial proteins, 1-12. doi: 10.1007/s12602-017-9314-6.
Ringo E. (2008). The ability of carnobacteria isolated from fish intestine to inhibit growth of fish pathogenic bacteria: a screening study. Aquaculture Research, 39, 171-180. doi: 10.1111/j.1365-2109.2007.01876.x [ Links ]
Rodgers, C. J., & Furones, M. D. (2009). Antimicrobial agents in aquaculture: practice, needs and issues. Options Méditerranéennes, 86, 41-59. doi: 10.13140/2.1.4697.0560. [ Links ]
Romero, J., Feijoó, C. G., & Navarrete, P. (2012). Antibiotics in aquaculture–use, abuse and alternatives. In Health and environment in aquaculture. InTech. doi: 10.5772/28157.
Sihag, R. C., & Sharma, P. (2012). Probiotics: the new ecofriendly alternative measures of disease control for sustainable aquaculture. Journal of Fisheries and Aquatic Science, 7(2), 72. doi: 10.3923/jfas.2012.72.103. [ Links ]
Tarnecki, A. M., Burgos, F. A., Ray, C. L., & Arias, C. R. (2017). Fish intestinal microbiome: diversity and symbiosis unraveled by metagenomics. Journal of applied microbiology. doi: 10.1111/jam.13415.
Temmerman, R., Pot, B., Huys, G., & Swings, J. (2003). Identification and antibiotic susceptibility of bacterial isolates from probiotic products. International Journal of Food Microbiology, 81(1), 1-10. https://www.ncbi.nlm.nih.gov/pubmed/12423913. [ Links ]
Vidal, L. V. O., Albinati, R. C. B., Albinati, A. C. L., Lira, A. D. D., Almeida, T. R. D., & Santos, G. B. (2008). Eugenol as an anesthetic for Nile tilapia. Pesquisa Agropecuária Brasileira, 43(8), 1069-1074. doi: 10.1590/S0100- 204X2008000800017. [ Links ]
Vijayabaskar, P., & Somasundaram, S. T. (2008). Isolation of bacteriocin producing lactic acid bacteria from fish gut and probiotic activity against common fresh water fish pathogen Aeromonas hydrophila. Biotechnology, 7(1), 124-128. doi: 10.3923/biotech.2008.124.128. [ Links ]
Vlková, E., Rada, V., Popelářová, P., Trojanová, I., & Killer, J. (2006). Antimicrobial susceptibility of bifidobacteria isolated from gastrointestinal tract of calves. Livestock Science, 105(1), 253-259. doi: 10.1016/j.livsci.2006.04.011. [ Links ]
Wang, M., & Lu, M. (2016). Tilapia polyculture: a global review. Aquaculture research, 47(8), 2363-2374. doi: 10.1111/are.12708. [ Links ]
Yang, H. L., Xia, H. Q., Ye, Y. D., Zou, W. C., & Sun, Y. Z. (2014). Probiotic Bacillus pumilus SE5 shapes the intestinal microbiota and mucosal immunity in grouper Epinephelus coioides. Diseases of aquatic organisms, 111(2), 119-127. doi: 10.3354/dao02772. [ Links ]
Zorriehzahra, M. J., Delshad, S. T., Adel, M., Tiwari, R., Karthik, K., Dhama, K., & Lazado, C. C. (2016). Probiotics as beneficial microbes in aquaculture: an update on their multiple modes of action: a review. Veterinary Quarterly, 36(4), 228-241. doi: 10.1080/01652176.2016.1172132. [ Links ]
Artículo recibido: 29-05-2018
Artículo aceptado: 09-09-2018
On line: 29-10-2018