INTRODUCCIÓN
El petróleo crudo, comúnmente llamado combustible fósil, comprende una mezcla de compuestos hidrocarburados y no hidrocarburados. La primera fracción, que corresponde a los hidrocarburados está formada por componentes alifáticos saturados, aromáticos, asfaltenos, ceras y alquitranes (Moubasher et al., 2015; Varjani & Upasani, 2017); y la segunda fracción que contiene a los no hidrocarburados, consiste en compuestos de azufre, oxígeno, nitrógeno (Ossai et al., 2020) y metales (Costa et al., 2012).
El uso del petróleo y sus derivados en el mundo contemporáneo se ha intensificado rápidamente, volviéndose imprescindible y ocupando un rol crucial en el desarrollo de la economía mundial con una demanda que va en ascenso; sin embargo, esta situación contribuye con la contaminación ambiental debido a incidentes durante el proceso de extracción, transporte y/o almacenamiento como los derrames accidentales de crudo de petróleo (Behesht et al., 2008; Mahjoubi et al., 2013; Ossai et al., 2020) e incluso con el uso del mismo en la vida cotidiana.
La presencia de hidrocarburos en ambientes tanto de suelo como agua, representan un peligro sustancial para el ser humano y otras formas de vida presentes en los ambientes contaminados (Aguilera et al., 2010; Hentati et al., 2013; Hreniuc et al., 2015; Sammarco et al., 2016). En el agua forman películas superficiales y otras fracciones moleculares superiores que se hunden (Mishra & Kumar, 2015). Posteriormente, la erosión de sedimentos costeros distribuye los hidrocarburos en toda la columna de agua agrediendo a la vida marina. (Al-Majed et al., 2012). En el suelo, los hidrocarburos de bajo peso molecular se volatilizan y el resto penetra verticalmente hasta las aguas subterráneas donde son extendidos lateralmente (Ossai et al., 2020).
Uno de los componentes fundamentales de los ecosistemas son las comunidades microbianas que desempeñan roles importantes en el metabolismo de la materia orgánica e intervienen en la degradación de una amplia gama de contaminantes (Brennerova et al., 2009) como los hidrocarburos de petróleo, siendo Proteobacteria y Actinobacteria los filos bacterianos que agrupan especies con genes codificantes de enzimas necesarias para el metabolismo de hidrocarburos (Gibson & Parales, 2000), primordialmente oxigenasas y peroxidasas que inician las vías de degradación (Ossai et al., 2020).
De esta manera, los microorganismos nativos de las zonas contaminadas aportan una alternativa eco-amigable para la descontaminación de hidrocarburos de petróleo: la biorremediación (Mahjoubi et al., 2013), que es una opción factible a nivel mundial (Van Stempvoort & Biggar, 2008) por su facilidad de aplicación, ecosostenibilidad y rentabilidad (Ossai et al., 2020). Algunos de los géneros bacterianos más estudiados para biorremediación son Pseudomonas, Bacillus, Acinetobacter, Serratia y Proteus (Baruah et al., 2017; Chebbi et al., 2017; Nalini & Parthasarathi, 2013; Pacwa-Płociniczak et al., 2016), debido a que cuentan con la capacidad de producir biosurfactantes que aumentan la biodisponibilidad de los hidrocarburos en el suelo o agua para su degradación (Pacwa-Płociniczak et al., 2016).
Para aplicar esta tecnología es importante comprender del todo los mecanismos de interacción entre la comunidad microbiana, el ambiente y el contaminante (Ossai et al., 2020); por ello es que generalmente se emplea el análisis metagenómico, que permite comprender las propiedades genómicas y rasgos funcionales de las comunidades a partir del ADN ambiental extraído (Bao et al., 2017).
El propósito de este trabajo fue aislar e identificar cepas bacterianas con potencial biorremediador a partir de agua y suelo amazónicos contaminados con petróleo y determinar sus comunidades bacterianas por metagenómica.
MATERIAL Y MÉTODOS
Muestreo
Las muestras de agua y suelo contaminadas con petróleo crudo fueron colectadas de la capa superficial (0 - 15 cm) a partir del derrame de petróleo de agosto del 2016 a la altura del km 364 +900 de la Quebrada de Uchichiangos en el tramo II del oleoducto norperuano en el distrito de Nieva, provincia de Condorcanqui, departamento de Amazonas. Empleando un muestreo por conveniencia, se recolectó una muestra de 1 Kg de suelo (dividido en 3 submuestras) y una de 1 l de agua (dividido en 3 submuestras) almacenadas en condiciones de esterilidad a 4 °C y cubiertas de la luz hasta su llegada a las instalaciones del laboratorio de la empresa IncaBiotec, ubicado en el departamento de Tumbes para los análisis microbiológicos y genómicos posteriores.
Aislamiento e identificación de cepas bacterianas
El aislamiento de las cepas se realizó siguiendo el protocolo modificado de Tuo et al. (2012) en medios de cultivo microbiológico suplementados con crudo de petróleo y por triplicado. A las muestras de suelo se les realizó un pretratamiento: se suspendieron 10 g de muestra en 100 ml de solución salina fisiológica (SSF) y se mantuvo en agitación constante por un día a 180 revoluciones por minuto (rpm) para extraer el sobrenadante con las baterías resuspendidas.
El pre-enriquecimiento consistió en sembrar 10 ml de agua o sobrenadante (en el caso de muestras de suelo) en 90 ml de caldo Luria Bertani (LB) suplementado con 1 ml de petróleo crudo estéril de la zona del derrame. Se mantuvo en agitación constante de 180 rpm por 5 días y posteriormente se inoculó el sobrenadante en caldo de Medio Mínimo Salino (MSM) suplementado con crudo de petróleo al 1 % y se mantuvo la agitación constante a 180 rpm por 48 horas. Subsecuentemente, se inocularon por esparcimiento las diluciones 10-9 y 10-12 de cada cultivo en placas de Petri con agar MSM suplementado con 100 ul de petróleo crudo para la selección y purificación de cepas bacterianas. Así, se aislaron preferentemente bacterias Gram negativas, debido a que, generalmente, éstas son capaces de degradar diferentes tipos de hidrocarburos (Bisht et al., 2015).
Para la identificación de las cepas aisladas se extrajo el ADN bacteriano empleando el método CTAB 2X; luego, se amplificó parcialmente el gen ARNr 16S por Reacción en Cadena de la Polimerasa (PCR) con los iniciadores universales de Lane (1991) 27F Forward AGAGTTTGATCMTGGCTCAG y 1492R Reverse TACGGYTACCTTGTTACGACTT, verificando la calidad de los productos amplificados por electroforesis en horizontal, en gel de agarosa al 1 % teñido con bromuro de etidio. Después, los productos amplificados fueron secuenciados por el método de Sanger. A partir de las secuencias forward y reverse se obtuvieron las secuencias consenso correspondientes en el programa DNA Dragon y, finalmente estas últimas se identificaron por comparación con la base de datos del servidor BLASTN de NCBI.
Extracción de ADN metagenómico
La extracción del ADN metagenómico se realizó con el kit PowerSoil DNA Isolation (Mo Bio Laboratories, EE.UU.) a partir de las muestras de suelo y del sedimento obtenido por centrifugación de las muestras de agua, siguiendo las instrucciones del fabricante. El ADN extraído fue secuenciado mediante la tecnología Ilumina, dirigido a las regiones V3 y V4 del gen ARNr16S.
Análisis bioinformático
El análisis de datos y asignación taxonómica se llevó a cabo con el software bioinformático Qiime2 (Quantitative Insights Into Microbial Ecology II) versión 2017.4 (Bolyen et al., 2019). Las secuencias fueron filtradas y se eliminaron quimeras con el plugin q2-dada2 mediante el método denoise-paired y luego se agruparon con el complemento feature-table group. Para el análisis de diversidad se usó el plugin q2-diversity con el pipeline core-metrics-phylogenetic. Las categorías taxonómicas asignadas a las secuencias representativas se realizaron usando el pluging q2- feature-classifier classify-sklearn y un clasificador Naive Bayes preparado con la base de datos de referencia GreenGenes 13_8 99 %.
RESULTADOS
Identificación de cepas bacterianas
Se aislaron siete cepas bacterianas a partir de las muestras de agua y seis de las muestras de suelo. La identificación por caracterización del gen ARNr 16S mostró que pertenecen a los géneros Klebsiella, Enteobacter, Pseudomonas, Serratia, Acinetobacter, Proteus y Morganella (Tabla 1).
Tabla 1 Identificación molecular de las cepas bacterianas aisladas.
| Muestra | Código | Descripción | N° de Accesión |
| A1 | Klebsiella oxytoca | NR_112010.1 | |
| A2 | Enterobacter cloacae | NR_102794.2 | |
| Agua | A3 | Pseudomonas koreensis | NR_025228.1 |
| Contaminada | A4 | Serratia marcescens | NR_036886.1 |
| A5 | Pseudomonas prosekii | NR_132724.1 | |
| A6 | Acinetobacter rudis | NR_115988.1 | |
| A7 | Proteus vulgaris | NR_115878.1 | |
| S1 | Pseudomonas moraviensis | NR_043314.1 | |
| S2 | Proteus hauseri | NR_104767.1 | |
| Suelo | S3 | Proteus vulgaris | NR_115878.1 |
| contaminado | S4 | Proteus terrae | NR_146019.1 |
| S5 | Morganella morganii | NR_113580.1 | |
| S6 | Serratia marcescens | NR_036886.1 |
Análisis metagenómico
La diversidad alfa de las comunidades bacterianas de las muestras estudiadas se muestra en la figura 2. En la figura 2A se presentan las curvas de rarefacción, en las que se observa como éstas tienen una pendiente cercana a cero, asegurando de esta manera que, aunque las muestras lleguen a tener una mayor profundidad de análisis de secuencias esto no resultaría en la adición de características (número de OTU’s observados). Por otro lado, las figuras 2B y 1C muestran los índices de diversidad alfa de Shannon y Faith donde las diferencias en los resultados comparativos de ambas muestras no son significativas; para Shannon 8,30 para agua y 7,87 para suelo mientras que en cuanto a Faith, fueron en el mismo orden 36,17 y 37,11.
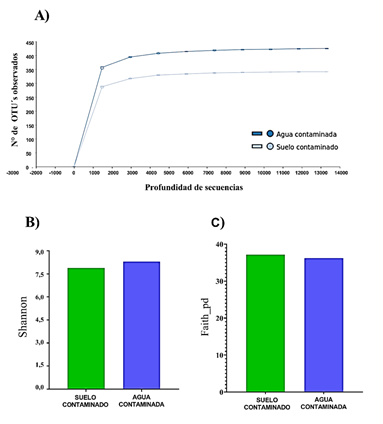
Figura 2 Diversidad alfa 1A) Curvas de rarefacción de OTU’s obervados. 1B) Índice de Shannon. 1C) Índice de diversidad filogenética de Faith.
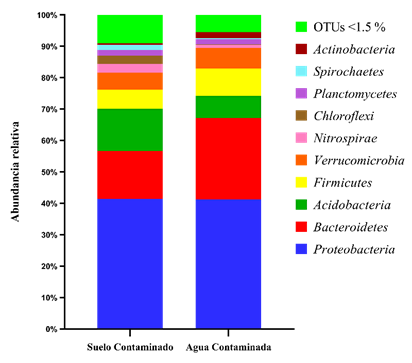
La asignación taxonómica a nivel de filos bacterianos (figura 3) a las muestras agrupadas de suelo contaminado (SC) y agua contaminada (AC) mostró la presencia de 10 de ellos superiores al 1,5 % de abundancia relativa; siendo los más representativos Proteobacteria (41,5 % en SC y 41,3 % en AC), Bacteroidetes (15,3 % en SC y 25,9 % en AC) y Acidobacteria (13,4 % en SC y 7,0 % en AC).
Con respecto a la asignación taxonómica a nivel de familia, el heat-map de la figura 4 muestra las familias más abundantes (con frecuencia de secuencias mayor a 80), siendo las más representativas en muestras de suelo y agua contaminadas Flavobacteriaceae y Moraxellaceae; además, se observa que Verrucomicrobiaceae y Acetobacteraceae que fueron más abundantes en agua y Enterobacteraceae que abundó más en suelo. Otras familias como Pseudomonadaceae y Micrococcaceae fueron detectadas sólo en agua contaminada y Anaerolinaceae y Methylococcaceae sólo en suelo contaminado.

Figura 4 Heat-map de asignación taxonómica a nivel de familia de las muestras de agua y suelo contaminados.
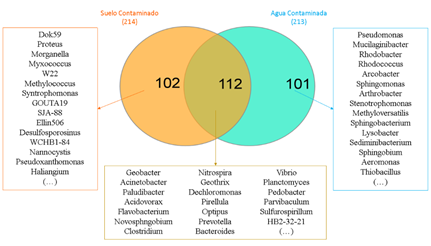
En la figura 5 se muestra el diagrama de Veen-Euler que se realizó con la herramienta de Heberle et al. (2015) y en donde se observa el número de géneros bacterianos compartidos e independientes de cada tipo de muestra. Se destaca entre los 112 géneros compartidos a Acinetobacter y Flavobacterium por ser ampliamente reportados como nativos de zonas contaminadas y con propiedades biorremediadoras de hidrocarburos.
DISCUSIÓN
La biorremediación in situ es considerada el método más eficaz para la biodegradación residual de hidrocarburos de petróleo (Al-Dhabaan, 2019), por ello, es razonable buscar en zonas contaminadas con petróleo bacterias que lo utilicen como fuente de carbono y energía y que presenten potencial biodegradador de este combustible (Al-Dhabaan, 2019; Sarkar et al., 2017).
Pseudomonas spp han sido aisladas de zonas contaminadas con hidrocarburos y se estudia su potencial biorremediador (Allamin et al. 2014; Baruah et al. 2017; Chebbi et al. 2017; Al-Dhabaan 2019; Smułek et al. 2020), mientras que también se ha demostrado la producción de biosurfactantes y moléculas degradadoras de compuestos alifáticos y aromáticos, así como la presencia de los genes alkB relacionados con la alcano hidrolasa en cultivos de Pseudomonas y Enterobacter (Sarkar et al., 2017), géneros bacterianos también aislados en la presente investigación.
En este trabajo también se aislaron cepas del género Serratia, reconocido por utilizar los hidrocarburos como fuente de energía y carbono, además de la producción de biosurfactantes (Nalini & Parthasarathi, 2013; Pacwa-Płociniczak et al., 2016; Smułek et al., 2020); también, Klebsiella spp, degradadoras particularmente de alcanos y cicloalcanos, igualmente productora de biosurfactantes y que puede ser empleada incluso como asistente en fitorremediación (Allamin et al., 2014; Liu et al., 2014); Asimismo, se aislaron cepas pertenecientes a los géneros Acinetobacter, Morganella y Proteus, que cuentan con la capacidad de degradación de hidrocarburos y producción de biosurfactantes o bioemulsificantes tanto en agua como en suelo contaminado con hidrocarburos (Mahjoubi et al., 2013; Pacwa-Płociniczak et al., 2016; Subramanian & Menon, 2015).
El perfil de la comunidad microbiana en ambientes contaminados es uno de los aspectos de mayor importancia para entender la interacción ambiente-contaminante. El índice de Shannon, de diversidad alfa, muestra la riqueza específica por muestra (Swift, M. J., Atlas, R. M., & Bartha, 1982); en este trabajo, la riqueza de especies fue ligeramente más alta en el agua contaminada, quizás porque en el agua la mayor parte de hidrocarburos se quedan en la superficie (Mishra & Kumar, 2015), a diferencia de en los suelos en los que descienden verticalmente (Ossai et al., 2020). El crudo de petróleo contiene una mezcla de hidrocarburos, por lo tanto, tiene varias configuraciones estructurales (Prabhu & Phale, 2003), las cuales mientras más peso molecular tengan, son más hidrófobos (Alegbeleye et al., 2017) lo que impide que se mezclen con facilidad en el agua. El índice de Faith_pd (Faith, 1992) mide la diversidad filogenética de las muestras que existe entre las especies bacterianas y permitió determinar a las muestras de suelo contaminado como las más diversas por una mínima diferencia.
Al igual que en el presente trabajo, otros estudios de análisis metagenómico demuestran la predominancia de los filos bacterianos Proteobacteria y Bacteroidetes como asociados a la degradación de hidrocarburos de petróleo en ambientes acuáticos (Hidalgo et al., 2020; Lee et al., 2018) y terrestres (Bao et al., 2017; Khan et al., 2018; Kostka et al., 2011; Sutton et al., 2013; Zhu et al., 2020) revelando que estos grupos están involucrados en la degradación aerobia y anaerobia de hidrocarburos de petróleo (Zhu et al., 2020).
Así mismo, las familias bacterianas Moraxellaceae (con el género Acinetobacter) y Flavobacteriaceae (con el género Flavobacterium) están relacionados a las funciones de degradación de hidrocarburos (Abbasian, F., Lockington, R., Megharaj, M., Naidu, 2016; Hidalgo et al., 2020; Kostka et al., 2011) y se determinó que participa en el metabolismo de compuestos hidrocarbonados mediante el análisis metagenómico.
Además géneros bacterianos cómo Pseudomonas (Garrido-Sanz et al., 2019; Kostka et al., 2011) y Geobacter (Hidalgo et al., 2020) han proporcionado aplicaciones putativas en biorremediación de hidrocarburos de petróleo mediante el uso de técnicas independientes de cultivo. Estos grupos bacterianos fueron detectados por metagenómica en la presente investigación.
CONCLUSIÓN
Se aislaron 13 cepas bacterianas de los géneros Klebsiella, Enterobacter, Pseudomonas, Serratia, Acinetobacter, Proteus y Morganella, con potencial degradador de hidrocarburos a partir de zonas contaminadas con crudo de petróleo, que pueden ser utilizadas para la biorremediación de estos ambientes. Así mismo el análisis metagenómico demostró que el petróleo influye en la composición de la comunidad bacteriana favoreciendo la dominancia de OTU´s tolerantes a los hidrocarburos de petróleo y que pueden metabolizarlo, agrupados en los filos Proteobacteria, Bacteroidetes y Acidobacteria principalmente.
Además, se demostró la importancia de la metagenómica como herramienta para la investigación de ambientes impactados con hidrocarburos de petróleo, permitiendo un mejor conocimiento de los microorganismos para el aislamiento dirigido a aquellos de interés para la biorremediación.
Finalmente, se sugiere el uso de las cepas aisladas en estudios futuros de biorremediación aplicada.












 uBio
uBio