Introducción
Actualmente, la microbiología predictiva, se ha convertido en una herramienta poderosa para analizar, describir y predecir cambios microbianos producidos en los alimentos. Esta ciencia, mediante la aplicación de modelos matemáticos tales como Gompertz, Logístico, Weibull entre otros, busca explicar los cambios dinámicos en biomasa y metabolitos en función al tiempo en condiciones específicas de sustrato; a su vez muchos factores tienen una influencia importante en la situación de crecimiento de los microorganismos, ya sea por factores intrínsecos (pH, actividad de agua, nutrientes, etc.) o extrínsecos (humedad, luz, gases, etc.). Por las razones descritas se han realizado numerosos estudios que produjeron modelos cinéticos para predecir la situación de crecimiento de los microorganismos, centrando especial atención en el tiempo de retraso, la tasa de crecimiento específica máxima y parámetros cinéticos que pueden ayudar a los investigadores ya sea en el control, eliminación de algunos de ellos o a la producción de alimentos que contengan microorganismos que beneficien al consumidor (Gu, Sun, Tu, Dong, y Pan, 2016).
La leche es uno de los alimentos más consumidos debido a su gran potencial nutricional, entre los cuales se destaca el aporte de minerales esenciales y una importante fuente de calcio biodisponible para el ser humano (Guevara, Montero, Valle, y Avilés, 2019). Asimismo, los beneficios de este alimento no se limitan exclusivamente a su valor nutricional, sino que se extiende más allá ya que puede constituir un factor de prevención de algunas patologías vasculares, algunos tipos de cáncer, hipertensión arterial y patologías óseas o dentales (Fernández et al., 2015). Pese a las bondades antes mencionadas el tiempo de vida útil de este producto es relativamente corto debido a diferentes factores como la degradación de lípidos y contaminación microbiana, es así que se realizan experimentos con adición de probióticos, que demuestran innumerables beneficios en el consumidor: como la regulación bacteriana a lo largo de la cadena digestiva, ya que estas contienen protectores naturales como es el caso de ácidos orgánicos, peróxido de hidrógeno, bacteriocinas y sustancias específicas, como péptidos antivirales o péptidos de bajo peso molecular, asimismo, efectos inhibitorios de ácidos grasos y finalmente el incremento de la vida útil (Pahumunto et al., 2020; Yerlikaya, 2019).
Entre los microrganismos de acción probiótica tenemos a Saccharomyces boulardii este microorganismo puede mantenerse viable y activo de modo que al ser ingerida pueda dar efectos fármaco dinámicos semejantes a los efectos fisiológicos de la flora intestinal normal (Peña, 2007; Vega, Martinez, Montañez, y Rodiles, 2016); esta actúa como un transportador liberando enzimas, proteínas y factores tróficos durante su tránsito interintestinal, mejorando las defensas inmunológicas del huésped, la digestión y la absorción de nutrientes es por eso que el interés en este microorganismo, haciendo que esta levadura se vuelva una fuente importante para la obtención de productos probióticos (Mejía, Montoya, Cortés, y Saavedra, 2016; Sen y Mansell, 2020).
Por tal motivo, el objetivo de esta investigación es evaluar modelos cinéticos sigmoidales aplicados al crecimiento de Saccharomyces boulardii en leche fresca de vaca.
Metodología
Recolección y análisis fisicoquímico
La leche fresca de vaca fue recolectada en la finca ganadera de la Universidad Peruana Unión campus Juliaca, en botellas estériles de 500 ml las cuales fueron transportadas a las instalaciones del centro de investigación en ciencia de los alimentos (CICAL) de la escuela profesional de ingeniería de industrias alimentarias para realizar la caracterización fisicoquímica de la misma la cual estuvo comprendida por: acidez, la cual se realizó mediante titulación alcalimetría según el método AOAC - 947.05 que consistió en la titulación de la muestra con NaOH 0.1N como titulante y solución alcohólica de fenolftaleína al 1% como indicador, los cálculos fueron expresados en gramos de ácido láctico por 100 ml (g/100ml) de leche. Los sólidos totales por desecación en una estufa de convección de marca Binder a 105 ± 2 °C expresados en g/100 ml, el porcentaje de proteínas por el método Kjeldahl AOAC 928.08, la densidad de la leche se determinó con un lactodensímetro QUEVENNE graduado mediante la metodología AOAC 925.22. El pH, fue medido en un potenciómetro de mesa SI Analytics modelo Lab 850 y el contenido de grasa por el método de digestión ácida en butirómetro con ácido sulfúrico concentrado aplicando la metodología AOAC 989.05.
Análisis microbiológico
Para determinar la presencia de coliformes totales y realizar el conteo correspondiente se utilizó la técnica de dilución y sembrado en placa, para tal efecto se tomó 10 ml de la muestra la cual fue diluida en un matraz Erlenmeyer conteniendo 90 ml de solución salina peptonada al 1%, esta mezcla constituyó la primera dilución (10-1) seguidas de diluciones en tubos con 9 ml de la misma solución, llegando hasta la dilución 10-4, luego se depositó 1 mL de cada dilución en placas Petri con agar MacConkey, las cuales fueron llevadas a incubación a 37 °C por 24 h, Asimismo, para el conteo de mesófilos aerobios se realizó la misma metodología con la única variante que fue el medio de cultivo, ya que en este caso se usó Plate Count Agar, los resultados para ambos casos se expresaron en unidades formadoras de colonia por mililitro (ufc/mL).
Cepa de levadura y condiciones de crecimiento
Los experimentos fueron realizados con cepas liofilizadas de Saccharomyces boulardii (Hansen CBS 5926) marca Merck de procedencia mexicana siendo la leche fresca de vaca el medio de cultivo para este microorganismo, la misma que fue separada en matraces de 250 ml, las cuales fueron esterilizadas en autoclave marca STURDY a 121 ºC por 15 min, parámetros bajo los cuales se evita la interferencia de otros microorganismos asegurando la esterilidad de los medios.
Luego se procedió a realizar la activación de Saccharomyces boulardii en una solución de sacarosa al 2%, luego se tomó 1 ml de la solución de la cepa y se pasó a inocular en la leche fresca de vaca a una temperatura de 37 ± 1 ºC, con una agitación constante de 20 RPM regulado por un baño maría BS - 11 y a cada una hora (0 - 6 horas de incubación) se retiraba cada matraz para el conteo correspondiente; asimismo, se realizó monitoreo de pH, porcentaje de acidez según la metodología antes descrita.
Crecimiento microbiano y modelamiento matemático
Para las lecturas del incremento de biomasa se utilizó un microscopio monocular LW SCIENTIFIC y una cámara Neubauer de 0,100 a 0,00025 mm2 expresando los resultados en unidades formadora de colonias por mililitro (ufc/ml) los cuales fueron convertidos a escala logarítmica y así realizar el respectivo modelamiento cinético mediante el algoritmo Gauss Newton en el software Minitab v 19 con las siguientes ecuaciones:
Modelo de Gompertz
Donde: N es el número de microorganismos a un tiempo t, No > 0 es el número de microorganismos en momento inicial, el parámetro C: logaritmo común de la diferencia entre la población inicial y final en la fase estacionaria, el parámetro B representa la pendiente de la curva y describe la tasa de crecimiento y M tiempo en el cual la tasa de crecimiento es de mayor magnitud (Castro et al., 2008).
Modelo Logístico
Modelo Logístico Modificado
Modelo de Weibull
Asimismo, se halló la velocidad especifica de crecimiento (µmáx), duración de la fase de latencia (λ) y el tiempo de generación (G) (Cayré, Vignolo, y Garro (2007).
La comparación de los diferentes modelos cinéticos se realizó tomando la factibilidad de los mismos para alcanzar el ajuste, determinando las constantes de crecimiento correspondientes a cada modelo matemático (Castro et al., 2008).
Modelos de bondad de ajuste
Para realizar el ajuste de los modelos para cada tratamiento se aplicó los criterios estadísticos sugeridos por Torres, Barbosa, Meyer, Noda, y Sarduy (2012) entre ellos tenemos a: iteraciones, suma final de los cuadrados del error, cuadrado medio del error, desviación estándar y el coeficiente de determinación R2.
Suma final de los cuadrados del error
Cuadrado medio del error
Coeficiente de determinación R2
Resultados
Análisis fisicoquímico y microbiológico
El análisis fisicoquímico muestra que la leche evaluada cumple con los parámetros establecidos por la NTP 201.001.2003, mostrados en la Tabla 1.
Tabla 1 Análisis fisicoquímico de la leche.
| Parámetro | Resultados | Requisitos* |
|---|---|---|
| Proteína (%) | 3,13 | - |
| Grasa (%) | 3,51 | 3,20 |
| Solidos totales (%) | 12,70 | 11,40 |
| Acidez láctica (%) | 0,18 | 0,13 - 0,17 |
| pH | 6,0 | - |
| Densidad (g/ml) | 1,028 | 1,029 - 1,034 |
*NTP 202.001.2016
La tabla 2, muestra los resultados del análisis microbiológico, en ella se puede observar que el parámetro de mesófilos aerobios es superior a coliformes totales.
Crecimiento microbiano y modelamiento matemático
En la tabla 3 se puede apreciar los valores de crecimiento de Saccharomyces boulardii tanto en ufc/mL como en escala logarítmica para su posterior modelado matemático, en estos valores se puede apreciar claramente un orden creciente en función al tiempo.
Tabla 3 Crecimiento microbiano de Saccharomyces boulardii
| Tiempo (Horas) | Crecimiento Celular | Varianza | Mínimo | Máximo | Error Típico | |
|---|---|---|---|---|---|---|
| ufc/mL (DE*) | LN | |||||
| 0 | 5,5 ± 1,3 | 0,00 | 1.7 | 4,0 | 7,00 | 0,28 |
| 1 | 14,0 ± 2,6 | 0.93 | 6.7 | 11,0 | 17,00 | 0,57 |
| 2 | 25,5 ± 3,1 | 1,53 | 9.7 | 22,0 | 29,00 | 0,86 |
| 3 | 165,5 ± 9,9 | 3,40 | 97.7 | 156 | 175 | 5,48 |
| 4 | 604 ± 160,4 | 4,69 | 25741.3 | 408 | 800 | 113,16 |
| 5 | 1875 ± 62,4 | 5,83 | 3900.0 | 1800 | 1950 | 43,30 |
| 6 | 3050 ± 44 | 6,31 | 1933.3 | 3000 | 3100 | 28,86 |
*DE: Desviación estándar; n = 3; LN: Logaritmo natural.
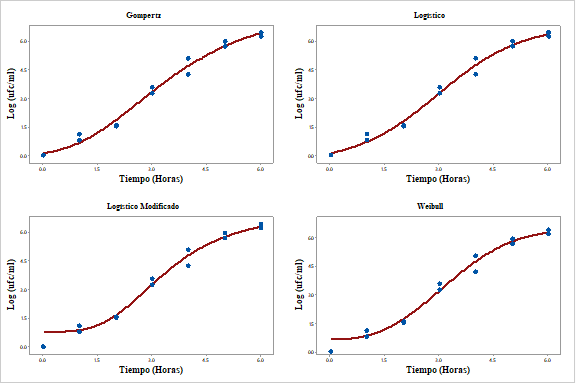
En la figura 1 se muestran los puntos experimentales, la cinética microbiana y los modelos matemáticos en los que se puede apreciar diferentes ajustes en las curvas con similitudes en los modelos de Gompertz y Logístico y de igual similitud en los modelos de Logístico modificado y Weibull.
En la tabla 4 se muestra los parámetros de la cinética de crecimiento, con respecto al logaritmo común de la diferencia entre la población inicial y final en la fase estacionaria (C) Gompertz reporta el mayor valor con 8,0 seguido de Logístico modificado con 7,1 y finalmente de logístico y Weibull con valores inferiores de 7,0 y 6,475 respectivamente, la tasa de crecimiento (B) fue mayor en el modelo Logístico modificado con 3,474 seguido de Weilbull con 2,626; sobre el tiempo de mayor magnitud en la tasa de crecimiento (M) los modelos Logístico y Logístico modificado presentaron los valores mayores (2,968 y 3,416 respectivamente) con respecto a µmáx, λ y G cada una de las ecuaciones presento diversos valores que pueden ser aplicados para diversos objetivos.
Tabla 4 Parámetros de la cinética de crecimiento por cada modelo matemático
| Modelo | Parámetros | ||||||
| C | B | M | D | µmáx | λ | G | |
| (h-1) | (h) | (h) | |||||
| Gompertz | 8,0 | 1,446 | 0,548 | - | 4,115 | 0,813 | 0,168 |
| Logístico | 7,0 | 1,152 | 2,968 | -0,413 | 20,274 | 0,051 | 0,034 |
| Logístico Modificado | 7,101 | 3,474 | 3,416 | 0,802 | 24,257 | 0,724 | 0,028 |
| Weibull | 6,475 | 2,626 | 0,031 | 0,682 | 0,200 | 52,45 | 3,453 |
Modelos de bondad de ajuste
La tabla 5, muestra los criterios estadísticos y el ajuste que se calculó correspondiente a cada modelo matemático; el coeficiente de determinación (R2) muestra que el mejor ajuste corresponde a Gompertz (R2= 0,98), sin embargo, todos los ajustes presentaron valores superiores 0,8, también los valores de las iteraciones sugieren que los modelos de Gompertz y Logístico presentan los valores más bajos (11,0) con un mismo dato encontrado ya en el caso de los modelos Logístico modificado y Weilbull con los valores superiores (15,0 y 17,0 respectivamente); con respecto a la suma de cuadrados del error el modelo Logístico modificado presento el valor más bajo (0,650) y el mayor valor el modelo de Gompertz (0,941), asimismo los grados de libertad del error nos muestran un mayor análisis de datos (11,0) en la ecuación de Gompertz con respecto a los demás, referente al cuadrado medio del error el modelo matemático de Logístico modificado presento el valor más bajo (0,081) a diferencia de Weibull que obtuvo el valor más alto (0,088) y finalmente la desviación estándar muestra que el modelo que tuvo menor variabilidad de datos fue el modelo Logístico modificado en comparación a los demás modelos cinéticos.
Tabla 5 Criterios estadísticos
| Criterios | Modelos cinéticos | |||
|---|---|---|---|---|
| Gompertz | Logístico | Logístico Modificado | Weibull | |
| R - cuadrado | 0,98 | 0,91 | 0,90 | 0,88 |
| Iteraciones | 11,0 | 11,0 | 15,0 | 17,0 |
| Suma de Cuadrados del Error (SCE) | 0,941 | 0,832 | 0,650 | 0,707 |
| Los grados de libertad para el error | 11,0 | 10,0 | 8,0 | 8,0 |
| El cuadrado medio del error | 0,085 | 0,083 | 0,081 | 0,088 |
| desviación estándar | 0,292 | 0,288 | 0,285 | 0,297 |
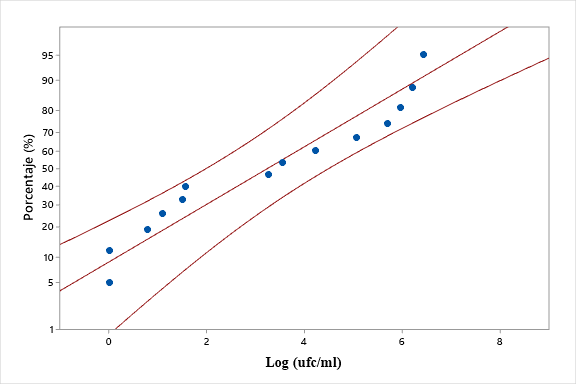
La figura 2 muestra en análisis de normalidad en la que se puede observar que los datos obedecen a una distribución normal indicando una alta confiabilidad de la investigación desarrollada, con un valor p > a 0,05 (0,181)
Monitoreo de pH y acidez
Con respecto al pH y la acidez se observa que no hubo cambios sustanciales en estos (Tabla 6) sobre el pH se observa un descenso de 6.9 a 6.0; con respecto al % de acidez se ve un incremento de 0,18 a 0,20.
Discusión
Con respecto a los valores hallados en la tabla 1 se halló similitud a los reportados por Brousett, Torres, Chambi, Mamani, y Gutiérrez (2015), con un % de proteínas promedio de 3.04 ± 0.18, con respecto al % de grasa con 3.31 ± 0.18, sobre él % de contenido de sólidos totales valores promedio de 12.40 ± 0.47, el % de acidez láctica con 0.18 ± 0.01, pH de 6.5 ± 0.3 y una densidad de 1.029 g/ml, todas estas son indispensables en el metabolismo de la levaduras sobre todo por su rica composición de compuestos nitrogenados que intervienen en el anabolismo y catabolismo. Asimismo, los valores de coliformes totales hallados (Tabla 2) son bajos en comparación a los de Ccopa (2009), con un valor de 2 x 104 ufc/ml; sin embargo, ocurre lo contrario con Mesófilos aerobios, con un valor de 174 x 102; es así que para Fuentes, Ruiz, Sánchez, Ávila, y Escutia (2013), estos parámetros son muy importantes ya que de ellos depende la calidad sanitaria de la leche.
Los valores de crecimiento microbiano (Tabla 3) obtenidos nos muestran que la leche fresca de vaca usada en el experimento contiene las cantidades suficientes en nutrientes para el buen desarrollo de la biomasa y metabolitos de SB. Según Mesa, Mas, Anaya, Cobo y Díaz (2016), atribuyen este fenómeno a que la leche contiene proteínas y azúcares, para realizar el metabolismo necesario para Saccharomyces boulardii. Asimismo, Zumbado, Esquivel, y Wong (2006) concuerdan con lo expuesto, ya que en su estudio en la levadura Kluyveromyces marxianus demostraron que los componentes de la leche funcionan como un buen sustrato para las levaduras obteniendo biomasa y metabolitos.
Las curvas de crecimiento de Saccharomyces boulardii (Figura 1) mostradas se adecuan a la elaboración de cinética microbiana y estas son: según Castro et al., (2008), mejores que los modelos de regresión lineal simple, puesto que los modelos no lineales pueden determinar las fases de crecimiento de los microorganismos aplicados en numerosos estudios y necesario para el desarrollo de maquinarias. Vanegas y Ramírez (2016) en su estudio del crecimiento de Pseudomonas aplicando el modelo de Gompertz, Logístico y Baranyi, describe que el modelo de Gompertz es óptima en la construcción de las curvas de crecimiento microbiano de modo que los resultados obtenidos corroboran lo demostrado en el presente trabajo. Por otro lado, los parámetros cinéticos hallados (Tabla 4) describen el comportamiento de cada una de las curvas nos permiten aprovechar cada una de sus características tales como la tasa máxima de crecimiento, la velocidad y demás mencionados, Castro et al. (2008) concuerda, en su estudio que existe similitud de parámetros entre los modelos sigmoidales ya que estos definen el crecimiento en curvas bien definidas; es así que los modelos matemáticos aplicados en la cinética microbiana pueden describir el comportamiento celular en bacterias como Pediococcus acidilactici (Chowdhury, Chakraborty, y Chaudhuri, 2007), para Belda et al. (2014), los modelos como Gompertz permite aprovechar las bondades de estas ecuaciones con respecto a las constantes cinéticas.
Por otro lado, los valores de los criterios estadísticos analizados nos muestran que los modelos presentan un buen ajuste los cuales concuerdan con los datos hallados por Torres et al. (2012); puesto que, en su estudio demostró que el modelo logístico presenta un mejor ajuste. En el caso de las iteraciones Candotti, Mavares, y Velásquez (2014) sugieren la aplicación de estas para la optimización en el uso de modelos matemáticos siendo los de mejor ajuste las ecuaciones de Gompertz y Logístico, la diferencia en los valores de las iteraciones se debe a la convergencia de los datos analizados para cada uno de los modelos, es decir a mayor valor la convergencia será más alta; por otro lado, estos influyen directamente en el SCE (Gil, 2006). Castro et al. (2008), muestra valores altos en SCE que oscila entre 3,43 a 3,93 siendo el más bajo otorgado al modelo de Richards y el más alto al de Gompertz que se asemeja a lo hallado en la presente investigación, sobre los grados de libertad del error González, Goicochea, Quintero, Rubio, y Aranguren (2007), mencionan que un valor alto en los grados de libertad es prueba de una alta significancia, la cual corresponde al modelo de Gompertz en nuestra investigación.
Asimismo, para Montgomery, Peck, y Vining, (2018), esta distribución de datos nos muestra que existe normalidad siendo el modelado de estos el correcto (Figura 2).
Nissen, di Carlo, y Gianotti (2020), en su trabajo de fermentación de bebidas por probióticos con diversos microorganismos mostró descensos de pH de 7.18 ± 0.01 a pH 4.74 ± 0.10 en un tiempo de seis horas, en contraste a lo observado en nuestro estudio (Tabla 6) se ve un descenso más lento; con respecto a la acidez Chavan, Gat, Harmalkar, y Waghmare (2018), reportó un incremento de esta con respecto al tiempo tal y como ocurre en nuestros tratamientos.
Conclusiones
Mediante el modelado matemático se logró evaluar las curvas de crecimiento microbiano, estas fueron muy precisos para describir los comportamientos de Saccharomyces boulardii en leche de vaca; cada modelo muestra un ajuste diferente siendo el modelo Logístico y Logístico modificado los que tuvieron un mejor desempeño en el modelamiento, seguido de Gompertz y Weibull.
Asimismo, se obtuvo los parámetros microbiológicos que permiten describir el comportamiento adecuado de la curva de crecimiento que permiten predecir, determinar condiciones óptimas para el crecimiento microbiano y así aprovechar cada una ellas a diferentes aplicaciones.
Finalmente, resulta importante seguir con estudios que permitan evaluar la utilización de múltiples sustratos con cepas probióticas y modelos matemáticos para el desarrollo de productos probióticos.












 uBio
uBio