Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO
Related links
-
 Similars in
SciELO
Similars in
SciELO  uBio
uBio
Share
Arnaldoa
Print version ISSN 1815-8242On-line version ISSN 2413-3299
Arnaldoa vol.25 no.3 Trujillo Sept./Dic. 2018
http://dx.doi.org/10.22497/arnaldoa.253.25310
ARTÍCULOS ORIGINALES
Estabilidad genética en plantas de Vitis vinifera cv. red globe, La Libertad, Perú, evaluados con los microsatélites VVMD5 y VsZAG79
Genetic stability in plants of Vitis vinifera cv. red globe, La Libertad, Peru, evaluated with microsatellites VVMD5 and VsZAG79
Pamela Luna Cardoso1, David Salirrosas1 & Zulita Prieto1*
1 Universidad Nacional de Trujillo, Trujillo, PERÚ
*Autora para correspondencia: zprieto@unitru.edu.pe
Resumen
El objetivo del presente trabajo fue estimar la variación genética en un cultivo de la variedad red globe (Vitis vinifera) mediante los microsatélites VVMD5 y VrZAG79. Para ello, se obtuvieron hojas de plantas del campamento San José de Chavimochic, La Libertad, Perú. Se procedió con la extracción de ADN, la amplificación por PCR y electroforesis para los microsatélites VVMD5 y VrZag79. Se observaron loci monomórficos para ambos marcadores, los tamaños de fragmentos amplificados fueron de 213,6 y 239,7 bp para VVMD5 y de 242,2 bp para VrZAG79. Como conclusión, los genotipos muestran estabilidad durante las réplicas que se realizaron por clonación vegetativa para obtener las plantas a partir de un genotipo inicial. Sin embargo, será necesaria la evaluación de los genotipos con marcadores adicionales.
Palabras clave: Vitis vinifera, vid, red globe, marcadores microsatélites.
Abstract
The aim of this work was to estimate the genetic variation in a crop of red globe variety (Vitis vinifera) by means of the microsatellites VVMD5 and VrZAG79. For this, plant leaves were obtained from San Jose camp at Chavimochic, La Libertad, Peru. Then, we proceeded with the extraction of DNA, the amplification by the polymerase chain reaction (PCR) and electrophoresis for the microsatellites VVMD5 and VrZag79. We observed monomorphic loci for both markers, the sizes of amplified fragments were of 213.6 and 239.7 bp for VVMD5 and of 242.2 bp for VrZAG79. As conclusion, the genotypes show stability during the replicas that were realized by vegetative cloning to obtain the plants from an initial genotype. Nevertheless, the evaluation of the genotypes will be necessary with additional markers.
Keywords : Vitis vinifera, grape, red globe, microsatellite markers.
Introducción
En Vitis vinifera L. (Vitaceae) se distinguen dos subespecies conocidas como Vitis vinifera spp. sativa, que agrupa a las variedades y poblaciones domesticadas, y Vitis vinifera spp sylvestris, poblaciones silvestres (Arroyo-García et al., 2006). Quiénes proponen, que V. vinifera tendría múltiples origenes e irradiación a diferentes países del mundo, existiendo dos principales grupos, un grupo conformado por la Península Iberiana, Europa Central y África Norte, y el otro grupo mayor, principalmente especies silvestres, Península Italiana, Península Blakan, Europa Este y otros países cercanos.
Del estudio de veinte loci de microsatélites nucleares establecieron rasgos distintivos de V. vinifera spp. sylvestris y V. vinifera spp. sativa, destacan la singularidad del acervo genético de importancia en estudios de la domesticación y conservación de los recursos genéticos de la "vid" (Imazio et al., 2013). De Lorenzis et al., 2015, presentaron evidencias que la domesticación de la "vid" tuvo lugar en la región caucásica y demostraron diferencias moderadas entre sativa y sylvestris en Giorgia. Otro país, con alta diversidad de genotipos se cita a Israel, en la que reportaron 10 loci SSR polimórficos y entre ellos, los microsatélites VVMD5 y VrZAG79 (Drori et al., 2017).
A partir de Vitis vinifera spp sativa se han derivado muchas variedades, que se cultivan en diferentes partes del mundo y en muchos casos tienen el mismo nombre para una variedad o de acuerdo a las regiones que crecieron ocurre una convergencia de nombre aun siendo diferentes cultivares, con el objeto de realizar aclaraciones, surge la ampelografía, encargada de identificar a las variedades de V. vinifera L. usando caracteres morfológicos (Chavez 1999); sin embargo, no es completamente fiable para la identificación de cultivares y portainjertos, debido a los cambios del fenotipo durante las etapas de crecimiento e influenciados por las condiciones climáticas (Lamboy et al., 1998; Sefc et al., 2001).
Ulanovsky et al. (2002), utilizaron microsatélites y cebadores arbitrarios (RAPD) y observaron variaciones entre y dentro de variedades, y reportaron accesiones duplicados con el mismo nombre. Estudios recientes, con nueve marcadores SSR (microsatélites) y ocho SNP, determinaron la existencia de dos grupos con una alta variabilidad entre cultivos procedentes de regiones occidentales de Irán (Vafaee et al., 2017).
En la región La Libertad, Perú, existen plantaciones de "vid" de la variedad Red Globe, que se distinguen por sus frutos de coloración roja y gran aceptación en el mercado local y de exportación, y requieren evaluaciones del grado de estabilidad genotípica de la variedad durante los proceso de propagación vegetativa. Por ello, el interés del presente trabajo fue determinar si existen diferencias genéticas, intrapoblacional, de un cultivo de V. vinifera cv. red globe en base a los marcadores microsatélites VVMD5 y VrZAG79.
Materiales y métodos
Material biológico
Las muestras fueron obtenidas del campamento San José del Proyecto Especial Chavimochic ubicado en Virú - La Libertad, Perú. Un total de dieciocho plantas fueron seleccionadas al azar de cultivares de Vitis vinifera L. cv. red globe. La identificación de la especie fue realizada en el Herbario Antenor Orrego (HAO) y el código asignado fue HAO-20058.
Las muestras, hojas frescas y jóvenes fueron preservadas a -80°C en el Laboratorio de Genética y Biología Molecular, Universidad Nacional de Trujillo.
Extracción de ADN
El ADN de cada accesión fue extraído mediante el protocolo de Lamboy et al. (1998) modificado, que consiste en moler la hoja con nitrógeno líquido, el polvillo de la hoja fue transferido rápidamente a un tubo de 1,5 ml, mezclado por inversión con 600 µl de buffer (250mM Tris-HCL pH 8,0; 250 mM NaCl; 50 mM EDTA; PVP 4%, SDS 1,5%; y beta-mercaptoetanol 1,0%) durante 10 min. Se retiró la fase líquida a otro tubo de 1,5 ml y se agregó la misma cantidad de cloroformo se mezcló y se llevó a la centrífuga refrigerada por 15 min a 13000 rpm a 4°C. Al precipitado, se agregó NaCl 5M, cantidad aproximada de 0,5 ml y se centrifugó 13000 rpm a 4ºC 15 min. Luego, se retiró el sobrenadante y se agregó isopropanol y nuevamente fue centrifugado. El pellet fue lavado con etanol al 70%, y la suspensión final en buffer TE (10 mM TRIS – 1mM EDTA con pH 8). La calidad del ADN extraído fue determinada por electroforesis en un medio de gel de agarosa al 1% en buffer TAE 1X. Se seleccionaron las diez mejores extracciones de ADN para continuar con la amplificación.
PCR (Reacción en cadena de la polimerasa)
Las secuencias de cebadores para VVMD5 fueron: F-CTAGAGCTACGCCAATCCAA, y R-TATACCAAAAATCATATTCCTAAA y para VrZAG79, AGATTGTGGAGGAGGGAACAAACCG y R-TGC CCCCATTTTCAAACTCCCTTCC (Rodríguez-Plaza et al. 2006).
Se realizó las pruebas para la hibridación óptima de cebadores a distintos gradientes de temperatura.
La amplificación se realizó según Karatas et al. (2007) y Jahnke et al. (2007), con algunas modificaciones. Que consistió en lo siguiente: para un volumen de 25 µl se usó tanto para los microsatélites VVMD5 y VrZAG79, 2,5 µl de 10x Coral Load PCR, 0,5 µl de dNTP mix (10 mM c/u), 1 µl de cada primer (forward y reverse), 0,125 µl de HotStart Taq plus DNA polymerase, 16,875 µl de agua ultra pura y 3 µl de ADN.
Las temperaturas y tiempos de amplificación, fueron realizadas según Cuhnaet al. (2010), Nookaraju & Agrawal (2002) y Poblete et al. (2011). Ciclo 1 de 95 °C durante 5 min, ciclos siguientes (35 ciclos) de 94 °C 1 min, 45 °C 1 min (para VVMD5) y 55°C para VrZAG79 y 72°C 1 min, con una extensión final 72°C por 10 min.
Electroforesis
Se tomó 5 µl de producto amplificado por reacción y se analizaron en un gel de agarosa al 2 % con 4 µl bromuro de etidio, en Buffer TAE 1X. Se utilizó un marcador de peso molecular Sigma de 100 pb. La electroforesis fue llevada a cabo en cámara horizontal a 100 V por 30 min. Posteriormente, el gel fue observado en UV Bio-Rad ChemiDoc XRS para la lectura de geles.
Análisis de datos
Se consideró como homocigotos a los genotipos por la presencia de una sola banda por locus.
Resultados
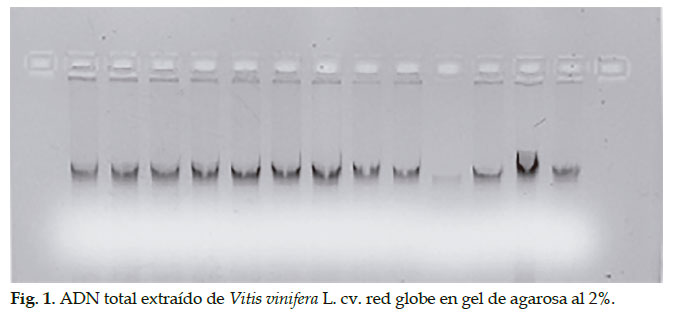
La extracción de ADN fue de buena calidad (Fig. 1) y la temperatura óptima para la hibridación de cebadores para el microsatélite VVMD5 fue 45ºC y para, VrZAG79 fue 55ºC.
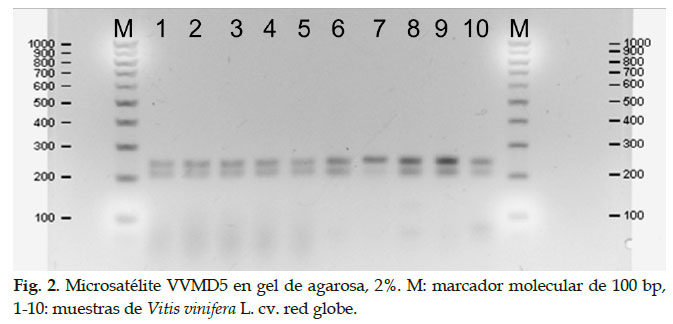
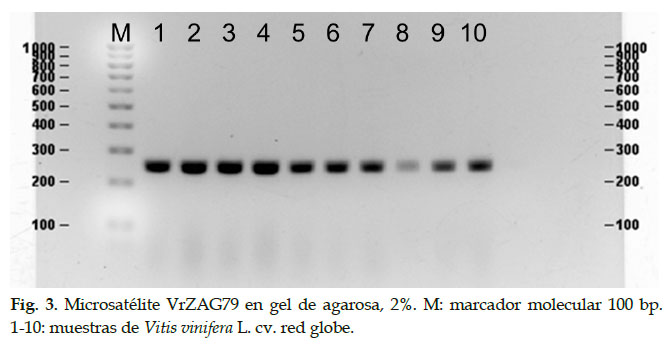
Los loci VVMD5 y VrZAG79 amplificaron en las diez muestras tomadas de Vitis vinífera L. cv. red globe. Para el microsatélite VVMD5 amplificó dos bandas en las 10 muestras (Fig. 2), y el microsatélite VrZAG79 amplificó una sola banda en las 10 muestras (Fig. 3). Ambos loci amplificaron entre 200 a 300 pares de bases con un promedio para el marcador VVMD5 la banda de migración rápida fue 213,6 ± 1,43 y la de migración lenta 239,7 ± 7,76 y para el VrZag79 de 242,2 ± 3,01.
Discusión
Los microsatélites de ADN son frecuentemente utilizados para la identificación de especies de Vitis vinifera, por su alta sensibilidad y especificidad. Para lo cual, la calidad del ADN extraído debe ser la adecuada, como la registrada en nuestros resultados.
En el microsatelite VVMD5, se observó dos bandas en todas las muestras. Ante este resultado, se puede deber a que en el cultivo muestreado, todas provienen de un genotipo inicial y que han sido propagadas por clonación. Otra explicación es que fortuitamente se presentan variaciones en la hibridación, que se podrían generar alineamientos incorrectos por las secuencias repetidas durante la amplificación generando bandas de diferente tamaño (Ellegren, 2004). Sin embargo, por la presencia homogénea de dos bandas en todos los genotipos, consideramos la primera explicación.
El perfil de bandas fue igual en todas las muestras de V. vinifera cv. red globe, para ambos microsatélites VVMD5 y VrZAG79, en ambos, monomórfico. Los tamaños de los fragmentos amplificados para VVMD5 y para el VrZAG79 fueron concordantes con lo reportado por Zulini et al. (2002) para la misma variedad, red globe y contrariamente a nuestro reporte, los mencionados autores registraron 7 alelos para VVMD5 y 10, para VrZAG79, con una heterocigosidad de 81,0 y 82,0%, respectivamente.
La ausencia de variación en las muestras red globe del presente trabajo, se explicaría por las réplicas de plantones (reproducción asexual) obtenidos a partir de un genotipo inicial, resultados que estarían confirmando la estabilidad genética de estos genotipos.
Reportes, para los mismos marcadores mencionados, en 25 variedades de "uvas" de procedencia de diferentes lugares del sur de Perú y Argentina, encontraron tamaños de fragmentos de 228 a 240 bp para VVMD5 y de 240 a 259 bp para VrZAG79, tamaños de ADN amplificados similares a los reportados por distintos autores, y en el conjunto de variedades mostraron diferencias genéticas entre variedades, número de alelos 6 y 13, para VVMD5 y VrZAG79, respectivamente, (Martínez et al., 2006). Resultados similares fueron reportados por Jahnke et al. (2009), en la comparación de 48 variedades de V. vinifera y entre los marcadores, destacan al marcador VrZAG79, polimórfico, presencia de 12 alelos, número de genotipos 28, frecuencia de uno de alelos 0,2604, diversidad alélica 0,8589, variación inter-varietal.
Contrariamente, en el análisis de 10 genotipos (clones) de la variedad white resling, observaron ausencia de diferencias genéticas entre los genotipos con los marcadores VVMD5 y VrZAG79 (Regner et al., 2000). Niveles bajos de variación fueron observados también en la variedad refosk (Kozjak et al., 2003). Similar a los registrados en el presente trabajo.
Son necesarias mayores investigaciones sobre el grado de variación del germoplasma de una especie tan apreciada a nivel mundial, dado las alertas de disminución de la variación genética en V. vinifera. Marrano et al. (2018), de los registros de vides cultivadas de diferentes países y las vides silvestres (V. vinifera), principalmente de Italia, dieron a conocer la reducción general de diversidad genética del estado silvestre, lo que conllevaría a la disminución progresiva de poblaciones nativas. La búsqueda de información sobre la diversidad genética y fenotípica en las vides, apertura nuevos enfoques de estudio de asociación genética que procuren información sobre los rasgos agronómicos y su interacción con el ambiente (Nicolas et al., 2016; Migicovsky et al., 2017).
Conclusión
Se demostró la estabilidad genética de los genotipos de V. vinifera var. red globe, en un cultivo de La Libertad, Perú, con los marcadores VVMD5 y VrZAG79.
Agradecimientos
Al Mblgo. José Peredo, por su apoyo en la obtención del material biológico y a las Blgo. Linda Sánchez y Mónica Arqueros por su colaboración en el proceso de síntesis de ADN (PCR).
Contribución de los autores
P.L.C. Ha realizado la colecta del material biológico, el proceso para la obtención de resultados y redacción del manuscrito. D.S. Ha contribuido con la identificación de la especie, apoyo en el proceso de obtención de datos y redacción del manuscrito. Z.P. Ha participado en el diseño y planificación del trabajo, interpretación de resultados, redacción y revisión del manuscrito final.
Conflicto de intereses
Los autores declaran no tener conflicto de intereses.
Literatura citada
Arroyo, R. L.; L. Ruiz; L. Bolling; R. Ocete; M. López; Arnold; A. Ergul; G. Söylemezoğlu; H. Uzun; F. Cabello; J. Ibáñez; M. Aradhy; A. Atanassov; I. Atanassov; S. Balint; J. Cenis; L. Costantini; S. Goris-Lavets; M. Grando; B. Klein; P. McGovern; Merdinoglu; I. Pejic; F. Pelsy; N. Primikirios; V. Risovannaya; K. Roubelakis-Angelakis; H. Snoussi; P. Sotiri; S. Tamhankar; P. Esta; L. Troshin; J. Malpica; F. Lefort & J. Martínez- Zapater. 2006. Multiple origins of cultivated grapevine ( Vitis vinifera L. ssp. sativa) based on chloroplast DNA polymorphisms. Molecular Ecology 15(12): 3707–3714. [ Links ]
Chavez, J. E. 2000. Identificación de variedades de vid ( Vitis vinifera L.) por ampelografía: capacidad discriminante y estabilidad. Detección de homonimias y sinonimias. Tesis Doctoral. Universidad Politécnica de Madrid, Escuela Técnica Superior de Ingenieros Agrónomos, Madrid. [ Links ]
Cunha, J.; M. Teixeira-Santos; M. Veloso; L. Carneiro;Eira-Dias & P. Fevereiro. 2010. The portuguese Vitis vinifera L. germplasm: genetic relations between wild and cultivated vines. Ciencia e Tec. Vitiv. 25(1): 25-37. [ Links ]
De Lorenzis, G.; R. Chipashvili; O. Failla & D. Maghradze. 2015. Study of genetic variability in Vitis vinifera L. germplasm by high-throughput Vitis18k SNP array: the case of Georgian genetic resources. BMC Plant Biol. 15: 154.
Drori, E.; O. Rahimi; A. Marrano; Y. Henig; H. Brauner; Mali Salmon-Divon; Y. Netzer; M. Prazzoli; M. Stanevsky; O. Failla; E. Weiss & M. Grando. 2017. Collection and characterization of grapevine genetic resources ( Vitis vinifera) in the Holy Land, towards the renewal of ancient winemaking practices. Sci Rep. 7: 44463. [ Links ]
Ellegren, H. 2004. Microsatellites: Simple Sequences with Complex Evolution. Nature Reviews /Genetics. 5: 435. [ Links ]
Imazio, S.; D. Maghradze; G. De Lorenzis; R. Bacilieri; V. Laucou; P. This; A. Scienza & O. Failla. 2013. From the cradle of grapevine domestication: molecular overview and description of Georgian grapevine (Vitis vinifera L.) germplasm. Tree Genetics & Genomes 9(3): 641–658. [ Links ]
Jahnke, G.; J. Ma´jer; A. Lakatos; J. Molna´r; K. Dea´; E. Stefanovits-Ba´nyai & P. Varga. 2009. Isoenzyme and microsatellite analysis of Vitis vinifera L. varieties from the Hungarian grape germplasm. Scientia Horticulturae 120: 213–221.
Jahnke, G.; J. Korbuly; J. Májer; A. Lakatos; M. Gyorffyne; E. Dea´k; E. Stefanovits-Bányai & P. Varga. 2000. Isoenzyme and microsatellite analysis of Vitis vinifera L. varieties from the Hungarian grape germplasm. Scientia Horticulturae 120: 213–221.
Karatas¸ H.; D. Degirmenci; R. Velasco; S. Vezzulli; C. Bodur & Y. Agaoglu. 2007. Microsatellite fingerprinting of homonymous grapevine (Vitis vinifera L.). Varieties in neighboring regions of South-East Turkey. Scientia Horticulturae 114:164– 169. [ Links ]
Kozjak, P.; Z. Korosec-Koruza & B. Javornik . 2003. Characterisation of cv. Refosk (Vitis vinifera L.) by SSR markers. Vitis 42(2): 83-86. [ Links ]
Lamboy, W. & C. Alpha . 1998. Using simple sequence repeats (SSRs) for DNA finger printing germplasm accessions of grape (Vitis L.) species. J. Amer. Soc. Hort. Sci. 123(2): 182-188. [ Links ]
Marrano, A.; D. Micheletti; S. Lorenzi; D. Neale & M. Grando. 2018. Genomic signatures of different adaptations to environmental stimuli between wild and cultivated Vitis vinifera L. Hortic Res. 5: 34. [ Links ]
Martínez, L. E.; P. Cavagnaro; R. W. Masuelli & M. Zuñiga. 2006. SSR-based assessment of genetic diversity in South American Vitis vinífera varieties. PlantScience 170: 1036-1044. [ Links ]
Migicovsky, Z.; J. Sawler; K. M. Gardner; M. K. Aradhya; B. H. Prins; H. R. Schwaninger; C. D. Bustamante; E. S. Buckler; G. Y. Zhong; P. J. Brown & S. Myles. 2017. Patterns of genomic and phenomic diversity in wine and table grapes. Hortic Res. 4: 17035. [ Links ]
Nicolas, S. D.; J. P. Péros; T. Lacombe; A. Launay; M. Le Paslier; A. Bérard; B. Mangin; S. Valière; F. Martins; L. Le Cunff; V. Laucou; R. Bacilieri; A. Dereeper; P. Chatelet; P. This & A. Doligez. 2016. Genetic diversity, linkage disequilibrium and power of a large grapevine ( Vitis vinifera L) diversity panel newly designed for association studies. BMC Plant Biol. 16: 74. [ Links ]
Nookaraju, A. & D. C. Agrawal. 2012. Genetic homogeneity of in vitro raised plants o grapevine cv. Crimson Seedless revealed by ISSR and microsatellite markers. South African journal botany 78: 302-306. [ Links ]
Poblete, I.; M. Pinto; M. T. de Andres & P. Hinrichsen. 2011. Genetic characterization of old grapevines collected in Oases of the Atacama Desert. Chilean Journal of Agricultural Research 71 (3): 476- 482. [ Links ]
Regner, F.; E. Wiedeck & A. Standlbauer. 2000. Differentiation and identification of white Riesling clones by genetic markers. Vitis, 39 (3): 103-107. [ Links ]
Rodríguez-Plaza, P.; R. González; M. V. Moreno- Arribas; M. C. Polo; G. Bravo; J. M. Martínez- Zapater; C. M. Martínez & A. Cifuentes . 2006. Combining microsatellite markers and capillary gel electrophoresis with laser-induced fluorescence to identify the grape ( Vitis vinifera) variety of musts. Eur Food Res Technol 223: 625-631. [ Links ]
Sefc, K. M.; F. Lefort; M. S. Grando; K. D. Scott; H. Steinkellner & M. R. Thomas. 2001. Microsatellite markers for grapevine: a state of the art. Molecular Biology & Biotechnology of grapevine 1-30. [ Links ]
Ulanovsky, S.; Y. Gogorcena; F. Martinez de Toda & J. M. Ortiz. 2002. Use of molecular markers in detection of synonymies and homonymies in grapevines ( Vitis vinífera L.). Scientia Horticulturae 92: 241-254. [ Links ]
Vafaeea, Y.; N. Ghaderia & A. Khadivi . 2017. Morphological variation and marker-fruit trait associations in a collection of grape (Vitis vinifera L.). Scientia Horticulturae 225: 771–782 [ Links ]
Zulini, I.; M. Russo & E. Peterlunger. 2002. Genotyping wine and table grape cultivars from Apulia (South Italy) using microsatellite markers. Vitis 41(4):183- 187. [ Links ]
Recibido: 20-VIII-2018
aceptado: 25-IX-2018
publicado online: 30-XI-2018
publicado impreso: 31-XII-2018