INTRODUCCIÓN
El aguacate (Persea americana Mill) es un fruto tropical originario de Mesoamérica (Galindo-Tovar et al., 2008), reconocido por sus beneficios a la salud humana, debido a su composición nutricional, ya que cuenta con un alto porcentaje de fibra dietética, potasio, sodio y magnesio, además de vitaminas A, C, E, K1, B-6, ácido fólico, ácidos grasos insaturados, además de ser uno de los frutos con la menor cantidad de carbohidratos (Dreher & Davenport, 2013). A nivel mundial, México se ha convertido en el principal productor y exportador de aguacate (Araújo et al., 2018). No obstante, el acelerado crecimiento en el establecimiento de plantaciones de monocultivo de aguacate, lo que ha llevado a un proceso de conversión de bosques naturales en agroecosistemas (Barsimantov & Navia, 2012), favoreciendo el incremento de plagas en este cultivo (Wysoki et al., 2002), a la par del desarrollo de resistencia a los insecticidas sintéticos, así como los efectos dañinos de éstos al ambiente y a la salud humana, esto ha fomentado la investigación y el desarrollo de estrategias de control biológico para un manejo integrado de plagas (MIP) (Arthurs & Dara, 2018).
Dentro de las múltiples alternativas de control biológico que se han propuesto, el aislamiento de hongos entomopatógenos (HE) y su posterior selección, es fundamental para el desarrollo de micoinsecticidas (Thakur & Sandhu, 2010). Entre las especies de HE más estudiadas, destacan Beauveria bassiana (Balsamo) Vuillemin y Metarhizium anisopliae (Metschn.) Sorokin del orden Hypocreales (Ascomycota), ambos enemigos naturales de un amplio espectro de insectos y arañas, con distribución cosmopolita (Roberts & St. Leger, 2004).
Uno de los hábitats donde se ha reportado la presencia de HE es el suelo (Pérez-González et al., 2014; Garrido-Jurado et al., 2015; Uzman et al., 2019), este ambiente natural sirve de resguardo para las esporas de los HE, las cuales encuentran protección contra factores bióticos y abióticos (Baydar et al., 2016), algunas especies de HE como M. anisopliae, B. bassiana e Isaria fumosorosea Wize han sido ampliamente detectadas en suelos agrícolas (Meyling & Eilenberg, 2006), forestales (Thakur & Sandhu, 2010) y áreas no perturbadas (Botelho et al., 2019). Para aislar a estos hongos del suelo, la estrategia más efectiva es el método del “insecto trampa”, en el cual se utilizan larvas del lepidóptero Galleria mellonella L., 1756, o pupas del coleóptero Tenebrio molitor L., 1758 (Goble et al., 2010).
El incremento de materia orgánica en suelos de hábitats naturales, favorece la diversidad y densidad de insectos que pueden ser huéspedes para los HE, lo cual ayuda en su multiplicación (Gebremariam et al., 2021), contrario a lo que ocurre con los agroecosistemas, donde las comunidades de insectos se ven reducidas por el uso de insecticidas y fertilizantes sintéticos y por consiguiente la presencia y diversidad de (Goble et al., 2010; Uzman et al., 2019), estas prácticas agrícolas convencionales limitan la aparición de múltiples especies de Metarhizium (Steinwender et al., 2014, Clifton et al., 2015), donde M. anisopliae suele ser el más persistente bajo estas condiciones (Uzman et al., 2019).
El conocer los factores responsables de la persistencia de algunas especies de HE en el ambiente, es un aspecto importante a considerar para el desarrollo estrategias en programas de control biológico de conservación (Steinwender et al., 2014), ya que se requieren aislados tolerantes a varios factores abióticos (principalmente radiación UV, temperatura y humedad relativa), para su uso como agentes microbianos de control biológico de plagas agrícolas (Imoulan et al., 2011), por lo tanto, se considera que el aislamiento de HE endémicos, para su posterior aplicación como estrategia de control biológico en el ambiente donde fueron obtenidas, puede redituar en una mayor eficacia y persistencia, así como en la reducción en los costos de aplicación (Goble et al., 2010; Fisher et al., 2011; Uzman et al., 2019).
El objetivo del presente estudio fue aislar HE mediante la técnica de insecto-trampa a partir de muestras de suelo de un huerto convencional de aguacate en Colima, México, así como identificar a nivel molecular la diversidad de especies presentes durante un ciclo anual.
MATERIAL Y MÉTODOS
Área de estudio y recolección de muestras de suelo
Los muestreos fueron realizados en un huerto de aguacate (Persea americana var. Hass) de ocho años de edad, en las coordenadas 19°24.828'N y 103°36.991' W a 1552 msnm, en la localidad de Montitlán, municipio de Cuauhtémoc, en el norte del estado de Colima, México. La recolecta de suelo se realizó mensualmente de julio 2016 a junio 2017, donde se tomaron tres muestras de suelo de 500 g cada una. Las muestras se almacenaron y transportaron en una hielera para su posterior análisis en el Laboratorio de la Colección de Hongos Entomopatógenos (CHE) del Departamento de Control Biológico, en Tecomán, Colima, México.
Aislamiento de hongos entomopatógenos
Los HE fueron aislados del suelo mediante el método de insecto trampa utilizando larvas de G. mellonella (Zimmerman, 1986). Las larvas fueron sometidas a un tratamiento para reducir su producción de cera, evitando que entren en estado de pupa durante el periodo de contacto con las muestras de suelo, sumergiéndolas en agua a 56 °C entre 10-15 s, después, se colocaron en agua a 25 ± 2 °C durante 10 s. Posteriormente se pusieron en papel absorbente para retirar el exceso de humedad y se colocaron en una caja de plástico con tapa, donde se sometieron a un periodo de obscuridad de 5 h antes de ser empleadas en los ensayos (Meyling, 2007). Al mismo tiempo, se tomaron 300 g de suelo de las muestras previamente colectadas en campo y se colocaron en un recipiente de plástico de 500 mL, cuando el suelo mostró un bajo porcentaje de humedad, se le agregó agua destilada estéril, evitando la saturación. A continuación, se transfirieron 10 larvas sobre la superficie de cada una de las muestras. Todas las cajas fueron incubadas a una temperatura de 25 ± 2 °C con un fotoperiodo 12:12 h luz/oscuridad, durante 10 días. Las cajas con las muestras de suelo fueron revisadas diariamente y se extrajeron las larvas que mostraban micosis aparente o síntomas de infección por HE. Todas las larvas fueron enjuagadas con agua destilada estéril para remover el suelo adherido. Los HE desarrollados sobre la superficie de las larvas se aislaron con ayuda de una aguja de disección y se sembraron con la técnica de puntos, en cajas de Petri con medio de cultivo Agar Dextrosa Sabouraud (Bioxon®, México) adicionado con extracto de levadura (Bioxon®, México) al 1% (SDAY) [g.l: dextrosa 40, peptona de carne 5, peptona de caseína 5, agar 15, extracto de levadura 10], además del antibiótico cloranfenicol (500 ppm). Todos los aislados se incubaron a una temperatura de 25 ± 2 °C durante 15 días, para posteriormente determinar su morfología macroscópica y microscópica.
Identificación de hongos entomopatógenos
A partir de los HE aislados, se observó el desarrollo macroscópico de las colonias (anverso, forma, elevación, margen y consistencia), en el reverso de la colonia se registró la coloración. Para la identificación micromorfológica se observaron las estructuras reproductivas asexuales de los HE (conidios) mediante laminillas teñidas con lactofenol/azul de algodón (10:1 v/v), utilizando un microscopio AXIO SCOPE A1 (Carl Zeiss, Alemania). Finalmente, para determinar el género y/o especie, se tomó como referencia las claves taxonómicas de Humber (2012).
Extracción de ADN
Los diferentes aislados fúngicos se crecieron en medio de cultivo SDY [g.l.: dextrosa 40, peptona de carne 5, peptona de caseína 5, extracto de levadura 10] y se incubaron a una temperatura de 25 °C en agitación (125 rpm) durante 2 días. El micelio se recuperó mediante filtración al vacío y posteriormente se sometió a un proceso de liofilización. La extracción de ADN genómico se realizó pulverizando 10 mg de micelio liofilizado con nitrógeno líquido, empleando el DNeasy Plant Mini Kit (Qiagen, Valencia, EUA) de acuerdo con las instrucciones del fabricante. La cuantificación y pureza del ADN fue analizada con el espectro-fotómetro Epoch™ Microplate (Biotek, Winooski, USA) usando el índice de absorbancia A260/A280. Las muestras de ADN se almacenaron a -20 °C hasta su posterior uso.
Identificación molecular de los hongos
Para identificar los aislados del género Metarhizium a nivel de especie, se utilizó la secuencia parcial de la región rica en intrones del extremo 5’ del factor de elongación de la traducción 1-α (5’TEF) (Rehner & Buckley, 2005). Este locus se seleccionó debido a su alto grado de diferenciación entre especies y a la congruencia de la topología con las especies del género Metarhizium (Bischoff et al., 2009). La región 5’TEF (~ 800 bp) se amplificó utilizando los cebadores EF1T (5’-ATGGGTAAGGARGACAAGAC-3’) y EF2T (5’-GGAAGTACCAGTGATCATGTT-3’) (Rehner & Buckley, 2005).
La mezcla de reacción de amplificación se preparó a un volumen final de 20 μL con los siguientes reactivos: Phire™ reaction buffer 1X (Thermo Fisher Scientific, Waltham, EUA), 0,2 mM de mezcla de dNTPs (Promega, Madison, EUA), 0,4 mM de cada oligonucleótido, 0,4 μL de Phire™ Hot Start II DNA Polymerase (Thermo Fisher Scientific, Waltham, EUA) y 1 ng de ADN genómico.
El programa de amplificación se realizó mediante PCR “touchdown” (Don et al., 1991), bajo las siguientes condiciones: 1 ciclo de desnaturali-zación inicial a 98 °C por 30 s; seguido de 10 ciclos de 98 °C por 5 s, 66 °C (disminuyendo 1 °C por ciclo) por 5 s y 72 °C por 15 s; en seguida 30 ciclos de 98 °C por 5 s, 56 °C por 5 s y 72 °C por 15 s; y por último un ciclo de extensión final a 72 °C por 1 min. Los amplicones resultantes, se resolvieron en geles de agarosa al 1% y se visualizaron en un transiluminador de luz UV, después de teñir con 3X GelRed™ (Biotium, EUA), en el equipo Gel Doc XR+ Imaging System (BIO RAD, USA). La purificación de los productos de PCR se realizó con el Wizard® SV Gel and PCR Clean-Up System (Promega, Madison, EUA). Los productos amplificados se enviaron a secuenciar a la empresa Macrogen (Seúl, Corea del Sur).
Análisis filogenético. Las secuencias de ADN fueron editadas y ensambladas en el programa BioEdit 7.2.0 (Hall, 1999). Posteriormente, para el análisis filogenético se utilizó el programa MEGA versión 6 (Tamura et al., 2013). El alineamiento múltiple de las secuencias de ADN se realizó mediante el programa MUSCLE (Edgar, 2004) y el árbol filogenético se realizó por el método de máxima verosimilitud, basado en el modelo Kimura (1980) de 2 parámetros con valores de soporte de 1000 repeticiones de Bootstrap.
RESULTADOS Y DISCUSIÓN
Durante el muestreo anual se obtuvo un total de 34 aislados a partir del 9,72% de larvas utilizadas en el método de insecto-trampa; la mayor presencia de HE se presentó de julio a septiembre de 2016 con 18 aislados, mientras que se observó una disminución de octubre (2016) a junio (2017), con un total de 16 aislados, con excepción de marzo, donde se registró un repunte en la presencia (8 aislados), y con una ausencia de HE durante los meses de abril y mayo (Tabla 1).
Tabla 1 Acrónimo y número de acceso de la colección, mes de colecta, tamaño de conidios (n = 50), identificación filogenética de las especies de Metarhizium y número de acceso GenBank del factor de elongación de la traducción 1-α (5’TEF)
| CHE-CNRCB | Mes de colecta | Conidios (µm) | |||
| Largo (min-max) ± DS | Ancho (min-max) ± DS | Identificación filogenética | Número de acceso GenBank | ||
| 800 | Julio | 6.61(5.63-7.19) ± 0.39 | 2.74(2.42-3.01) ± 0.13 | M. robertsii | OK358818 |
| 801 | Julio | 7.13(5.51-7.95) ± 0.51 | 3.33(2.69-3.85) ± 0.23 | M. guizhouense | OK358819 |
| 802 | Julio | 6.66(5.88-7.54) ± 0.38 | 3.02(2.67-3.54) ± 0.20 | M. robertsii | OK358820 |
| 803 | Julio | 7.94(6.68-9.67) ± 0.65 | 3.36(2.72-3.8) ± 0.25 | M. guizhouense | OK358821 |
| 805 | Julio | 6.87(5.63-7.67) ± 0.41 | 3.25(2.12-3.73) ± 0.32 | M. robertsii | OK358823 |
| 804 | Julio | 6.47(5.54-7.71) ± 0.55 | 2.75(2.2-3.24) ± 0.18 | M. guizhouense | OK358822 |
| 806 | Agosto | 6.56(5.74-7.17) ± 0.31 | 2.54(2.25-2.92) ± 0.14 | M. robertsii | OK358824 |
| 807 | Agosto | 6.67(5.46-8.16) ± 0.70 | 2.60(2.03-3.29) ± 0.29 | M. robertsii | OK358825 |
| 808 | Agosto | 5.59(4.65-6.39) ± 0.38 | 2.79(2.2-3.61) ± 0.35 | M. robertsii | OK358826 |
| 809 | Agosto | 6.19(5.39-7.09) ± 0.31 | 2.50(2.1-3.09) ± 0.19 | M. robertsii | OK358827 |
| 810 | Agosto | 6.81(5.81-8.17) ± 0.44 | 3.21(2.8-3.65) ± 0.21 | M. guizhouense | OK358828 |
| 811 | Agosto | 6.75(6.09-7.86) ± 0.40 | 3.14(2.58-3.65) ± 0.25 | M. guizhouense | OK358829 |
| 815 | Agosto | 5.54(4.91-6.53) ± 0.41 | 2.65(2.03-3.24) ± 0.27 | M. guizhouense | OK358833 |
| 816 | Agosto | 6.56(5.65-7.36) ± 0.35 | 2.43(2.04-2.75) ± 0.14 | M. guizhouense | OK358834 |
| 822 | Agosto | 6.55(5.51-7.46) ± 0.38 | 2.70(2.44-2.98) ± 0.15 | M. guizhouense | OK358840 |
| 812 | Sept | 7.19(6.49-8.35) ± 0.44 | 3.48(2.85-3.96) ± 0.27 | M. robertsii | OK358830 |
| 813 | Sept | 7.69(6.76-8.82) ± 0.52 | 3.30(2.59-3.84) ± 0.24 | M. robertsii | OK358831 |
| 814 | Sept | 7.30(6.6-8.62) ± 0.37 | 3.42(2.8-4.02) ± 0.27 | M. robertsii | OK358832 |
| 817 | Octubre | 6.18(5.28-6.92) ± 0.37 | 2.77(2.4-3.07) ± 0.16 | M. robertsii | OK358835 |
| 818 | Nov | 6.54(5.62-7.47) ± 0.39 | 2.60(2.24-3.01) ± 0.16 | M. robertsii | OK358836 |
| 819 | Nov | 6.15(5.33-6.81) ± 0.30 | 2.74(2.5-3.04) ± 0.11 | M. guizhouense | OK358837 |
| 820 | Dic | 7.07(6.09-7.64) ± 0.32 | 3.12(2.63-3.7) ± 0.20 | M. robertsii | OK358838 |
| 821 | Enero | 6.22(5.26-7.59) ± 0.42 | 2.81(2.52-3.27) ± 0.16 | M. robertsii | OK358839 |
| 823 | Enero | 6.07(5.39-6.6) ± 0.31 | 2.52(2.26-2.78) ± 0.12 | M. robertsii | OK358841 |
| 825 | Febrero | 7.21(6.11-7.91) ± 0.42 | 3.03(2.65-3.43) ± 0.16 | M. guizhouense | OK358842 |
| 826 | Marzo | 6.32(5.35-6.98) ± 0.41 | 2.48(2.06-2.86) ± 0.16 | M. robertsii | OK358843 |
| 827 | Marzo | 6.26(5.42-7.39) ± 0.47 | 2.82(2.27-3.64) ± 0.29 | M. robertsii | OK358844 |
| 828 | Marzo | 7.45(6.53-8.49) ± 0.41 | 3.39(2.83-3.84) ± 0.23 | M. guizhouense | OK358845 |
| 829 | Marzo | 6.92(5.74-8.09) ± 0.63 | 2.50(2.13-2.78) ± 0.14 | M. robertsii | OK358846 |
| 830 | Marzo | 5.77(4.53-7.17) ± 0.54 | 2.51(1.66-3.14) ± 0.33 | M. robertsii | OK358847 |
| 831 | Marzo | 6.14(5.23-7.04) ± 0.48 | 2.69(2.16-3.34) ± 0.23 | M. robertsii | OK358848 |
| 832 | Marzo | 6.14(5.73-6.85) ± 0.26 | 2.66(2.24-2.88) ± 0.14 | M. robertsii | OK358849 |
| 834 | Marzo | 6.40(5.04-7.32) ± 0.34 | 2.45(2.06-2.85) ± 0.17 | M. robertsii | OK358850 |
| 835 | Junio | 5.72(5.11-6.55) ± 0.31 | 2.48(2.21-2.78) ± 0.14 | M. robertsii | OK358851 |
Los aislados mostraron características morfológicas de anverso, forma, elevación, margen y consistencia similares al género Metarhizium (Figura 1a y Figura 1b). Las especies agrupadas en los clados PARB (Metarhizium pingshaense Chen & Guo, M. anisopliae, Metarhizium robertsii Bisch., Rehner & Humber y Metarhizium brunneum Petch) y MGT (M. majus Johnst., Bisch., Rehner & Humber, Metarhizium guizhouense Chen & Guo y Metarhizium taii Liang) son cercanamente iguales en tamaño y forma (Figura 1c y Figura 1d), esto hace que no sea posible distinguir entre especies basado en la morfología de conidios (Bischoff et al., 2009), por lo que el análisis molecular fue necesario para la identificación específica de los aislados.
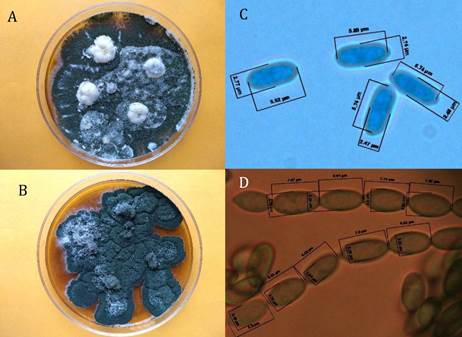
Figura 1. Crecimiento de M. robertsii (A) y M. guizhouense (B) en medio de cultivo. Morfología y tamaño de conidios de M. robertsii (C) y M. guizhouense (D).
La identificación filogenética mediante la secuencia del 5’TEF permitió confirmar la identidad de los 34 aislados, reportados como Metarhizium robertsii (J.F. Bisch, Rehner & Humber) y Metarhizium guizhouense (Metchnikoff) Sorokin (1883). De éstos, 23 aislados se agruparon en el clado de la especie M. robertsii con un valor de bootstrap de 98%, mientras que los otros 11 aislados fueron agrupados en el clado de la especie M. guizhouense con un valor de bootstrap de 85% (Figura 2). Las secuencias 5´TEF fueron depositadas en el GenBank (Tabla 1).
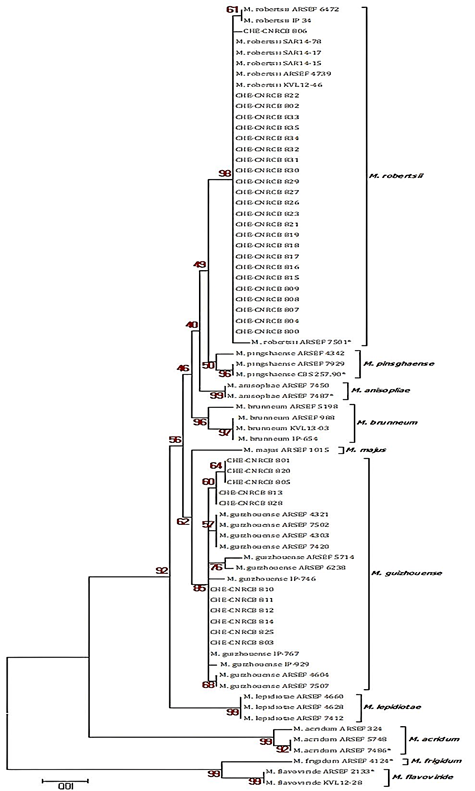
Figura 2. Árbol filogenético de Metarhizium, inferido mediante el método de máxima verosimilitud (ML) de la región del intrón 5´TEF (factor de elongación 1-α), basado en el modelo Kimura de 2 parámetros. Utilizando los 34 aislados de HE (identificados con los acrónimos CHE-CNRCB) y 38 secuencias de referencia del GenBank, las cepas de referencia tipo se señalan con un (*). El soporte de la rama se midió a través de 1000 réplicas de bootstrap.
Este estudio es importante desde diferentes perspectivas; principalmente, para conocer las especies de HE que habitan de manera natural en un agroecosistema, aportar al conocimiento de la biodiversidad de microorganismos del suelo en un cultivo convencional de aguacate en el estado de Colima, y segundo, por la importancia económica que este grupo de organismos representa en la estrategia y desarrollo del control biológico de plagas en este cultivo. Los HE son importantes antagonistas de insectos plaga que habitan en el suelo (Uzman et al., 2019), y su presencia ha sido ampliamente estudiada en México en diversos cultivos como cítricos (Galán-Franco et al., 2011), hortalizas (Pérez-González et al., 2014), tejocote (Muñiz-Reyes et al., 2014) y maíz (Pérez-González et al., 2014).
No obstante, para el estado de Colima, este es el primer estudio realizado sobre HE asociados al suelo de cultivo del aguacate, donde se logró identificar las especies de M. robertsii y M. guizhouense mediante un análisis filogenético con el marcador altamente discriminante 5´TEF. Resultados similares, utilizando el mismo marcador molecular, fueron reportados en Suiza, donde encontraron la presencia del género Metarhizium en muestras de suelo de pastizales, tierra de labranza y bosques, identificando a M. brunneum en los tres sitios, mientras que M. robertsii y M. guizhouense solo fueron encontrados en tierra de labranza y pastizales (Fernández-Bravo et al., 2021). En Arkansas, EUA y utilizando como insecto trampa el ácaro Amblyomma americanum (Acarina: Ixodidae), lograron aislar e identificar molecularmente a las especies de M. robertsii y M. guizhouense (Goldsmith et al., 2021); lo que demuestra la capacidad de adaptación de estas dos especies a diferentes condiciones ambientales y hábitats.
En este estudio, la mayor abundancia de HE se presentó durante la temporada de lluvias, presentando el 64,7% de las larvas micosadas, seguramente favorecido por la humedad que se presenta en esa época del año, la cual beneficia la germinación de conidios y su propagación en el suelo (Ignoffo, 1992). Por otra parte, se ha demostrado que el suelo es el sitio más común para el aislamiento de HE de los géneros Metarhizium y Beauveria (Clifton et al., 2015). En el caso del género Beauveria, no se registró en el área de estudio, posiblemente su ausencia se debe a que es menos común en suelos agrícolas (Barker & Barker, 1998), donde la presencia de B. bassiana se ve disminuida por la aplicación de plaguicidas (Muñiz-Reyes et al., 2014), confirmando que la diversidad de especies está inversamente relacionada con el grado de disturbio humano (Aung et al., 2008; Garrido-Jurado et al., 2015). Por el contrario, se ha indicado que Metarhizium es un género que resiste a las perturbaciones provocadas por la agricultura, además de ser el más común encontrado en suelos cultivados (Meyling & Eilenberg, 2007; Quesada-Moraga et al., 2007; Jabbour & Barbercheck, 2009; Keyser et al., 2015; Hernández-Domínguez et al., 2016; Uzman et al., 2019), esto debido a que sus conidios pueden sobrevivir por más tiempo aún sin hospederos (Vänninen, 1996), incluso, algunas especies del género Metarhizium pueden ser endófitas facultativas (Deaver et al., 2019), lo que les permite sobrevivir en las proximidades de las raíces de las plantas sin llegar a colonizarlas (Moonjely & Bidochka, 2019). La predominancia del género Metarhizium en el suelo del cultivo del aguacate, puede deberse a su capacidad para colonizar fácilmente la rizosfera de las plantas, formando asociaciones endofíticas (Behie et al., 2015), incluso, se ha reportado que especies como M. robertsii puede transferir nitrógeno derivado de insectos a las plantas (Botelho et al., 2019; Barelli et al., 2020).
En este estudio la abundancia de M. robertsii puede explicarse debido a su capacidad endófita, donde no solo tiene actividad patógena sobre insectos, también ha sido reportado como un promotor de crecimiento de raíces (Sasan & Bidochka, 2012), por lo que sugiere un mayor éxito en su capacidad de adaptación en la rizosfera del cultivo del aguacate, sugiriendo que evolutivamente las especies de Metarhizium han mantenido su nicho ecológico como simbiontes de plantas (Moonjely & Bidochka, 2019).
CONCLUSIONES
Los resultados obtenidos reflejan la escasa diversidad de especies de HE asociados al suelo en el cultivo del aguacate, posiblemente por el manejo convencional, que implica el uso de fungicidas, herbicida y aplicaciones de nitrógeno, lo que finalmente repercute negativamente sobre los microorganismos benéficos del suelo, es por esto, que se deben establecer nuevos estudios sobre la presencia, taxonomía y ecología de Metarhizium en los agroecosistemas de México, lo cual ayudaría a comprender el papel que desempeñan en la rizosfera, permitiendo desarrollar programas de control biológico específicos para cada cultivo, y así garantizar su éxito en el control de plagas.












 uBio
uBio 


