INTRODUCCIÓN
Dentro del grupo de bacterias halófilas y halotolerantes se encuentran especies del género Halomonas (Le Borgne et al., 2008), bacterias Gram negativas, heterotróficas, de rápido crecimiento y tolerantes a los ambientes salinos (Kim et al., 2013). Según varias investigaciones realizadas se aislaron especies de Halomonas en plantas marinas (Bibi, 2016), lagos salinos (Singh et al., 2018; Ming et al., 2020; Mamani et al., 2019), llanura costera (Koh et al., 2017), sedimento del Océano Índico, (Qiu et al., 2021), suelo salino (Li et al., 2022; Gan et al., 2018), piscina de minerales (Romano et al., 2019), desiertos (Hintersatz et al., 2023; Patel, & Thaker, 2015), reservorios de petróleo (Mnif et al., 2009), efluentes provenientes del procesamiento del olivo (Liebgott et al., 2008), efluentes prove-nientes del procesamiento de licor (Yang et al., 2010), minas de sal (Cardinali-Rezende et al., 2016), criaderos de Palaemon serratus “camaro-nes” (Xue et al., 2018), y de Litopenaeus vannamei (Kasan et al., 2017).
L. vannamei, conocido como “camarón blanco”, es nativo de la costa oriental del Océano Pacifico, México (ciudad de Sonora) hasta Perú (ciudad de Tumbes), se encuentran en hábitats marinos tropicales. En las etapas juvenil, adolescente, pre-adulta y adulta la desarrollan en estuarios, lagunas costeras y manglares, su tiempo de cosecha es entre 6 y 7 meses (FAO, 2006). Sin embargo, uno de los problemas principales que afecta la producción de estos, es la toxicidad debido a los residuos de alimentación y los desechos orgánicos que producen niveles altos de amonio en solución (Duan et al., 2021).
Una alternativa para el tratamiento de aguas residuales salinas de amonio es el uso de especies de los géneros bacterianos Nitrosomonas, Nitroso-cococcus, Nitrococcus, Nitrospira y Nitrobacter (Yuan et al., 2022; Paungfoo et al., 2007), por su capacidad nitrificante. En investigaciones se ha evaluado la actividad de H. indigenous (Sangnoi et al., 2017), H. boliviensis (Quillaguamán et al., 2004), H. zhanjiangensis (Chen et al., 2009), H. campisalis (Guo et al., 2013) así como también la halotolerancia de H. salifodinae con capacidad de nitrificación heterotrófica y desnitrificación aeróbica (Hu et al., 2022).
El tratamiento de aguas residuales generadas en los criaderos de L. vannamei, es dificultoso, debido a las condiciones extremas que da las concentraciones de sal, y que impiden la nitrificación de bacterias comunes (Sangnoi et al., 2017); en ese sentido, la aplicación de especies halófilas y halotolerantes es recomendado antes que el tratamiento físico-químico, que es una opción muy frecuente (Huan et al., 2021).
El amonio es un compuesto tóxico del nitrógeno causante de la disminución en la tasa de producción, en cuanto, a la densidad de L. vannamei; además la presencia de altas concentraciones de sal, disminuyen el rendimiento del proceso biológico para la eliminación del nitrógeno. Por tal motivo, es justificable haber realizado la investigación para el hallazgo de especies de Halomonas tolerantes a ambientes con altas concentraciones de sal, además de la capacidad nitrificante que presentan, siendo así eficaz para el tratamiento de aguas residuales salinas contaminadas con amonio.
El objetivo de la investigación fue identificar cuáles son las especies del género Halomonas, con capacidad nitrificante, aisladas de sedimentos de criaderos de Litopenaeus vannamei provenientes de langostineras de Tumbes-Perú.
MATERIAL Y MÉTODOS
Obtención de muestra
Se muestreo dos langostineras (EXAPAL SRL y MACORI SRL) ubicadas en la ciudad de Tumbes- Perú, en las cuales se realizó un muestreo dirigido, obteniéndose 3 muestras de sedimento, de 3 criaderos en tiempo de cosecha; de cada criadero se eligieron 5 puntos y se colectaron 50 g que luego se mezclaron para constituir una sola muestra. Los criaderos en promedio tuvieron un área de 3,4 ha cada uno, y registraron valores promedio de pH del agua 7,7 ± 0,3, pH del sedimento 7,2 ± 1, densidad poblacional 10 langostinos/m2, porcentaje de salinidad de 26% y temperatura de 26,3 ± 1 °C (Guan et al., 2020). Las muestras fueron etique-tadas y transportadas al laboratorio en un cooler refrigerado a 4 °C.
Pre-enriquecimiento del sedimento muestreado
Se homogenizó la muestra y se agregaron 25 g (Vreeland, 2015) en un matraz con 250 mL de Caldo Marino estéril. Se incubaron a 20±2 °C por 48 horas (Yang et al., 2010) a 120 rpm (Vreeland, 2015). Se realizó el mismo procedimiento con las demás muestras de cada criadero.
Aislamiento primario
Se realizaron diluciones en serie hasta 10-10 utilizando agua salina artificial (NaCl 5 %, p/v) (Guan et al., 2020). De las diluciones se inocularon por superficie en placa de Petri con Agar Marino, se incubaron (Memmert SN-55) a 30 °C por 2 días (Yang et al., 2010). Este procedimiento se realizó para cada una de las muestras.
Luego, se seleccionaron aquellas colonias con las siguientes características: pigmentación blanca, marrón claro, beige-crema, amarillo-anaranjado, con bordes circulares lisos, brillantes u opacas. A estas colonias se les realizó la técnica de gram, teniéndose en cuenta los siguientes criterios: bacilos Gram negativos, cortos o largos, rectos o curvos (Mata et al., 2002).
Se codificaron las colonias seleccionadas y se cultivaron por estría en placas de Petri estériles con Agar Marino al 10 % de salinidad con un pH de 8 ± 1. Se incubaron a 30 °C por 48 horas. Se verificó la pureza mediante coloración Gram.
Identificación fenotípica de especies de Halomonas
Los cultivos puros fueron cultivados en placas de Petri con Agar Marino al 1,9% de salinidad. Se incubaron a 30 °C por 48 horas. Se realizaron pruebas fenotípicas, siguiendo la metodología sugerida descrita en el Manual de Sistemática de Arqueas y Bacterias Bergey’s (Vreeland, 2015), tales como: reacción Gram, morfología, pigmentación, motilidad, producción de H2S, catalasa, oxidasa, producción de ácidos a partir de glucosa, maltosa, manosa, sacarosa, galactosa, crecimiento en citrato, producción de indol, hidrólisis de urea, almidón, esculina, tween 80, ADN, rango de NaCl (%, w/v) (Mata et al., 2002; Kaye et al., 2004).
Evaluación cualitativa de la capacidad nitrificante de las especies de Halomonas
Los cultivos de Halomonas aislados, fueron sub-cultivados en caldo péptido extracto de carne de vacuno al 10% de inoculo, e incubados a 30 °C y 150 rpm. Luego de 3 días de incubación, el consumo de amonio fue evaluado por el método de Griess-llosvay. En 1 mL del medio de cultivo, se añadieron de 5-7 gotas del reactivo de nitrito. Una reacción positiva se evidenció por el cambio del color del medio a rosado (Sangnoi et al., 2017).
RESULTADOS Y DISCUSIÓN
Los ambientes salobres como es el caso de los criaderos de langostinos presentan las condiciones necesarias para el desarrollo de bacterias halófilas del género Halomonas, los cuales según estudios realizados (Abosamaha, et al., 2022; Agha et al., 2018), se identificaron estas especies en criaderos de Litopenaeus vannamei.
A partir de la muestra del sedimento de criaderos de L. vannamei, se aislaron 5 cultivos bacterianos. Identificados fenotípicamente, según las caracte-rísticas presentadas en estudios científicos de investigación (Ye & Chen, 2021). Todos los cultivos aislados fueron bacilos Gram negativos, con características de colonias como: pigmentación beige-crema, bordes circulares lisos, brillantes en cultivos de 48 horas a 30 °C en agar Marino al 10% de salinidad.
Las características fenotípicas diferenciales de las especies de Halomonas aisladas, se describen en la Tabla 1.
Tabla 1 Característicasdiferenciales fenotípicas de especies nativas de Halomonas aisladas de sedimento de las langostineras de Tumbes, Perú
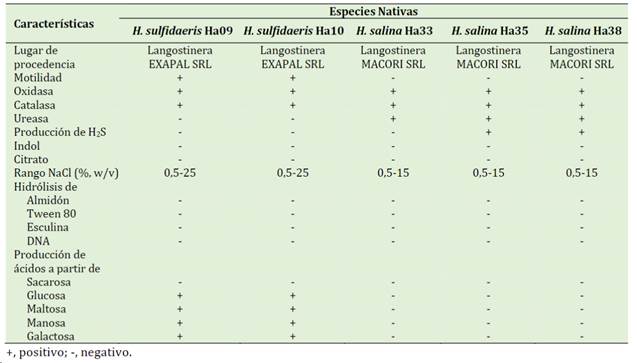
El género Halomonas es un grupo que se encuentra en ambientes con moderada a elevada concen-traciones de sal (Ming et al., 2020; Manya et al., 2021). El crecimiento a diferentes concentraciones de NaCl permitió diferenciar las especies de Halomonas, en el caso de H. sulfidaeris crece en un rango de 0,5% - 24% de NaCl (Ahmad et al., 2023; Saito, 2019), los cultivos codificados como Ha09 y Ha10 presentaron crecimiento dentro de este rango. En el caso de H. salina su crecimiento tolera entre 0,5% - 15% de NaCl (Saito, 2019; Ahmad et al., 2023) rango en el cual crecieron los cultivos Ha33, Ha35, Ha38. También otras pruebas diferenciales que se evaluaron para identificar las especies de H. sulfidaeris y H. salina fueron necesarias para su diferenciación.
H. sulfidaeris presenta movilidad debido a la presencia de flagelos peritricos (Ahmad et al., 2023), como el caso de los cultivos aislados Ha09 y ha10 el cual presentaron estas características. H. salina no presenta movilidad, Ha33, Ha35 y Ha38 tienen esta característica.
En cuanto a la producción de ácidos a partir de azúcares como: sacarosa, glucosa, maltosa, manosa y galactosa, la especie H. sulfidaeris produjo ácidos a partir de todos los azúcares excepto sacarosa (Qiu et al., 2021; Ahmad et al., 2023), mientras que la especie H. salina no presentó esta característica en ninguna de estos azucares (Mata et al., 2002), lo cual, concuerda con los resultados obtenidos en la investigación.
En cuanto a la producción de H2S para la especie de H. salina fue variable (Hintersatz et al., 2023), solo los cultivos Ha35 y Ha38 presentaron producción de H2S.
Según estudios reportados algunas especies del género Halomonas presentan capacidad nitrifi-cante (Lui et al., 2020). Se ha descrito que H. sulfidaeris y H. salina reducen nitrato a nitrito eliminando el amonio por lo cual presentan esta capacidad (Vreeland, 2015; Lui et al., 2020), corroborando los resultados obtenidos ya que todos los cultivos evaluados cualitativamente mostraron capacidad nitrificante.
Este estudio podría ayudar a desarrollar a las especies de Halomonas como opción para el tratamiento de aguas residuales salinas de amonio en langostineras de L. vannamei
CONCLUSIONES
Esta investigación ha reportado el aislamiento de las especies nativas del género Halomonas. Estas especies son H. sulfidaeris y H. salina, con capacidad nitrificante aisladas de sedimentos de criaderos de Litopenaeus vannamei ubicadas en Tumbes-Perú. Las dos especies presentaron características fenotípicas del género, así como la confirmación de la capacidad nitrificante que determina que la eliminación del amonio la cual se desarrolla como una opción eficaz para el tratamiento de agua residuales salinas de amonio.












 uBio
uBio 


