Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO
Links relacionados
-
 Similares en
SciELO
Similares en
SciELO
Compartir
Revista Peruana de Medicina Experimental y Salud Publica
versión impresa ISSN 1726-4634
Rev. perú. med. exp. salud publica vol.36 no.2 Lima jun./set. 2019
http://dx.doi.org/10.17843/rpmesp.2019.362.4269
SECCIÓN ESPECIAL
Aplicación de modelos paramétricos alternativos para el análisis de supervivencia de pacientes con cáncer
Application of alternative parametric models for the survival analysis of cancer patients
Andrea Valencia-Orozco1,2,a, Luis G. Parra-Lara2,b, José W. Martínez3,b,c, José R. Tovar-Cuevas1,d
1 Escuela de Estadística, Facultad de Ingeniería, Universidad del Valle. Cali, Colombia.
2 Centro de Investigaciones Clínicas, Fundación Valle del Lili. Cali, Colombia.
3 Facultad de Ciencias de la Salud, Universidad Tecnológica de Pereira. Pereira, Colombia.
a Magister en Estadística; b médico; c doctor en Epidemiología; d doctor en Estadística.
RESUMEN
En el presente artículo se describe una metodología que permite tener un acercamiento a modelos probabilísticos alternativos para el análisis de supervivencia, con censura por la derecha, distintos a los que usualmente se estudian (distribución: exponencial, gamma, Weibull y log-normal), ya que es posible que los datos no se ajusten siempre con suficiente precisión por las distribuciones existentes. La metodología utilizada permite mayor flexibilidad de modelar observaciones extremas, ubicadas generalmente en la cola derecha de la distribución de los datos, lo cual admite que algunos eventos aún tengan la probabilidad de ocurrir, lo que no sucede con los modelos tradicionales y el estimador de Kaplan-Meier, el cual estima para los tiempos más prolongados, probabilidades de supervivencia aproximadamente iguales a cero. Para mostrar la utilidad de la propuesta metodológica, se consideró una aplicación con datos reales que relaciona tiempos de supervivencia de pacientes con cáncer de colon.
Palabras clave: Análisis de Supervivencia; Modelos Probabilísticos; Funciones de Verosimilitud; Estimación de Kaplan-Meier; Neoplasias del Colon (fuente: DeCS BIREME).
ABSTRACT
This article describes a methodology that allows an approach to alternative right-censored probabilistic models for the analysis of survival, different to those usually studied (exponential, gamma, Weibull, and log-normal distribution) since it is possible that the data do not always fit with sufficient precision due to existing distributions. The methodology used allows for greater flexibility when modeling extreme observations, generally located in the right tail of data distribution, which admits that some events still have the probability of occurring, which is not the case with traditional models and the Kaplan-Meier estimator, which estimates for the longest times, survival probabilities approximately equal to zero. To show the usefulness of the methodological proposal, we considered an application with real data that relates survival times of patients with colon cancer (CC).
Keywords: Survival Analysis; Probabilistic Models; Likelihood Functions; Kaplan-Meier Estimate, Colonic Neoplasms (source: MeSH NLM).
INTRODUCCIÓN
El análisis de supervivencia es un conjunto de métodos, técnicas y/o procedimientos estadísticos para analizar datos que corresponden a tiempos de ocurrencia de algún evento en particular (1). Los métodos tradicionalmente utilizados se dividen en dos grupos: paramétrico (distribución exponencial, gamma, Weibull y log-normal) y no paramétrico (curvas de Kaplan-Meier y los métodos actuariales) (2-4). Los investigadores en ciencias médicas a menudo tienden a preferir el enfoque no paramétrico en lugar del paramétrico debido a que se necesitan suposiciones menores; sin embargo, algunos autores como Efron (5) y Oakes (6), postulan que, en ciertas circunstancias, los modelos paramétricos realizan un análisis más sólido, ya que teniendo en cuenta algunas suposiciones y la selección de una distribución de probabilidad hipotética para los tiempos de supervivencia, la inferencia estadística es más precisa, lo que hace que las desviaciones estándar se vuelvan más pequeñas que cuando no existen tales suposiciones (7). A pesar de que este tipo de enfoque involucra un proceso más riguroso, el análisis de supervivencia paramétrico ha sido abordado en diferentes estudios clínicos, en el cual se han evaluado los modelos tradicionales (exponencial, gamma, Weibull, log-normal), encontrando que la distribución Weibull muestra un mejor comportamiento para el conjunto de los datos analizados (8-10).
No obstante, las distribuciones exponencial, gamma, Weibull y log-normal, no necesariamente reflejan el comportamiento natural de los tiempos observados en un estudio de seguimiento, presentando restricciones relacionadas con la forma de la densidad del modelo (11). Se ha observado en algunas situaciones, conjuntos de datos que presentan valores extremos de importancia, por ejemplo, pacientes que sobreviven periodos de tiempo mayores a los esperados por los médicos tratantes. Tales situaciones provocan que los métodos estadísticos usados arrojen predicciones inexactas de los tiempos y las probabilidades, debido a que la función de verosimilitud asociada a la distribución de probabilidad usada para el modelamiento, no consigue incluir suficiente información sobre los valores extremos, afectando las estimaciones posteriores (11).
Ante estas situaciones, se presenta una propuesta metodológica para modelar tiempos de supervivencia censurados a la derecha, utilizando familias de distribuciones asimétricas como modelos probabilísticos alternativos a los modelos que usualmente se estudian; los cuales permiten tener mayor flexibilidad en cuanto a la forma de la función de densidad, apropiadas cuando ocurren valores extremos con probabilidades pequeñas que no se deben ignorar. Entre los modelos propuestos se encuentran las distribuciones Lindley, power Lindley inversa, exponencial power Lindley y Lévy estándar (12-16). El objetivo del presente estudio es mostrar que existen otras formas paramétricas para estimar la supervivencia, asimismo compararlas con los métodos paramétricos y no paramétricos tradicionales (Kaplan-Meier). Para ilustrar esta propuesta, se utiliza un conjunto de datos relacionados con tiempos de supervivencia de pacientes con cáncer de colon (CC).
MODELOS
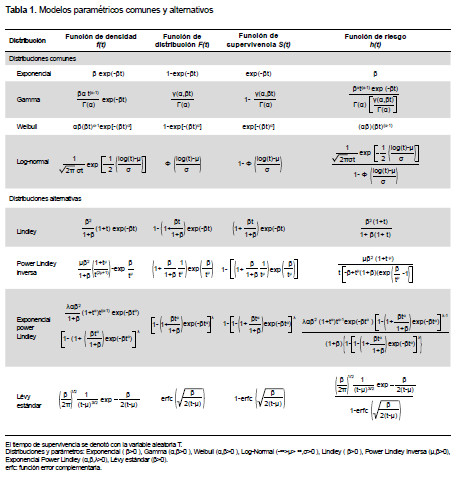
En la Tabla 1 se muestran las distribuciones de probabilidad (comunes y alternativas) aplicadas en el estudio, acompañadas por sus principales funciones del modelo de supervivencia. Las distribuciones comunes son reportadas generalmente en la literatura (11,17). Por otra parte, los modelos propuestos (alternativos) se seleccionaron considerando la asimetría de la distribución, expresiones conocidas de las funciones de densidad y riesgo, y el algoritmo generador de datos.
A continuación, se describen cada uno de los modelos paramétricos alternativos:
DISTRIBUCIÓN LINDLEY
Distribución introducida por Lindley (12) en el contexto de la inferencia bayesiana. Su función de densidad se obtiene mediante la mezcla de las distribuciones exponencial y gamma, resultando una distribución de parámetro de escala β. Ghitany et al. (13), describieron las propiedades y aplicaciones de esta distribución en el contexto de análisis de supervivencia, mostrando que sus propiedades matemáticas son más flexibles que las de la distribución exponencial.
DISTRIBUCIÓN POWER LINDLEY INVERSA
Es una generalización de la distribución Lindley, con el fin de obtener distribuciones de probabilidad más flexibles en relación con los comportamientos de las funciones de densidad y riesgo, comparadas con otras distribuciones existentes para tiempos de vida. Fue propuesta por Sharma et al. (14), quien consideró el inverso de una variable aleatoria con distribución Lindley Inversa. El modelo paramétrico resultante depende de dos parámetros, localización μ y parámetro de escala β.
DISTRIBUCIÓN EXPONENCIAL POWER LINDLEY
Fue propuesta por Ashour y Eltehiwy (15) como una generalización de la distribución Lindley. Esta distribución contiene varios sub-modelos especiales (power Lindley, exponencial Lindley, Lindley). Los parámetros asociados son α, β y λ siendo los dos primeros de forma y escala, respectivamente. El tercer parámetro controla la asimetría de la distribución.
DISTRIBUCIÓN LÉVY ESTÁNDAR
Fue propuesta por Paul Levy en 1925 y pertenece a la familia de distribuciones estables, las cuales permiten modelar la asimetría y colas arbitrariamente más grandes que la distribución normal (16). Esta distribución depende del parámetro de escala β.
ESTIMACIÓN DE LOS PARÁMETROS
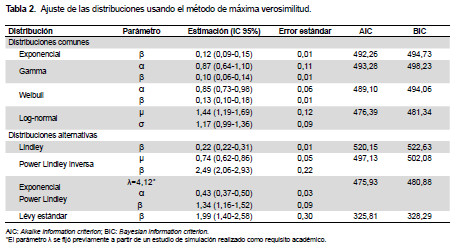
Se utilizó desde la inferencia clásica el método de máxima verosimilitud. Debido a que los datos de supervivencia presentaban censura por la derecha, se estableció la contribución de cada observación (censurada o no-censurada) a la función de verosimilitud, teniendo en cuenta la función de densidad para la parte no censurada y la función de supervivencia para los tiempos censurados (11,17,18). Dada la complejidad de las expresiones obtenidas, se usaron procedimientos numéricos (Newton-Raphson y Nelder-Mead) para el cálculo de las estimaciones correspondientes, usando el programa RStudio® 1.1.456, en donde fue necesario especificar un vector de valores iniciales. Estos procedimientos controlan su convergencia teniendo en cuenta la diferencia absoluta entre sucesivas iteraciones, con una tolerancia de error de 10-8. En el Anexo 1 se presentan las expresiones utilizadas para la estimación de los parámetros.
CRITERIOS DE EVALUACIÓN
Respecto a la comparación de los modelos paramétricos, se utilizó la curva de densidad teórica estimada y el histograma muestral, adicionalmente las funciones de distribución empírica y teórica del modelo. Por otro lado, usando la estimación por máxima verosimilitud, se calcularon los criterios de información, AIC (Akaike information criterion) y BIC (Bayesian information criterion), respectivamente.
FUNCIONES DE SUPERVIVENCIA Y RIESGO
La función de supervivencia permite obtener la probabilidad de que un individuo sobreviva desde la fecha de entrada en el estudio hasta un momento determinado en el tiempo, en cambio la función de riesgo expresa la dinámica del proceso estudiado, al dar sus valores una adecuada aproximación a la tasa de incidencia del evento de interés (3,4,19). A partir de estas definiciones, se estimaron ambas funciones para cada uno de los modelos propuestos, con el fin de caracterizar la población objetivo. Para la función de supervivencia se estimó el intervalo de confianza, utilizando el método bootstrap. Adicionalmente, esta función se comparó con el estimador de Kaplan-Meier y sus intervalos de confianza, asumiendo normalidad asintótica del estimador.
El análisis de datos se realizó mediante el software RStudio® 1.1.456. En el Anexo 1 se muestra un mapa conceptual que resume el desarrollo metodológico del estudio y el código usado en RStudio® 1.1.456 (Anexo 2).
APLICACIÓN
Como una aplicación especial de la propuesta metodológica, se empleó un conjunto de datos proporcionados por una Institución Prestadora de Servicios de Salud (IPS) en Colombia. El archivo de datos contiene información básica sobre individuos atendidos en la consulta de Oncología en el periodo comprendido entre octubre de 2003 y enero de 2016. La información consta de 88 pacientes que ingresaron al estudio escalonadamente a medida que llegaban a consulta. El evento considerado de interés fue «muerte asociada a una falla atribuible al CC», el cual se presentó en 79 de los individuos. Para determinar el tiempo de supervivencia en meses de los pacientes, se tomaron en cuenta dos fechas: fecha de consulta por primera vez con el especialista en oncología y fecha de la última vez que asistió a control con el especialista.
ANÁLISIS EXPLORATORIO DE DATOS
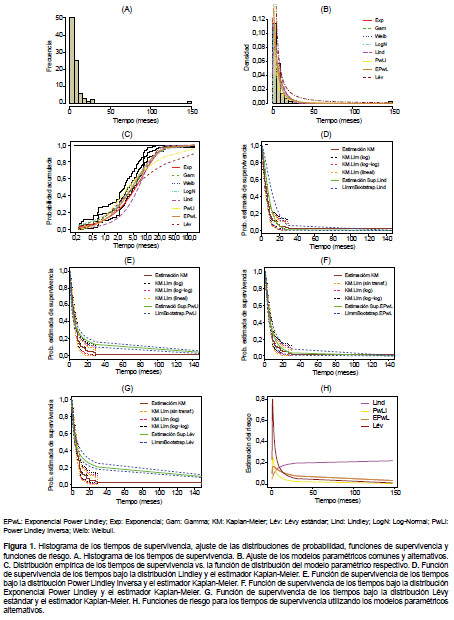
La Figura 1A, muestra la distribución de la variable tiempo. Se encontró una observación mayor que el resto de los datos, por tanto, un histograma con una cola larga hacia la derecha que contiene el mayor número de observaciones, las cuales se concentraron hasta los ocho meses aproximadamente. El tiempo de supervivencia más corto entre los pacientes fue de siete días. El paciente que más tiempo tardó con la enfermedad llegó hasta los 148 meses. El porcentaje de censura fue del 10,2%. En el Anexo 1 aparecen los histogramas de los tiempos de supervivencia censurados y no censurados, respectivamente.
De acuerdo a lo anterior, es posible que las distribuciones más comunes del análisis de supervivencia, no reflejen el comportamiento natural de los tiempos observados, debido a que las curvas de las funciones de densidad de probabilidad tienden a caer relativamente rápido. Sin embargo, con la metodología que se propone, usando otras familias de distribuciones, se logra obtener mayor flexibilidad en cuanto a la disminución de sus colas, siendo apropiadas cuando ocurren valores extremos con probabilidades pequeñas que no se deben ignorar. Como resultado, se presenta a continuación, la estimación de los parámetros y el ajuste de las distribuciones candidatas.
ESTIMACIÓN DE PARÁMETROS
Dos de los modelos paramétricos propuestos (Lévy estándar y exponencial power Lindley) mostraron un buen desempeño en cuanto a la modelación del conjunto de tiempos de supervivencia, según los criterios de información AIC y BIC (Tabla 2). Esto no sucedió con los modelos tradicionales, los cuales presentaron un descenso relativamente rápido hacia cero de su función de densidad, conllevando a que toda la información de la muestra no se incluya al momento de realizar la estimación correspondiente (Figuras 1B y 1C).
Adicionalmente, se muestran las distribuciones de frecuencia empírica y las funciones de distribución teóricas, tal y como se muestra en la Figura 1C; según esta figura, la distribución Lindley fue la que menos se ajustó, ya que se observó un comportamiento distinto de la curva del modelo con las probabilidades empíricas de la muestra, además la curva de la distribución Lévy estándar presentó un comportamiento diferente a los demás modelos, el cual reflejó un AIC más bajo (Tabla 2). Esto se debe a que la distribución Lévy estándar, por ser más flexible, permitió modelar ese dato extremo (148 meses) (Figura 1B).
FUNCIONES DE SUPERVIVENCIA
El modelo exponencial power Lindley presentó una probabilidad asociada a la supervivencia semejante a la obtenida por el estimador de Kaplan-Meier para tiempos muy cortos (seis meses aproximadamente) (Figura 1F). Respecto a los demás tiempos, esta probabilidad fue mayor a la obtenida por el estimador no paramétrico. En la distribución Lévy estándar, las probabilidades asociadas a la supervivencia fueron cambiando a medida que avanzó el tiempo (después de los diez meses aproximadamente) comparado con Kaplan-Meier (Figura 1G). Los límites de confianza de las distribuciones exponencial power Lindley y Lévy estándar presentaron menor amplitud, principalmente en los extremos, similar a los límites del estimador de Kaplan-Meier (Figuras 1F y 1G).
FUNCIÓN DE RIESGO
En los modelos exponencial power Lindley y Lévy estándar, la función de riesgo se comportó de forma semejante al inicio del intervalo de tiempo. Con la distribución Lévy estándar, el riesgo fue más intenso para los tiempos menores, es decir, el modelo presentó un cambio temporal drástico a la ocurrencia del evento (muerte), desde el primer momento en que se empezó a contar el tiempo de vida para el paciente (ver Figura 1H).
En el Anexo 1 se muestran las probabilidades de supervivencia para cada observación de la muestra, teniendo en cuenta las distribuciones alternativas que mejor modelaron a los tiempos de supervivencia, y el método de Kaplan-Meier. Conviene subrayar que las probabilidades obtenidas corresponden a la supervivencia global de la enfermedad (CC) sin importar el estadio clínico del paciente u otra variable de interés.
Este estudio se realizó a partir de una base de datos secundaria con autorización previa de la institución de salud y no requirió aprobación de un Comité de Ética (IRB).
DISCUSIÓN
Este trabajo desarrolló una propuesta metodológica que permite modelar conjuntos de datos con comportamiento asimétrico en el análisis de supervivencia, en donde algunas unidades de la muestra pueden presentar valores muy grandes en comparación con los otros, incluyendo la presencia de datos censurados a la derecha. Para analizar este tipo de datos se utilizaron las distribuciones alternativas, Lindley, power Lindley inversa, exponencial power Lindley y Lévy estándar; como una posible opción a las distribuciones disponibles en la literatura (distribución exponencial, gamma, Weibull, log-normal) y el método no paramétrico Kaplan-Meier. Cabe destacar que este trabajo extiende la posibilidad de incorporar en la modelación, conjuntos de datos censurados a la derecha. Con esta nueva propuesta, los investigadores pueden considerar estas distribuciones en su procedimiento de inferencia, ya que podrían estar utilizando metodologías inadecuadas para los tiempos, ignorando posibles factores de influencia, como la presencia de valores extremos o tiempos prolongados. Una situación particular son los tiempos de supervivencia de pacientes con cáncer, los cuales pueden contener información de personas que sobreviven periodos de tiempo mucho mayores a los esperados por los médicos tratantes, generalmente producidos por factores como la genética, la estadificación del cáncer, la exposición a agentes cancerígenos, o las infecciones, entre otros.
En la literatura, los estudios de supervivencia para cáncer con frecuencia utilizan métodos tradicionales para el análisis de los datos. Entre los estudios que más se divulgan, se encuentran los de cáncer de mama, cérvix, colon, recto, próstata, pulmón y estómago (20-28). En estos estudios fue muy común encontrar durante el análisis estadístico, técnicas que no exigen ningún modelo poblacional a los datos; particularmente el estimador de Kaplan-Meier. Sin embargo, con este estimador no es posible obtener una relación funcional para la curva de supervivencia, ya que solamente se puede estimar una aproximación a la curva real (3,4). Por lo tanto, se propone buscar con la metodología del estudio, que los tiempos puedan ser caracterizados específicamente por un modelo paramétrico, y por las diferentes funciones asociadas, como la función de distribución acumulada, función de densidad de probabilidad, función de supervivencia y función de riesgo, a fin de poder ilustrar distintos aspectos de los datos, y la extracción de inferencias acerca del patrón de supervivencia en la población.
Como una motivación para el presente trabajo, se consideró un conjunto de datos médicos sobre pacientes con diagnóstico confirmado de CC, los cuales permitieron reflejar la flexibilidad de los modelos paramétricos propuesto, ya que el conjunto de datos de cáncer consideraba pocas personas que habían sobrevivido cantidades mayores al resto de observaciones. Bajo la estimación por máxima verosimilitud, se observó que, entre las distribuciones evaluadas, la que se destacó fue la exponencial power Lindley. Esta decisión se tomó a partir de los criterios de información AIC y BIC. Desde el punto de vista de las probabilidades de supervivencia, las distribuciones alternativas permitieron asignar valores diferentes de cero a los eventos que ocurrieron en periodos de tiempos tardíos o prolongados, mientras que por el estimador Kaplan-Meier, el valor de estas probabilidades fue aproximadamente igual a cero. Por lo tanto, las probabilidades obtenidas por estas distribuciones pueden estar más ajustadas a la realidad, puesto que, si la probabilidad fuera cero, no tendría sentido que se presentaran esos tiempos al evento. El uso de estos modelos puede ser complejo, por la dificultad en la interpretación de sus parámetros. Sin embargo, la mayoría de estas distribuciones presentan parámetros de escala, los cuales están asociados a la variabilidad de los datos, es decir, que las distribuciones permiten modelar los tiempos de supervivencia controlando su dispersión mediante este parámetro. Otro de los parámetros que aparecen es la forma, este no tiene una interpretación científica, más bien se considera una característica matemática de la función para poder ajustarse a los datos.
Se debe tener en cuenta que los resultados obtenidos para los datos de CC se calcularon en base a los modelos evaluados en la propuesta metodológica; sin embargo, no implican que sean los únicos modelos que puedan ajustarse a esta situación. Por el contrario, la metodología podría desarrollarse utilizando otras distribuciones de probabilidad, por ejemplo, se pueden considerar distribuciones capaces de modelar funciones de riesgo con forma de bañera, siendo ésta una función difícil de encontrar dentro los modelos de supervivencia tradicionales (29). Igualmente, distribuciones con colas pesadas que favorezcan la modelación de conjuntos de datos que presentan valores extremos de importancia, los cuales dependerán del contexto en que se analicen (30).
Dentro de las limitaciones, los resultados de esta investigación pretendieron presentar modelos alternativos para el análisis de supervivencia, por consiguiente, no se consideró dentro del diseño del estudio, el cálculo del poder estadístico. Por un lado, este estudio surgió de un problema clínico relacionado con el comportamiento de los datos y los resultados sólo son válidos para los datos observados. Por otro lado, se podría considerar un análisis estratificado por estadio del cáncer, el cual tendría tamaños de muestras más reducidos, en este sentido sería útil realizar una aproximación bayesiana que incluya información externa para mejorar las estimaciones.
La metodología es un punto de partida para futuros proyectos de investigación relacionados con el análisis de supervivencia, dado que con frecuencia las fuentes de información cuentan con datos censurados y tiempos de supervivencia prolongados. Esto permitirá a los investigadores contar con herramientas de predicción flexibles en relación con el comportamiento de los datos y las funciones de riesgo, permitiendo estimar de manera más precisa los tiempos de supervivencia.
Contribución de autoría: AV, LGP, JWM y JRT contribuyeron equitativamente en este trabajo. AV y JRT diseñaron la investigación. AV, JGP, JJWM y JRT desarrollaron la investigación, analizaron los datos y escribieron el manuscrito. Todos los autores hicieron importantes contribuciones intelectuales al manuscrito y todos los autores aprobaron la versión final antes de someter el manuscrito. JRT supervisó todo el proceso.
Fuentes de financiamiento: autofinanciado.
Conflictos de interés: Los autores declaran que no tienen conflictos de intereses.
Material suplementario: Disponible en la versión electrónica de la RPMESP.
REFERENCIAS BIBLIOGRÁFICAS
1. Barragan M. Imputación múltiple para riesgos competitivos: análisis de supervivencia para pacientes con cáncer de próstata. Ciudad de México: Universidad Autónoma Metropolitana Iztapalapa [internet]. 2013. [citado el 9 de noviembre de 2018]; 123p. Disponible en https://mat.izt.uam.mx/mcmai/documentos/tesis/Gen.11-O/MARCO_ANTONIO_BARRAGAN.pdf. [ Links ]
2. Liu X. Survival Analysis: Models and Applications. 1a ed. Chichester: Wiley; 2012. 464 p. [ Links ]
3. Mosquera J. Estimación no paramétrica de las funciones características en tiempos de falla en fiabilidad industrial. Universidad del Valle Cali [internet]. 2012. [citado el 3 de diciembre del 2018]; p. 1–27. Disponible en https://campusvirtual.univalle.edu.co/moodle/course/view.php?id=12678.
4. Kaplan E, Meier P. Nonparametric estimation from incomplete observations. Journal of the American Statistical Association. 1958;53(282):457-81. [ Links ]
5. Efron B. The efficiency of Cox’s likelihood function for censored data. Journal of the American Statistical Association. 1977;72(359):557-65.
6 Oakes D. The asymptotic information in censored survival data. J Biometrika. 1977;64(3):441-48. [ Links ]
7. Hashemian A, Beiranvand B, Rezaei M, Reissi D. A Comparison Between Cox Regression and Parametric Methods in Analyzing Kidney Transplant Survival. J World App Sci. 2013;26(4):502–7. [ Links ]
8. Montaseri A , Charati J, Espahbodi F. Application of parametric models to a survival analysis of hemodialysis patients. Nephro-urology monthly. J Nephro-Urol Mon. 2016;8(6):e28738. [ Links ]
9. Nakhaee F, Law M. Parametric modelling of survival following HIV and AIDS in the era of highly active antiretroviral therapy: data from Australia. J Eastern Mediterranean Health. 2011;17(3):231-7. [ Links ]
10. Zhu H, Xia X, Chuan H, Adnan A, Luis S, Du Y. Application of Weibull model for survival of patients with gastric cancer. J BMC Gastroenterology. 2011;11(1):1-6. [ Links ]
11. Hosmer D, Lemeshow S, May S. Applied survival analysis: regression modeling of time-to-event data. John Wiley & Sons, 2011. 416 p. [ Links ]
12. Lindley DV. Fiducial Distributions and Bayes Theorem. J R Stat Soc. 1958;2:102–7. [ Links ]
13. Ghitany M, Atieh B, Nadarajah S. Lindley distribution and its application. J Mathematics and Computers in Simulation. 2008; 78(4):493-506. [ Links ]
14 Sharma V, Singh S, Singh U, Agiwal V. The inverse Lindley distribution: a stress-strength reliability model with application to head and neck cancer data. J Industrial and Production Engineering. 2015; 32(3):162-73. [ Links ]
15. Ashour S, Eltehiwy M. Exponentiated power Lindley distribution. J Advance Research. 2015; (6):895-905. [ Links ]
16. Samorodnitsky G, Taqqu M. Stable Non-Gaussian Random Processes: Stochastic Models with Infinite Variance. 1a ed. New York: Chapman and Hall; 1994. 632 p. [ Links ]
17 Klein J, Moeschberger M. Survival analysis: techniques for censored and truncaded data. Springer Science & Business Media; 2006. 536 p. [ Links ]
18. Matadamas M. Inferencia para modelos de supervivencia de un solo evento y extensiones para modelos de riesgos competitivos. Ciudad de México: Universidad Autónoma Metropolitana Iztapalapa [internet]. 2010. [citado el 13 de abril del 2019]; 123p. Disponible en http://mat.izt.uam.mx/mat/documentos/produccion_academica/toda_la_produccion/Tesis%20dirigidas-14-66.pdf. [ Links ]
19. Arribalzaga E. Interpretación de las curvas de supervivencia. Rev Chilena de Cirugía. 2007;59(1):75-83. [ Links ]
20. Villalobos M, Wendling C, Sierra C, Valencia O, Carcamo M, Gayan P. Supervivencia de cáncer cervicouterino escamoso y adenocarcinoma en pacientes atendidas en el Instituto Nacional del Cáncer 2009-2013. Rev Biomédica. 2016;15(5):263-7. [ Links ]
21. Cortes A, Bravo L, García L, Collazos P. Incidencia, mortalidad y supervivencia por cáncer colorrectal en Cali, Colombia, 1962-2012. Rev Salud Pública de México. 2014;56(5):457-64. [ Links ]
22. Restrepo J, Bravo L, García H, García L. Incidencia, mortalidad y supervivencia al cáncer de próstata en Cali, Colombia, 1962-2011. Rev Salud Pública de México. 2014;56(5):440-7. [ Links ]
23. Ospino R, Cendales R, Sanchez Z, Bobadilla I, Galvis J, Cifuentes J. Supervivencia en pacientes con cáncer de mama temprano tratadas con cirugía conservadora asociada a radioterapia en el Instituto Nacional de Cancerología. Rev Colombiana de Cancerología. 2011;15(2):75-84. [ Links ]
24. Ocón O, Fernández M, Pérez S, Dávila C, Expósito J, Olea N. Supervivencia en cáncer de mama tras 10 años de seguimiento en las provincias de Granada y Almería. Rev Española de Salud Pública. 2010;84:705-15. [ Links ]
25. Pardo C, Cendales R. Supervivencia de pacientes con cáncer de cuello uterino tratadas en el Instituto Nacional de Cancerología. Rev Biomédica. 2009;29(3):437-47. [ Links ]
26. Suárez L, Delgado L, Afonzo, Y, Barrios E, Musé I, Viola A. Sobrevida de pacientes con cáncer de pulmón a células no pequeñas estadio IV. Posible beneficio de la quimioterapia en la práctica asistencial. Rev Médica del Uruguay. 2004;20(3):187-92. [ Links ]
27. Barbosa I, Bernal M, Costa I, Jerez J, De Souza D. Supervivencia del cáncer de pulmón en pacientes tratados en un hospital de referencia en Zaragoza (España). Rev Medicina de Familia SEMERGEN. 2016;42(6):380-7. [ Links ]
28. Pardo C, De Vries E. Supervivencia global de pacientes con cáncer en el Instituo Nacional de Cancerología. Rev Colombiana de Cancerlogía. 2017;21(1):12-8. [ Links ]
29. De Pascoa M, Ortega E, Cordeiro G. The Kumaraswamy Generalized Gamma distribution with application in survival analysis. J Statistical Methodology. 2011;8(5):411-33. [ Links ]
30. Achcar J, Coelho E, Tovar J, Mazucheli J. Use of Lévy distribution to analyze longitudinal data with asymmetric distribution and presence of left censored data. J Communications for Statistical Applications and Methods. 2018; 25(1):43-60. [ Links ]
Correspondencia: Andrea Valencia.
Dirección: Escuela de Estadística, Facultad de Ingeniería, Universidad del Valle, Cali, Colombia.
Teléfono: +57 3212100, ext. 7001-7003.
Correo electrónico: andrea.valencia@correounivalle.edu.co
Recibido: 06/02/2019
Aprobado: 22/05/2019
En línea: 28/06/2019