INTRODUCCIÓN
La enfermedad de Carrión (EC), causada por la infección de Bartonella bacilliformis (B. bacilliformis), es un grave problema de salud pública en Perú, los casos de esta enfermedad presentan incrementos de tendencia cíclicas según reportes de la Dirección General de Epidemiología (DGE). Asimismo, se presentaron brotes en diferentes provincias de los departamentos de Ancash, Cajamarca, La Libertad, Amazonas, Cuzco y Madre de Dios 1-5.
Esta enfermedad es bifásica, la primera fase está caracterizada por anemia hemolítica y fiebre pudiendo producir la muerte; y la segunda fase es más benigna y está caracterizada por erupciones cutáneas verrugosas sangrantes autolimitadas. El control de esta enfermedad se basa en el control vectorial, diagnóstico precoz y medicación oportuna 2-5; sin embargo, en la actualidad todavía se reportan casos.
Por lo descrito, resulta indispensable el desarrollo de una vacuna, la cual requiere años de evaluación sistemática de antígenos. Una alternativa para acortar y mejorar el proceso de selección de antígenos de manera eficiente es usar la información genómica disponible, junto con herramientas inmunoinformáticas 6,7. Por otro lado, existe poca información acerca de posibles vacunas para la enfermedad de Carrión 8.
Las proteínas de membrana externa (OMP de outer membrane protein, por sus siglas en inglés) de muchos patógenos son consideradas buenas candidatas a vacunas. La inmunización con estas proteínas podría ser eficiente para la producción de anticuerpos que bloquearían la infección en modelos experimentales. Las proteínas OMP16, OMP19 y OMP31 de Brucella ovis son antígenos inmunodominantes, anticuerpos contras estas proteínas confieren una importante protección contra la brucelosis en el modelo murino 9,10. Para Chlamydia trachomatis se ha reportado que la proteína MOMP purificada puede generar protección en modelo murino y protege contra infección vaginal 11,12.
También se ha demostrado que las OMP purificadas de Neisseria lactamica protegen contra la infección de Neisseria spp serogrupo B y C aisladas de cuadros de meningitis 13. Otros estudios en vacunas basadas en OMP para Neisseria meningitidis del grupo B han demostrado ser eficaces en inducir respuesta protectora basada en anticuerpos en adultos sanos en Cuba, Brasil, Chile y Europa, con una eficacia entre el 50% al 80% 14-17. Se ha reportado que B. bacilliformis presenta al menos 14 proteínas de membrana externa, las cuales tienen un peso molecular entre 11 KDa a 75 KDa. De estas proteínas de membrana externa, las que presentan 31 KDa, 42 KDa y 45 KDa son las más antigénicas 18.
En el presente estudio se diseñó y evaluó una proteína multiepítope como candidato a vacuna contra la enfermedad de Carrión.
MENSAJES CLAVE
Motivación para realizar el estudio. La ausencia de una vacuna para prevenir la enfermedad de Carrión en zonas endémicas de Perú generó la necesidad de desarrollar una propuesta de vacuna contra este problema de salud pública. Al no existir un modelo animal para esta enfermedad abordamos un modelo de infección in vitro.
Principales hallazgos. La proteína multiepítope M1 es inmunogénica en el modelo murino. Además, los anticuerpos generados contra esta proteína inhiben la invasión de Bartonella bacilliformis a eritrocitos humanos en ensayos in vitro.
Implicancias. Este estudio contribuye al desarrollo de propuestas de vacunas experimentales contra la enfermedad de Carrión.
MATERIALES Y MÉTODOS
El presente estudio es cuantitativo analítico experimental.
SELECCIÓN DE EPÍTOPES Y DISEÑO DE LA PROTEÍNA MULTIEPÍTOPE
Todas las proteínas deducidas del genoma de B. bacilliformis (cepa KC583, número de acceso GenBank CP000524) fueron utilizadas para seleccionar proteínas de membrana externa, estas proteínas fueron consideradas como antígenos candidatos para la elaboración de una vacuna.
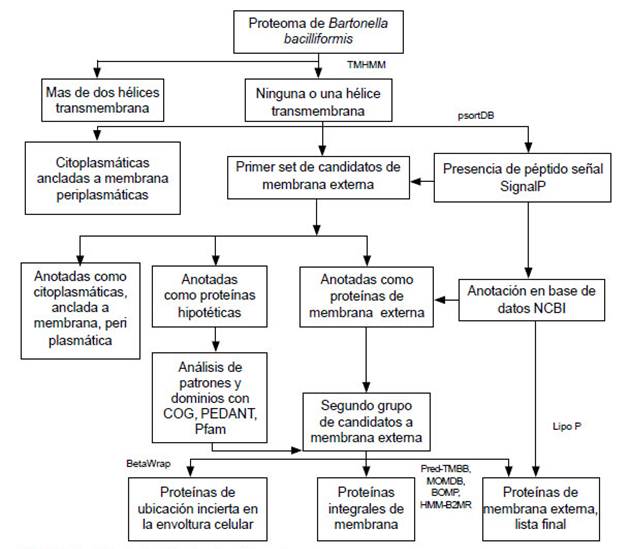
Las proteínas a analizar fueron seleccionadas según el algoritmo indicado en la Figura 1. Primero se seleccionó proteínas que tengan una o ninguna hélice transmembrana. Para investigar si contenían una región transmembrana alfa hélices se usó TMHMM 2.0 server (http://www.cbs.dtu.dk/ services/TMHMM/). Luego, de las proteínas seleccionadas se evaluó su posible localización como proteínas de membrana externa, para ello se usó la base de datos cPsortdb database (http://db.psort.org), se seleccionó si eran proteínas de membrana o eran proteínas cuya localización no se podía determinar. De aquellas proteínas que no se pudo predecir su localización celular se evaluó si presentaban péptido señal usando el programa SignalP 3.0 server (http://www.cbs.dtu.dk/ services/SignalP/), seleccionándose aquellas que presentaron péptido señal. A partir de este primer set de proteínas se seleccionó aquellas anotadas como proteínas de membrana externa o proteínas hipotéticas.
Del grupo de proteínas hipotéticas se seleccionó aquellas que presentaron dominios y patrones compatibles con otras proteínas de membrana externa, para ello se usó las bases de datos PEDANT database (http://pedant. gsf.de), Pfam (http://pfam.sanger.ac.uk/) y COG (http:// www. ncbi.nlm. nih.gov/COG). Se realizó una predicción de lipoproteínas usando el programa LipoP 1.0 server (http://www.cbs.dtu.dk/services/ LipoP/). Para evaluar la probabilidad que las proteínas formen beta hélices se usó el programa BetaWrap (http://groups.csail.mit.edu/ cb/betawrap/). Para identificar posibles estructuras «beta barrel» se usó los programas Betabarrel Outer Membrane protein Predictor (BOMP) (http://www.bioinfo.no/ tools/bomp), el Prediction of TransMembrane Beta-Barrel Proteins server (PREDTMBB) (http://bioinformatics.biol. uoa.gr/PRED-TMBB/), el Markov Chain Model for Beta Barrels prediction program (MCMBB) (http://athina.biol.uoa.gr/ bioinformatics/mcmbb/) y el B2TMR-HMM predictor (http://gpcr.biocomp.unibo.it/ predictors/).
Una vez seleccionadas las proteínas de membrana externa, se evaluó si estas proteínas presentaban epítopes que se unieran al complejo de histocompatibilidad mayor (MHC) I y II utilizando parámetros por defecto en los programas netpanMHC (http://www.cbs.dtu.dk/ services/NetMHCpan/), y netpanMHCII (http://www. cbs.dtu.dk/ services/NetMHCIIpan/). Además, se utilizó BIMAS (HLA Peptide Binding Predictions), SYPEITH (Epitope prediction), LBtope (Linear B-Cell epitope Prediction server - Imtech), BepiPred (BepiPred 1.0 Server), BCPREDs (B-cell epitope prediction server) y EIBD (Linear B cell epitope prediction tool). Se hizo una tabla de epítopes según un ranking de asociación con el MHC I y II.
Para determinar la conservación de los epítopes se comparó la secuencia de los epítopes de todos los genomas reportados mediante BLASTp (www.ncbi. nlm.nih.gov/BLAST). La toxicidad de los epítopes fue evaluada usando el servidor ToxinPred (http://crdd.osdd. net/ raghava/toxinpred/). Además, se seleccionó epítopes que no sean homólogos a epítopes de proteínas humanas mediante BLASTp (http://www.ncbi.nlm.nih.gov/BLAST). Los epítopes fueron evaluados midiendo su índice de accesibilidad con el servidor Emmi Surface Accessibility Prediction (http://tools.immuneepitope.org/bcell). Por otro lado, las proteínas fueron modeladas usando el servidor RaptorX (http://raptorx.uchicago.edu) y sus estructuras visualizadas con PyMol versión 2.3 (https://pymol.org). El modelo de estructura obtenido fue evaluado según el índice de Ramachandra (https://swift.cmbi.umcn.nl/ servers/html/ramchk.html).
Durante el diseño de la proteína multiepítope, se verificó que no generen epítopes nuevos que se pudieran unir a MHC usando netpanMHC y netpanMHCII. Para calcular la cobertura teórica se utilizó el programa Population Coverage del servidor Epitope Prediction and Analysis Tools (http://tools.iedb.org/population/). A la secuencia de aminoácidos de la proteína multiepítope se adicionó seis residuos de histidina en el extremo carboxilo terminal para permitir su purificación mediante resinas de afinidad cargadas en níquel. Luego, a partir de la secuencia aminoacídica de la proteína multiepítope se realizó traducción inversa y optimización de codones para su adecuada expresión en Escherichia coli. Finalmente, el gen sintético obtenido fue sintetizado comercialmente (Integrated DNA Technologies Inc., USA).
CLONAMIENTO, EXPRESIÓN Y PURIFICACIÓN DE PROTEÍNA MULTIEPÍTOPE EN Escherichia coli
El gen de la proteína multiepítope sintetizado en el plásmido pUC (Integrated DNA Technologies Inc., USA) fue liberado mediante digestión enzimática con las enzimas Xho I y Nco I, el fragmento liberado fue purificado y ligado al plásmido de expresión pET28 (Invitrogen Inc, USA) previamente digerido con las mismas enzimas de restricción.
Para unir el fragmento liberado y el plásmido se usó la enzima ligasa T4 (Promega Corp., USA), luego se transformó E. coli BL21 pLys (Invitrogen Inc, USA) competentes preparadas mediante el método de cloruro de calcio 19. La proteína multiepítope fue expresada mediante inducción con 0,5 mM IPTG (siglas de isopropil β-D-1-tiogalactopiranósido) (Sigma-Aldrich subsidiaria Merck, USA), y luego esta proteína fue purificada mediante cromatografía usando la resina de afinidad cargada de níquel Ni-NTA agarosa (QIAGEN N.V., Germany) siguiendo las recomendaciones de los fabricantes (Anexo 1).
INMUNIZACIÓN DE RATONES BALB/C CON PROTEÍNA MULTIEPÍTOPE
Para obtener anticuerpos policlonales se realizó ensayos de inmunización, empleando ratones hembras BALB/c de cinco a ocho semanas de edad. Se inoculó por vía subcutánea a 15 ratones, la primera dosis consistió en 5 µg de proteína multiepítope suplementada con adyuvante completo de Freund (Sigma-Aldrich subsidiaria Merck, USA) a una concentración final de 50%. Luego se realizó tres refuerzos con 5 µg de proteína multiepítope suplementada con adyuvante incompleto de Freund (Sigma-Aldrich subsidiaria Merck, USA). Estos refuerzos fueron administrados los días 7, 14 y 21 después de la inoculación inicial.
Un segundo grupo de 15 ratones fue inmunizado con el mismo esquema, pero usando como adyuvante alúmina (Sigma-Aldrich subsidiaria Merck, USA). Para el grupo control se siguió el mismo esquema de inmunización con diez ratones, reemplazando la proteína multiepítope por PBS (siglas de Phosphate-Buffered Saline) estéril y usando el adyuvante de Freund completo para la primera dosis e incompleto para los refuerzos. Alos 30 días después de la primera dosis se sacrificó los ratones y se colectó sangre total. Después de 30 minutos de coagulación, se separó los sueros mediante centrifugación a 1700 rpm por 15 minutos. Los sueros fueron almacenados a -20 °C hasta su uso.
ELISA
El ensayo de ELISA (siglas de Enzyme-Linked ImmunoSorbent Assay) se realizó según el procedimiento reportado anteriormente con algunas modificaciones 20. Las placas de ELISA fueron impregnadas con 100 ng de antígeno M1 recombinante purificado.
ENSAYO DE INVASIÓN
Eritrocitos humanos fueron obtenidos de donantes voluntarios (los cuales no nacieron ni viajaron a zonas endémicas de enfermedad de Carrión), estos eritrocitos fueron lavados dos veces con medio RPMI 1640 (siglas de Roswell Park Memorial Institute, Sigma-Aldrich) y cuantificados en cámara de Neubauer. B. bacilliformis fue cultivada en medio bifásico, cosechada y resuspendida en medio RPMI. La bacteria fue cuantificada por espectrofotometría a 600 nm.
Aproximadamente 106 bacterias (en un volumen de 50 µL) fueron mezcladas con 50 µL de un pool de sueros de ratones inmunizados o con 50 µL de medio RPMI como control e incubadas por 30 minutos a 30 ºC. Después de esta incubación se añadió la mezcla a 100 µL de eritrocitos lavados (105 eritrocitos), se centrifugó a 500 g por cinco minutos y los viales fueron incubados por tres horas a 30 ºC. Luego de esta incubación, se separó las bacterias libres de los eritrocitos usando gradiente de densidades, para ello se añadió 400 µL de ficoll-hypaque (Sigma-Aldrich) a un vial de 1,5 mL estéril y sobre el ficoll-hypaque se añadió cuidadosamente la muestra de eritrocitos infectados o los controles, estas muestras fueron centrifugadas a 500 g por 30 minutos. Se eliminó el sobrenadante y se lavó los eritrocitos dos veces con RPMI. Se purificó ADN a partir de toda la muestra de eritrocitos infectados utilizando el kit de purificación de PureLink Genomic DNA (Invitrogen) siguiendo las recomendaciones del fabricante con algunas modificaciones, se lisó las células incubando la mezcla de muestra/buffer de lisis/proteinasa K a 65 ºC por una hora y se eluyó el ADN genómico en 200 µL de solución de elusión.
Para calcular el número de genomas equivalentes presentes en la muestra se realizó una reacción en cadena de polimeresa (PCR, por sus siglas en inglés) en tiempo real cuantitativo, para ello se utilizó por cada reacción: 12,5 µL de Sybr Green Master Mix (Roche), 1 µL de 5 µM de primer forward (5’-ATGTSAATGGRAATTTAGG-3’), 1 µL de 5 µM de primer reverse (5’-GGAGCYGTATYAAAGAARTA-3’), y 5 µL de ADN purificado de cada experimento. Se aplicó el siguiente programa de amplificación: 95 ºC por 10 minutos seguido de 45 ciclos de 95 ºC por 20 s, 42 ºC por 20 s, 50 ºC por 20 s y 72 ºC por 30 s. Asimismo, se realizó una curva estándar con ADN genómico purificado de la cepa ATCC (siglas de American Type Culture Collection) KC583 B. bacilliformis. Los ensayos de invasión fueron realizados por duplicado y repetidos dos veces. Por otro lado, los primers utilizados fueron diseñados para el presente estudio, estos primers son sensibles y específicos para la detección de B. bacilliformis por PCR en tiempo real (datos no publicados).
ANÁLISIS ESTADÍSTICOS
Los análisis estadísticos fueron realizados usando R Studio versión 1.2.1335. Para evaluar la significancia estadística de las medias de los títulos de anticuerpos y ensayos de invasión se utilizó la prueba no paramétrica de U de Mann-Whitney para muestras independientes aleatorias. Se consideró como estadísticamente significativos los valores de p menores a 0,05.
RESULTADOS
SELECCIÓN DE EPÍTOPES Y DISEÑO DE LA PROTEÍNA MULTIEPÍTOPE
Las proteínas reportadas del genoma completo de B. bacilliformis fueron 1285 proteínas anotadas, estas fueron descargadas de la base de datos internacional Genbank, esta anotación presenta el estado de provisional. De estas proteínas, 142 tenían un dominio transmembrana tipo hélice (11,1%) y 979 proteínas no presentaban ninguno (76,3%) al ser evaluadas con TMHMM server. De estas proteínas seleccionadas, 24 fueron proteínas de membrana externa (1,9%) y 365 proteínas de localización desconocida (28,4%) al ser evaluadas por pSORT. Luego, se evaluó la presencia de péptido señal en la lista de proteínas cuya localización no podía ser determinada utilizando el programa SignalP, seleccionándose 103 proteínas. Así, se conformó la primera lista de 127 antígenos candidatos.
Esta primera lista se analizó su anotación en el GENBANK y se consultó las bases de datos PEDANT database, Pfam y COG, así se depuró diez proteínas citoplasmáticas y siete proteínas de localización periplasmática. Se incluyó dos proteínas anotadas como proteínas de membrana externa a partir del grupo de proteínas de localización desconocida y que no presentaban un dominio tipo péptido señal predecible. Asimismo, esta lista de proteínas fue depurada con los programas BOMP, PREDTMBB, MCMBB y B2TMR-HMM. Así, se obtuvo una lista final conformada por 112 proteínas. Esta lista incluye tres proteínas autotransportadoras, 25 proteínas de membrana externa (cinco de ellas anotadas como lipoproteínas), diez proteínas asociadas a membrana y 74 proteínas hipotéticas de función desconocida.
Se utilizó la lista de 112 proteínas de membrana externa para seleccionar epítopes que sean reconocidos por el complejo mayor de histocompatibilidad (MHC) de clase I y II correspondientes a los alelos peruanos. Para ello se usaron ocho programas para minimizar los falsos positivos (netpanMHC, netpanMHCII, BIMAS, SYPEITH, LBtope, BepiPred, BCPREDS y EIBD), cada uno de estos programas con sus propios parámetros para la predicción. Se obtuvo una lista de epítopes los cuales fueron priorizados. Asimismo, todos los epítopes presentaron un índice de accesibilidad mayor a 1,00 usando Emmi Surface Accessibility Prediction. Se verificó que todos los epítopes estaban en la superficie de la estructura de sus proteínas modeladas con RaptorX. Asimismo, los epítopes seleccionados no fueron tóxicos. Se verificó la similitud de los epítopes seleccionados con la secuencia del genoma humano, para evitar la homología de secuencia con la proteína humana evitando que se formen autoanticuerpos utilizando Standard Protein BLAST.
Se verificó la conservación de los epítopes comparando las proteínas seleccionadas con cepas de la base del GenBank Hosp800-02, Car600-02, VERO075, PERU38, VERO097, CUSCO5, Cond044, PERU-18, Str. Heidi Mejia y SanPedro600. La lista de epítopes son indicados en la Tabla 1. La combinación elegida de epítopes no formó nuevos epítopes que se unan a MHC, la secuencia aminoacídica de la proteína multiepítope M1 se presenta en la Figura 2.
Tabla 1 Epítopes seleccionados para conformar la proteína multiepítope M1, estos son reconocidos por alelos del complejo mayor de histocompatibilida I y II presente en la población peruana
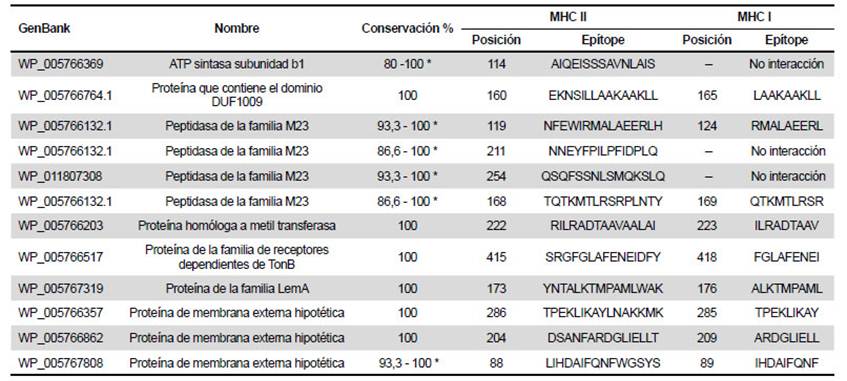
MHC: complejo mayor de histocompatibilidad, No interacción: el epítope no interacciona con MHC A: alanina, R: arginina, N: asparagina, D: ácido aspártico, C: cisteína, Q: glutamina, E: ácido glutámico, G: glicina, H: histidina, I: isoleucina, L: leucina, K: lisina, M: metionina, F: fenilalanina, P: prolina, S: serina, T: treonina, W: triptófano, Y: tirosina, V: valina * El epítope presenta cambios aminoacídicos respecto a la cepa Ver097 (cepa muy divergente considerada como posible subespecie)

Figura 2 Secuencia aminoacídica de la proteína multiepítope M1, conformada por epítopes seleccionados de la Tabla 1, la numeración indica la posición de los aminoácidos en la secuencia de la proteína. En el extremo carboxilo terminal presenta seis residuos de histidina para facilitar su purificación.
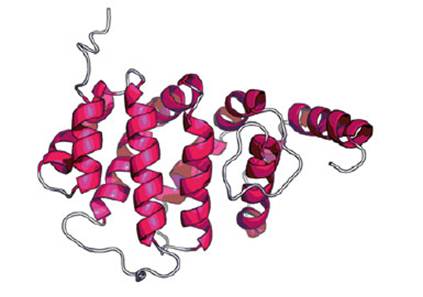
La proteína multiepítope M1 no forma estructuras secundarias complejas según su modelo teórico obtenido con RaptorX (Figura 3). El modelo de la estructura de la proteína multiepítope presenta un índice de Ramachandra de 98,4% (90,3% de residuos en posición favorable y 8,1% en regiones permitidas). Asimismo, la cobertura teórica de una vacuna basada en la proteína multiepítope M1 calculada por el programa Population Coverage es del 99,9% para población peruana.
NIVELES DE ANTICUERPOS OBTENIDO EN RATONES
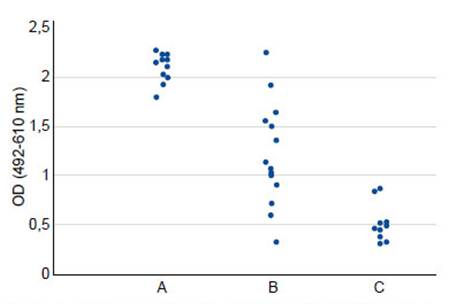
Los ratones inmunizados con la proteína M1 suplementada con coadyuvante de Freund presentaron niveles de anticuerpos IgG más elevados (promedio 2,107; desviación estándar 0,141) que aquellos inmunizados con la proteína M1 suplementada con alúmina (promedio 1,190; desviación estándar 0,507). Estos dos grupos presentaron niveles de anticuerpos IgG más elevados que el grupo control (promedio 0,506; desviación estándar 0,190) (Figura 4). Se evidenció diferencias estadísticas entre los promedios de los grupos inmunizados y el control (p <0,005).
INHIBICIÓN DE LA INVASIÓN DE B. bacilliformis A ERITROCITOS HUMANOS
El control de invasión de eritrocitos incubados con B. bacilliformis presentó cantidades similares de bacteria por ensayo (promedio 2,25 x 105; desviación estándar 0,126) que el grupo tratado con sueros de ratones control inmunizados con PBS/adyuvante de Freund (promedio 2,253; desviación estándar 0,206). Mientras que los ensayos de invasión tratados con los sueros del grupo de ratones inmunizados con M1/adyuvante de Freund (promedio 1,453; desviación estándar 0,170) y el grupo M1/adyuvante alúmina (promedio 1,093; desviación estándar 0,167) presentaron valores menores de bacteria por ensayo, indicando que los anticuerpos contra la proteína multiepítope contenido en los sueros inhiben la invasión de B. bacilliformis a los eritrocitos humanos en ensayos in vitro (Figura 5). Además, se observa una mayor inhibición cuando se utiliza como adyuvante a alúmina. Se evidenció diferencia estadística entre los promedios de los grupos tratados con los anticuerpos anti-M1 y el control (p <0,005).
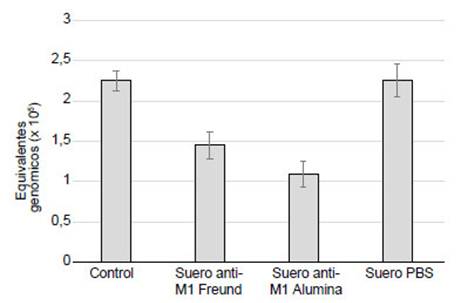
Figura 5 Inhibición de la invasión de Bartonella bacilliformis a eritrocitos humanos en ensayoin vitro. Control: eritrocitos infectados con Bartonella bacilliformis. Suero anti-M1 Freund: la bacteria fue incubada previamente con un pool de sueros policlonales anti-M1 de ratones inmunizados con la proteína M1 y adyuvante de Freund. Suero anti-M1 Alúmina: los sueros policlonales anti-M1 de ratones inmunizados con M1 y adyuvante Alúmina. Suero PBS: los sueros policlonales de ratones inmunizados con PBS más adyuvante de Freund. Las barras representan el promedio de equivalentes genómicos cuantificados luego del ensayo de invasión mientras que los bigotes representan un rango de dos veces la desviación estándar
DISCUSIÓN
La proteína multiepítope M1 es inmunogénica en el modelo murino. Además, los anticuerpos anti-M1 inhiben la invasión de B. bacilliformis a eritrocitos humanos en ensayos in vitro.
Nuestro estudio apunta al desarrollo de una vacuna contra la enfermedad de Carrión que permitiría reducir la mortalidad y morbilidad de esta enfermedad reforzando las medidas de control en Perú. Además, permitiría ahorrar los fondos empleados para el tratamiento de los casos, tanto a los programas de salud como a las familias de los casos. El uso del patógeno atenuado como vacuna para esta enfermedad tendría el riesgo de la reversión de la atenuación. Además, la preparación de antígenos totales o fracciones tampoco es viable en este caso pues es una bacteria de difícil crecimiento. El uso de antígenos recombinantes es una opción que ofrece la ventaja de que es posible obtener cantidades de antígenos adecuadas como para la producción de una vacuna. Asimismo, una proteína multiepítope permitiría el uso de los mejores epítopes del proteoma del patógeno incrementando dramáticamente la calidad del candidato a vacuna. Esta misma aproximación ha sido utilizada para diseñar proteínas multiepítopes candidatas a vacuna contra otras infecciones bacterianas, virales y parasitarias 22-26.
Estos resultados refuerzan la importancia del uso de proteínas de membrana externa para el diseño de vacunas contra infecciones de bacterias gramnegativas como se ha reportado previamente 9-11. Las proteínas de membrana externa constituyen buenos candidatos para el diseño de vacunas. Este algoritmo ha demostrado ser altamente sensible y ha sido utilizado con éxito para predecir proteínas candidatas para una vacuna recombinante contra la clamidiosis, en este estudio la mayor parte de candidatos (74/112) fueron proteínas hipotéticas 22. Mientras que para Brucella, se seleccionó ocho proteínas hipotéticas de 32 23. Este hallazgo concuerda con el hecho que cerca del 27% de proteínas que conforman el proteoma de B. bacilliformis son proteínas hipotéticas 27. Además, las OMP que se han detectado en B. bacilliformis se correlacionan con las reportadas anteriormente 18. Cabe señalar que estos resultados deberán ser confirmados mediante análisis proteómico de OMP de B. bacilliformis.
La proteína multiepítope M1 ha sido diseñado contra infecciones conB. bacilliformis; sin embargo, la inmunización con esta proteína podría generar algún grado de protección contra infecciones con otras especies de Bartonella de manera cruzada ya que los epítopes seleccionados presentan cierto grado de conservación con epítopes de otras especies. Asimismo, la metodología descrita en este artículo podría ser útil para diseñar vacunas contra infecciones de otras especies de Bartonella de importancia en salud pública como: Bartonella ancashensis, Bartonella rochalimae, Bartonella henselae, Bartonella quintana, y Bartonella clarridgeiae.
Entre las limitaciones tenemos que no existe un modelo animal para la enfermedad de Carrión que sea útil para el desarrollo de una vacuna. Es por ello que hemos optado por utilizar un modelo de infección in vitro. Otro modelo plausible es la infección de monos Aotus nancymaae con B. bacilliformis21; sin embargo, este modelo no reproduce los síntomas clínicos como en la infección natural en el hombre. Por otro lado, la base de datos de HLA de población peruana actualmente disponible es muy limitada, es recomendable que esta base de datos sea ampliada para poder realizar predicciones más exactas.
Cabe resaltar que los anticuerpos obtenidos utilizando alúmina como coadyuvante muestra mejores resultados en el ensayo de invasión, esta observación puede ser atribuido a las distintas propiedades de estos adyuvantes. Alúmina incrementa la disponibilidad del antígeno y la activación de células presentadoras de antígeno, mientras que es un pobre inductor de una respuesta inmune mediada por células. Por otro lado, el adyuvante completo de Freund reforzado con adyuvante incompleto estimula preferentemente una respuesta inmune Th2 28,29.
Por otro lado, se espera que la proteína multiepítope M1 pueda despertar una respuesta inmune celular ya que se ha incluido epítopes para HLA tipo I en su diseño. Sin embargo, esto debe de ser evaluado experimentalmente. Los resultados indican que la proteína multiepítope M1 diseñada podría ser utilizada como vacuna contra la enfermedad de Carrión. Mas estudios son recomendables para caracterizar sus propiedades inmunogénicas y las condiciones más adecuadas para su posible utilización como vacuna.