INTRODUCCIÓN
La resistencia a los antibióticos es un problema mundial cada vez más importante, el uso excesivo de antibióticos promueve la generación de cepas resistentes, limitando las opciones terapéuticas disponibles en el campo clínico 1 . Escherichia coli es una bacteria comensal del tracto gastrointestinal; sin embargo, es capaz de causar infecciones entéricas en niños de países en desarrollo como el Perú 2. Esta bacteria gram negativa juega un papel esencial en el desarrollo de la resistencia endémica general 3 y con frecuencia adquiere genes de resistencia al exponerse a la flora normal resistente 4,5.
La mayor evidencia disponible de resistencia antimicrobiana en nuestro país proviene de zonas urbanas o periurbanas. Sólo un estudio previo evidenció altas tasas de resistencia a los antibióticos en cepas comensales de E. coli en niños de Moyobamba (6) . Por este motivo, resulta útil evidenciar la presencia de E. coli comensal albergando genes de resistencia en el tracto gastrointestinal en comunidades rurales del Perú y su condición en el tiempo 3,7-9 , datos actualizados pueden ser útiles para generar conciencia sobre el uso racional de antibióticos.
De este modo, el objetivo del presente estudio fue determinar la resistencia a los antibióticos en cepas comensales aisladas de niños sanos de comunidades rurales de Moyobamba y Urubamba en Perú.
MENSAJES CLAVE
Motivación para realizar el estudio. Es importante tener datos actualizados sobre la resistencia a antibióticos en diferentes poblaciones, incluyendo comunidades rurales, ya que permiten orientar la terapéutica de una manera específica. El análisis de cepas comensales es útil para evaluar la resistencia de una población en general.
Principales hallazgos. Las proporciones de resistencia más altas fueron para cotrimoxazol, ampicilina, ácido nalidíxico y azitromicina, lo que respalda hallazgos de estudios previos en el Perú. Además se encontró un aumento de la resistencia en un periodo de seis meses.
Implicancias. Los altos niveles de resistencia en cepas comensales en niños debería alarmarnos y generar conciencia acerca del uso racional de antibióticos.
EL ESTUDIO
Este estudio es una cohorte prospectiva en el que se analizó a un subgrupo de muestras de un proyecto más extenso en la Sierra y Selva del Perú 10 . La población estudiada es una cohorte de 93 niños sanos entre las edades de seis y 13 meses, de Yucay, Huayllabamba y Ccotohuincho, en Urubamba, y Yantalo y Calzada, en Moyobamba. En el estudio principal, se excluyeron a los niños con historia de desnutrición aguda o crónica, enfermedades congénitas, prematuridad o complicaciones neonatales severas. Los niños fueron seguidos durante un total de seis meses, desde diciembre de 2014 hasta inicios de junio de 2015 10 . Para el análisis, se utilizaron las muestras obtenidas en la primera semana de recolección del primer mes, a tres meses ± 15 días y a seis meses ± 15 días después del inicio del estudio.
El muestreo fue realizado por conveniencia considerando muestras de heces de niños sanos, sin diarrea durante al menos una semana y que no presentaran ningún otro síntoma gastrointestinal. Todos los aislamientos obtenidos identificados como E. coli por métodos bioquímicos, fueron sometidos a un PCR en tiempo real para descartar la presencia de genes de virulencia de E. coli diarrogénicas (DEC) 11 . Mediante el método de difusión de disco se analizaron los siguientes antibióticos: amoxicilina-ácido clavulánico (AMC), ampicilina (AMP), azitromicina (AZM), cefotaxima (CTX), cefoxitina (FOX), ceftriaxona (CRO), cefuroxima (CXM), ciprofloxacino (CIP), gentamicina (CN), imipenem (IPM), ácido nalidíxico (NAL), nitrofurantoína (NIT) y cotrimoxazol (SXT). La interpretación de la prueba fue mediante el uso del manual M100-S25 Performance Standards for Antimicrobial Susceptibility Testing12 . La presencia de betalactamasas de espectro extendido (BLEE) y betalactamasas de tipo AmpC fue detectado fenotípicamente por el método de doble disco.
Se determinó el porcentaje de resistencia para cada tipo de antibiótico y se compararon las tasas de resistencia antibiótica entre Urubamba y Moyobamba para cada antibiótico, utilizando la prueba de chi cuadrado.
El presente estudio contó con la aprobación del Comitéde Ética de la Universidad Peruana Cayetano Heredia (UPCH).
HALLAZGOS
De los 93 niños que participaron en el estudio, 52 eran de Moyobamba y 41 eran de Urubamba. De las 179 muestras, sólo se recogieron 86 al inicio del estudio, 59 a los tres meses y 34 a los seis meses. Asimismo, se perdieron 19 niños durante el seguimiento 10) .
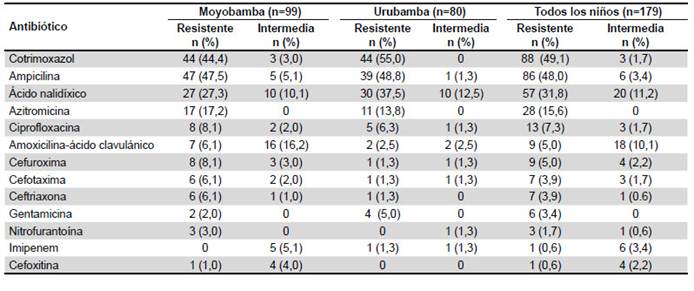
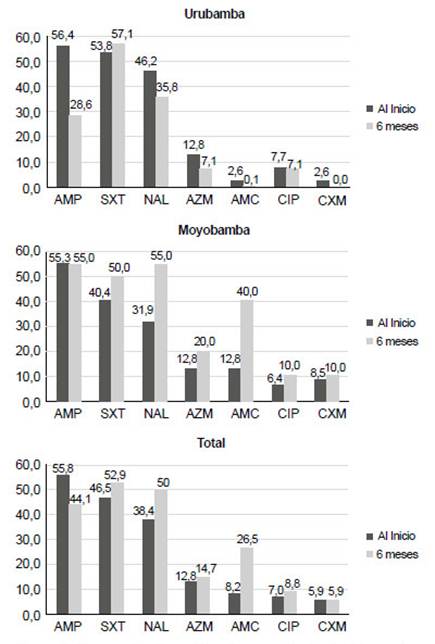
Se encontraron altos niveles de resistencia a cotrimoxazol (49%), ampicilina (48%) y ácido nalidíxico (32%), mientras que el resto de los antibióticos fueron inferiores al 15%. Además, se encontró que el 11% de las muestras mostraron niveles intermedios de resistencia al ácido nalidíxico, lo que resulta hasta en un 43% de muestras con algún grado de resistencia al mismo (Tabla 1). Excepto por la ampicilina, todos los demás antibióticos mostraron una mayor proporción de resistencia a lo largo del tiempo, aunque las diferencias no fueron estadísticamente significativas. La principal diferencia fue para el ácido nalidíxico (38,4% versus 50,0%) y amoxicilina-ácido clavulánico (8,2% versus 26,5%) (Figura 1).
Durante los seis meses de vigilancia se detectó que 22 de 59 muestras con datos pareados disponibles, que se reportaron al inicio como sensibles, se reportan más tarde como resistentes al cotrimoxazol, 19 resistentes al ácido nalidíxico, 17 a la ampicilina y 15 a la azitromicina. Hubo un número menor de muestras que cambiaron de sensible a intermedio, e intermedio a resistente (Tabla 2).
Tabla 2 Cambios en la resistencia a antibióticos en Moyobamba y Urubamba.
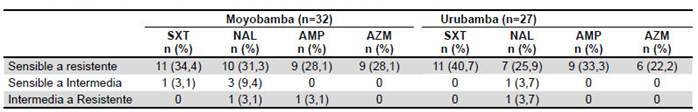
SXT: cotrimoxazol; NAL: ácido nalidíxico; AMP: ampiclina; AZM: azitromicina.
Adicionalmente, el 34% de las muestras se clasificaron como multidrogoresistentes, definido como resistencia a tres o más antibióticos. Aunque se encontró 3,9% de resistencia a ceftriaxona, ninguna de ellas cumplió los criterios para clasificarse fenotípicamente como portadoras de BLEE. Sin embargo, hubo una muestra fenotipícamente productora de betalactamasa de tipo AmpC. El patrón de resistencia de ampicilina-cotrimoxazol-ácido nalidíxico fue el más común con 23%, y ampicilina-cotrimoxazolazitromicina, presente en el 11% de todas las cepas (Tabla 3). Se encontró además una cepa resistente a imipenem, proveniente de Urubamba.
De los 52 niños incluidos en el estudio que vivían en Moyobamba, sólo seis recibieron antibióticos durante el estudio y dos recibieron antiparasitarios. Hubo sólo un caso en el que la exposición a un antibiótico específico estuvo asociado con el desarrollo de resistencia.
DISCUSIÓN
En este estudio, encontramos altas proporciones de resistencia a varios antibióticos comúnmente prescritos en el nivel de atención primaria. Las tasas más altas se encontraron para cotrimoxazol (49,1%), ampicilina (48,0%), ácido nalidíxico (31,8%). Estos resultados fueron consistentes con otros estudios realizados en comunidades rurales de Perú y Bolivia por Bartoloni et al. hace diez años, en los que reportó altas tasas de resistencia para cotrimoxazol (94%) y ampicilina (95%) y un valor similar para el ácido nalidíxico (35%) 6 . La diferencia entre estos resultados probablemente pueda explicarse por el método utilizado para el análisis de muestras; Bartoloni et al. utilizaron un método rápido de placa directa en el que se aplicaron los discos de antibiótico directamente en la placa sembrada sin aislamiento previo de la bacteria teniendo una mejor representación de la flora bacteriana intestinal de cada paciente. Por otro lado, Bryce et al. reportaron en un meta-análisis una prevalencia de resistencia de 37,6% a ampicilina y 28,6% a cotrimoxazol obtenida de un conjunto de datos de diversos países de manera similar a lo encontrado en este estudio 3 . Finalmente, Mosquito et al. reportó resistencia para ampicilina (85%), ácido nalidíxico (68%) y cotrimoxazol (60%) encepas de E. coli comensales en Perú 13) .
Se debe hacer énfasis en la tasa de resistencia significativa encontrada para el ácido nalidíxico, considerando que este es un marcador de resistencia a las quinolonas, para las cuales se han reportado altas tasas de resistencia en Perú, como en el estudio Pons et al. en el que encontró una tasa de 32% para ácido nalidíxico y 12,2% para ciprofloxacino en niños de zonas periurbanas de Lima 14 , así como en el estudio de Montañez et al. quienes encontraron datos de resistencia a ciprofloxacino entre 31% y 69,8% en E.coli causantes de infecciones del tracto urinario a lo largo de los años 15 . Además, Mosquito et al. reportó que las cepas comensales mostraron tasas de resistencia significativamente mayores a quinolonas que las cepas con genes de virulencia para DEC. Estos antibióticos se usan comúnmente en infecciones bacterianas gastrointestinales, del tracto urinario e incluso respiratorio; sin embargo, la mayoría de las infecciones que se observan en niños menores de dos años se deben a etiologías virales.
Los resultados también mostraron un aumento de las proporciones de resistencia en el tiempo para casi todos los antibióticos (Tabla 1) (Figura 1). Aunque los datos obtenidos no fueron estadísticamente significativos, existeb una clara tendencia ascendente, que podría analizarse en estudios futuros con una población más grande. Esto podría explicarse por la exposición a antibióticos en otros miembros de la familia, en los alimentos y el agua, y la transmisión de genes entre las bacterias en el tracto gastrointestinal 3).
Otro hallazgo importante fue el porcentaje de cepas multidrogoresistentes (34%). El patrón más común encontrado entre las bacterias multidrogoresistentes fue ácido nalidíxico-ampicilina-cotrimoxazol (NAL-AMPSXT), antibióticos que todavía se usan empíricamente en el tratamiento de infecciones comunes. Este hallazgo coincide con lo descrito por Ochoa et al. quienes encontraron el patrón de ampicilina-cotrimoxazol en un porcentaje similar en cepas de E. coli diarreogénicas en niños menores de un año en Lima 16. Estos hallazgos reflejan la magnitud del problema encontrado con la resistencia a los antibióticos en las comunidades rurales, así como la necesidad de la vigilancia de las prescripciones de antibióticos por parte de los profesionales de la salud y la automedicación 17 .
Tabla 3 Patrones de resistencia a múltiples fármacos
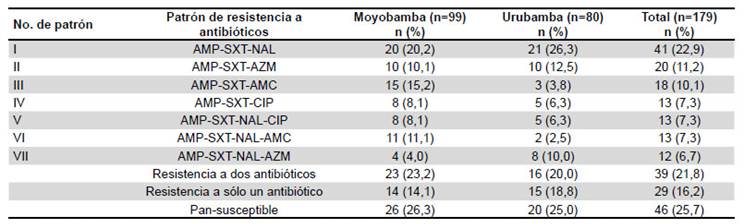
AMP: ampicilina; SXT: cotrimoxazol, NAL: ácido nalidíxico; AZM: azitromicina; AMC: amoxicilina-ácido clavulánico; CIP: ciprofloxacino.
Encontramos resistencia a ceftriaxona pero no cepas productoras de BLEE. No obstante, en el estudio realizado por Riveros et al. encontraron 87% de BLEE en estas cepas, usando el método de placa directa en las mismas muestras 18 . Por otro lado, se encontró una cepa resistente a carbapenems en Urubamba, lo cual podría marcar una condición emergente que debería ser estudiada.
La principal fortaleza de este estudio fue el aislamiento e identificación de cada cepa de E. coli previa al análisis de resistencia. Al hacer esto, se identificaron otras bacterias comensales, como Klebsiella spp. La heterogeneidad de las bacterias en las muestras de heces podría alterar las tasas de resistencia encontradas en el estudio de placa 6,9 directa, como en el llevado a cabo por Bartoloni et al. . Otra fortaleza fue la recolección de muestras seis meses después en los mismos niños. Aunque nuestro tamaño de muestra fue relativamente pequeño, es de gran interés encontrar tasas de resistencia crecientes en un periodo de observación tan corto.
Sin embargo, hubo algunas limitaciones importantes. En primer lugar, el tamaño de la muestra fue relativamente pequeño y no se obtuvieron muestras de cepas comensales para todos los niños en los intervalos de tiempo establecidos, lo que probablemente explica la falta de significación estadística de los resultados. Asimismo, debido a que la metodología fue creada en base a los objetivos del estudio principal y por tanto, se tenía una muestra limitada, el método de muestreo por conveniencia fue elegido. Esto implica distintos tipos de sesgo debido a la falta de aleatoriedad, representatividad de la población y estimación exacta de la tasa de resistencia a antibióticos, por lo que los datos tan sólo indican una tendencia.
En conclusión, este estudio respalda hallazgos previos de resistencia en cepas comensales, inclusive de cepas multiresistentes en comunidades rurales en el Perú y el aumento en las proporciones de resistencia a lo largo del tiempo. Asimismo, se podría incluir el impacto del uso de antibióticos en estos cambios. Recomendamos el desarrollo de más estudios que evalúen los cambios en la resistencia a lo largo del tiempo en poblaciones más grandes ya que encontramos una tendencia sorprendentemente ascendente durante un corto periodo de seis meses en nuestro estudio.